株高和穗位高是玉米育种中的重要农艺性状。本研究利用我国玉米育种中骨干亲本黄早四与来自不同杂种优势群的其他11个骨干自交系组配11个RIL群体, 开展基于单环境、联合环境的QTL分析, 分别检测到269个和176个QTL。通过区段整合, 检测到21个株高主效QTL及15个穗位高主效QTL, 这些QTL分布在第1、第2、第3、第6、第7、第8、第9、第10染色体上。相对于共同亲本黄早四而言, 部分QTL在不同RIL群体中的效应方向一致, 来自共同亲本黄早四的等位基因在不同群体中能够稳定地表达。同时, 还分别定位到在多环境下稳定表达的5个株高、4个穗位高“环境钝感QTL”。此外, 进一步鉴定出5个重要的株高和穗位高QTL富集区段(bin 1.01-1.02, 1.08-1.11, 3.05, 8.03-8.05和9.07), 这些区段均包含多个株高和穗位高相关QTL, 如bin3.05位点包含7个QTL, bin8.03-8.05位点分别包含9个QTL, 且这些QTL至少在3个不同环境中能够被检测到, 这些区域对QTL的精细定位和克隆有重要参考价值。
Plant height and ear height are two important agronomic traits in maize. In this study, 11 RIL populations developed by crossing a common parent (Huangzaosi, a foundation inbred line of maize breeding in China) with other 11 elite inbred lines were applied to QTL mapping for plant height and ear height based on phenotype data of three locations in two years. A total of 269 QTL detected by single-environment analysis and 176 QTL by joint analysis were identified across all of six environments, respectively. Collectively, 21 major-effect QTL for plant height and 15 major effect QTL for ear height were detected, which were located on chromosome. 1, 2, 3, 6, 7, 8, 9, and 10. It was found that the common parent (Huangzaosi) contributed the positive alleles for some QTL across different RIL populations. Five and four environment-insensitive QTL for plant height and ear height were also identified, respectively. Five QTL clustering regions related to plant and ear height, such as bin1.01-1.03, 1.08-1.11, 3.05-3.06, 8.03-8.05, and 9.07, were dissected, of which each region were covered under more than three environments. These genomic regions would be valuable for fine mapping and cloning of height related QTL in maize.
玉米株高和穗位高是重要的农艺性状, 也是玉米育种的重要目标性状。剖析玉米株高和穗位高的遗传结构, 对制定有针对性的育种方案具有重要指导意义。20世纪80年代以来, 分子标记技术的迅速发展和数量性状位点统计模型的日臻完善, 为从分子水平研究玉米株高、穗位高的遗传基础提供了有力的支持。国内外研究者对玉米株高和穗位高QTL定位已有诸多报道, 截至2012年10月, MaizeGDB网站(http://www.maizegdb.org/)已经登录了2278个玉米相关性状QTL信息, 其中包括319个株高QTL和43个穗位高QTL。株高QTL主要分布于第1、第3、第8染色体bin1.03、1.06、1.08、bin3.04-3.06、bin8.05-8.06等区域, 穗位高QTL主要分布于第1、第3、第6、第10染色体bin 1.08、3.04、6.06和10.03等区域, 也有较多分布于bin 8.05区域。另外, 还有几个关于玉米株高、穗位高的基因已被成功克隆, 如Winkler等[1]克隆出编码细胞色素P450的基因 Dwarf3( D3), 该基因参与早期赤霉素的生物合成; Bensen等[2]克隆了 Anther ear1( An1)基因, 该基因编码一个参与合成对映-贝壳杉烯的生化酶, 而对映-贝壳杉烯参与了赤霉素的生物合成; 张祖新等[3]利用综3及其染色体代换系SL15将株高位点 qPH3.1 ( ZmGA3ox2)定位在侧翼标记为ND87-ND88的12.6 kb的空间内并成功克隆到该QTL, 该基因编码GA3- β羟化酶的生物合成。
需要注意的是, 这些研究主要利用一个或少数几个传统意义上的双亲杂交群体进行QTL定位, 导致仅能发现较少的基因组区域及其等位变异。多重相关性作图群体可以弥补这方面的不足[4], 例如, 可提高作图精度, 还可以深入了解QTL在不同遗传背景下的表达稳定性状况, 为QTL精细定位和克隆以及未来的分子设计育种提供指导。
本研究以我国玉米优异骨干自交系黄早四与来自不同杂种优势群的其他11个重要自交系分别组配的大规模重组自交系(RIL)家系群为试验材料, 结合多年多点的表型鉴定, 发掘在不同环境条件下稳定表达的主效QTL, 解析共同亲本黄早四等位变异在不同遗传背景中的表达情况, 以阐明玉米株高、穗位高的遗传基础, 旨在为玉米株高和穗位高分子标记辅助选择、相关候选基因克隆提供支持和参考。
以黄早四为共同亲本, 与11个来自不同杂种优势群的重要自交系杂交, 通过单粒传法获得11个RIL群体, 共计1972个家系的F7重组自交系。其他11个自交系分别为K12、掖478、郑58、获白、齐319、威风322、旅28、黄野四3、多229、Pa405和Mo17。11个RIL群体及其家系数目列于表1。
于2009年和2010年, 同时在北京中国农业科学院作物科学研究所昌平试验基地、河南新乡中国农业科学院试验基地和新疆农业科学院乌鲁木齐安宁渠试验站3个地点, 种植11个重组自交系群体及其亲本。河南新乡为夏播, 北京、新疆均为春播。这3个试验点位于我国不同的玉米生态区, 具有广泛的农业生态环境多样性。采用随机区组设计, 2次重复, 单行区, 行长3 m, 每行10株, 行距0.6 m, 种植密度5.25万株 hm-2, 其他田间管理措施同当地生产管理。
散粉15 d后调查株高、穗位高, 从每小区第3株开始, 连续调查5株。株高即从地面至植株雄穗顶部的高度, 以cm表示; 穗位高为从地面到最上部果穗着生节的高度, 以cm表示。
采用CTAB法[5]提取各家系及群体亲本的基因组DNA (每个家系混合提取10个单株的幼叶), 然后利用787个SNP标记鉴定基因型。各群体多态性SNP标记数在155~211之间。采用MAPMAKER/EXP3.0软件构建各群体的连锁图谱。采用Haldane[6]函数将重组率转化为遗传距离(cM), 用“map”命令确定标记之间的遗传距离, 再用“ripple”命令检测标记顺序, 用Mapchart2.1绘制连锁图。
采用SAS软件的PROC GLM程序, 进行株高和穗位高的六环境联合方差分析, 用SAS的PROC UNIVARIATE和PROC CORR程序做各性状的正态分布检验和相关分析。按照公式 h2= σG2/( σ2G+ σ2GE/ n+ σ2e/ nr)计算遗传力, 这里 σG是遗传方差, σGE是基因型与环境互作的方差, σe是误差, n为环境数, r为重复数[7]。利用软件PLABSTAT (基于dos操作界面的一种软件, 将数据按照软件要求格式整理成*.dat文件即可)分别计算11个RIL群体株高、穗位高遗传相关系数。
利用基于混合线性模型的QTL Network 2.1软件分别进行单环境及6个环境的联合QTL分析[8]。以 P=0.05为统计检测阈值, 即当标记的 P值小于统计检测阈值时, 认为该标记处存在1个与性状有关的QTL, 同时估算QTL与环境及QTL之间的互作效应。
将在联合分析和单环境下都能够检测到, 且单环境下表型贡献率大于10%的QTL定义为“主效QTL”; 将在5个或以上环境中都检测到的QTL定义为“环境钝感QTL”, 将不同群体的QTL定位结果映射到IBM 2008 Neighbors遗传图谱上, 相同染色体区域覆盖多个主效QTL的染色体区域定义为株高、穗位高“QTL富集区段”。
2.1.1 株高和穗位高的遗传变异 11个RIL群体株高、穗位高家系间差异均达到极显著水平, 表明11个群体株高、穗位高在不同环境下的表现差异显著(表1)。11个RIL群体株高和穗位高在6个环境下的遗传力均高于0.90。与穗位高相比, 除多229/黄早四、Mo17/黄早四群体的株高遗传力较高外, 其他群体株高遗传力相当或较低。
2.1.2 相关分析 由表2可知, 各群体的表型相关及遗传相关均表现为显著正相关。表型相关系数变化范围在0.71 (旅28/黄早四)~0.83 (齐319/黄早四), 遗传相关系数在0.67 (Pa405/黄早四)~0.85 (郑58/黄早四和齐319/黄早四)之间。表明, 11个RIL群体的株高和穗位高可能受到一些共同基因和代谢途径的调控, 遗传基础具有相似性。
| 表1 11个RIL群体6个环境下的联合方差分析Table 1 Analysis of variance (ANOVA) for plant height and ear height in the 11 RIL populations under six environments |
| 表2 联合环境条件下11个RIL群体的株高和穗位高表型及遗传相关统计分析表Table 2 Phenotypic and genetic correlations between plant height and ear height in the 11 RIL populations across six environments |
同时发现, 株高与穗位高呈现一种线性的相关性(图1)。在不同环境条件下, 两者线性相关的决定系数 R²变化范围在0.36 (旅28/黄早四)~0.76 (郑58/黄早四)之间。表明在不同的生态地理区域及不同的年份间, 尽管气候条件和田间管理措施等存在差异, 目标性状间却呈现出显著的线性相关关系, 进一步表明株高和穗位高之间在遗传基础上具有 较大的相似性; 但各群体间相关性的差异, 则说明在不同群体中株高和穗位高的遗传基础可能不尽相同。
11个RIL群体在单环境分析条件下, 分别检测到124个株高和145个穗位高QTL (未列出)。除第4、第5染色体外, 其他染色体上均检测到大量的株高、穗位高主效QTL (图2)。其中, 大部分主效QTL聚集在第1、第3、第8染色体上。就单个QTL解释表型变异的变化范围来说, 株高为5.27%~17.88%, 穗位高为6.21%~21.85%。
 | 图1 11个RIL群体6个环境下的株高(PH)、穗位高(EH)线性相关拟合图Fig. 1 Relationships between plant height (PH) and ear height (EH) for all the 11 RIL populations under six environments |
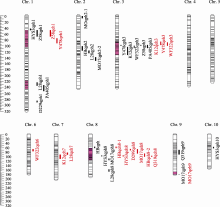 | 图2 11个RIL群体株高和穗位高主效QTL定位状况黑色代表株高主效QTL, 红色代表穗位高主效QTL, 染色体上标注的紫红色区域代表株高和穗位高相关的重要的候选基因区段。Fig. 2 Genomic distributions of detected QTL for plant height and ear height in the 11 RIL populationsColors indicate QTL regions of plant height by black and ear height by red, amaranth bars indicate important candidate regions about plant and ear height. |
在6个环境联合分析条件下, 11个RIL群体中分别检测到83个和93个株高和穗位高QTL。表3和表4分别显示了株高和穗位高在6个环境联合分析下定位到的主效QTL, 其中部分区域在多个环境下均能被检测到, 例如位于Bin1.02的穗位高QTL Z58qeh1 (表4), 在6个单环境和多环境联合分析下均能被检测到。对定位到的株高和穗位高QTL, 根据定位区间的染色体位置进行整合, 最终确认了21个株高主效QTL和15个穗位高主效QTL (表3、表4和图2)。
在11个RIL群体中, 针对株高和穗位高分别检测到5个和4个“环境钝感QTL” (表3和表4)。这些“环境钝感QTL”一般能够解释较高的表型变异, 9个钝感QTL中的6个解释的表型变异率超过10%。其中8个钝感QTL的增效等位基因来自共同亲本黄早四。同时, 还鉴定出5个覆盖多个株高和穗位高QTL的“QTL富集区段”(表5和图2), 除bin1.08- 1.11的QTL富集区段仅控制株高性状以外, 其余QTL富集区段均包括同时控制株高和穗位高的QTL。同时还发现, 这些区段中, 除2个QTL富集区段的增效等位基因来自于不同亲本以外, 其余3个QTL富集区段的增效等位基因均来自共同亲本黄早四(表5)。
株高和穗位高QTL定位研究前人已有很多报道, 本研究的部分结果与已有研究结果一致性较高, 但也发现了一些新的QTL区域。例如, 在bin1.02-1.03区域, 其他研究者检测到控制株高的重要QTL[9,10], 本研究在郑58/黄早四群体、黄野四3/黄早四群体的此区域也检测到相应的株高主效QTL ( Z58qph1、 HYS3qph1), 掖478/黄早四群体和郑58/黄早四群体在的区域还检测到2个主效穗位高QTL ( Y478qeh1、 Z58qeh1)。同样, 在bin1.07-1.09区域, 前人发现存在株高主效QTL[9,11,12,13,14,15,16,17], 本研究在旅28/黄早四群体和Pa405/HZ4群体中也检测到2个主效QTL ( L28qph1、 PA405qph1)。前人在bin3.05-3.06处定位到株高主效QTL[9,18,19], 张岩等[19]还检测到穗位高主效QTL, 本研究中除郑58/黄早四群体和Pa405/ HZ4群体在此处仅检测到株高QTL Z58qph3和 PA405qph3外, K12/HZ4群体、掖478/黄早四群体和威风322/黄早四群体均同时检测到株高和穗位高QTL ( K12qph3、 Y478qph3、 WF322qph3、 K12qeh3、 Y478qeh3和 WF322qeh3)。相似的是, 前人在bin 8.03-8.05区域检测到株高QTL[14,20,21,22], 张岩等[19,22]还检测到穗位高QTL, 本研究除在齐319/黄早四群体和多229/黄早四群体在的此区域仅检测到穗位高QTL Q319qeh8和 D229qeh8外, 获白/黄早四群体、黄野四3/黄早四群体和Mo17/HZ4群体均同时检测到株高和穗位高QTL ( HBqph8、 HYS3qph8、 Mo17qph8、 HBqeh8- 1、 HBqeh8- 2、 HYS3qeh8和 Mo17qeh8)。本研究多229群体/黄早四在bin1.11处还发现表型解释率较大的株高主效QTL D229qph1, 这与张岩等[19]的报道一致。另外, 在本研究中还发现Mo17/黄早四群体在bin9.07处存在1个解释表型变异较大的穗位高QTL ( Mo17qeh9), 这在前人的研究中暂未发现。在本研究中, 利用多个有关联的RIL群体, 检测到一系列株高和穗位高QTL, 其中大部分主效QTL与前人定位的QTL区间位置相同或临近。本研究的定位结果, 一方面进一步验证了已有研究的可靠性, 另一方面也说明这些能够被多次重复定位的基因组区段和新发现的主效区段均是控制玉米株高和穗位高的重要区域。针对这些区段的深入研究和挖掘对剖析玉米株高和穗位高的遗传基础具有重要意义。
| 表3 6个环境联合分析下在11个RIL群体中检测到的株高主效QTLTable 3 QTL for plant height based on joint analysis across six environments in the 11 RIL populations |
| 表4 6个环境联合分析下在11个RIL群体中检测到的穗位高主效QTLTable 4 QTL for ear height based on joint analysis across six environments in the 11 RIL populations |
| 表5 不同遗传背景下确认的株高和穗位高QTL富集区段Table 5 Common QTL regions for plant height (PH) and ear height (EH) across all the populations and genetic backgrounds |
本研究在多重相关群体中, 定位到一些覆盖多个QTL, 并且在多个环境下能够稳定表达的株高和穗位高相关“QTL富集区段”。这些区段覆盖的主效QTL一般可以解释较大的表型变异, 其中部分QTL与已克隆的功能基因所在位置相邻或重叠, 例如, 位于第1染色体bin1.08、bin1.09临近区域的 L28qph1和 PA405qph1, 与已发现的与赤霉素途径有关的基因 Anther ear1( An1)[3]、 Dwarf8[23]相邻; 位于第3染色体bin3.03区域的已克隆株高相关基因 ZmGA3ox2[4]与 Y478qph3、 Z58qph3、 WF322qph3、 PA405qph3、 K12qeh3、 Y478qeh3和 WF322qeh3相邻; Vgt2[24,25] 、Vgt1[26,27,28]所在的bin8.04、8.05区域也覆盖了多个株高、穗位高相关QTL, 如 HYS3qph8、 MO17qph8、 HBqeh8-2、 Q319qeh8和 HYS3qeh8。值得一提的是, Vgt1不仅控制玉米花期, 同时影响玉米株高的形态建成。另外, 位于第9染色体bin9.03区域的 Q319qph9与已克隆的编码细胞色素P450基因 Dwarf3[2]位于同一位置。由此可见, 本研究的多个主效QTL区域已成功克隆到相关的功能基因, 我们推测在bin1.02-1.03、bin9.07等区域, 可能还存在控制株高和穗位高的相关基因, 有待进一步研究。
本研究以具有共同亲本的多重关联RIL群体进行株高和穗位高QTL定位有以下优势, 一方面, 有利于检测可能存在的复等位变异; 另一方面, 可定位到在多个遗传背景下能够稳定表达的QTL区域, 从而显著提高定位结果的可靠性和应用潜力。与双亲作图群体相比, 利用具有共同亲本的多重关联RIL群体进行作图, 可以一次性挖掘更多的目标性状相关QTL区域。同时, 还挖掘到在不同遗传背景下稳定表达的QTL区域, 这些遗传区段更易于应用于目标性状的分子标记辅助选择。
株高和穗位高是玉米形态建成的两个重要指标, 一般认为两者具有很大的关联性。本研究中, 进行了11个RIL群体株高和穗位高表型及遗传相关性分析(表2), 结果表明, 株高和穗位高在各RIL群体内均呈极显著正相关, 而QTL定位结果也表明许多株高和穗位高的主效QTL区域在染色体上相邻或重叠(图2), 进一步显示了两者紧密的遗传联系。严建兵等[29]研究发现, 玉米和水稻在染色体上均存在普遍的QTL富集现象。汤华等[30]的研究也表明数量性状间的表型相关可能源于控制数量性状的QTL之间的紧密连锁。本研究也说明控制株高和穗位高的部分基因可能是相同的(即一因多效), 但也有可能控制株高和穗位高的QTL具有集中分布呈紧密连锁等特点。但需要注意的是, 研究中还检测到一些单独控制株高或穗位高的QTL, 例如 Q319qph9仅控制株高, WF322qeh6仅控制穗位高, 表明株高和穗位高仍具有相对独立的遗传基础, 存在不同的遗传调控机制。
兰进好等[31]研究发现玉米株高和穗位高主要受加性和显性作用的控制(RIL群体中不存在显性作用), 同时受到上位性作用的影响。虽然上位性效应相对单位点分析中的加性和显性效应较小, 但从作用位点的数量上看, 上位性效应的位点大量存在, 而且分布较为广泛。上位性作用在本研究群体中的情况还需进一步分析。
QTL与环境互作(G×E, QEI)可能是由于不同的基因在不同的环境下选择性表达, 以适应不同的生态环境的过程。Li等[32]认为可能存在以下3种情况: (1)某一特定的QTL在某一环境下表达, 而在另一环境下不表达, 不同环境给予其不同的表达信号; (2)某一特定的QTL在某一环境下强表达, 而在另一环境下弱表达, 表明在不同的环境下其效应值会发生改变; (3)某一特定的QTL在不同环境条件下差异性表达, 且在不同环境条件下效应值相反。本研究分别检测到5个株高和4个穗位高相关的能够在多个环境中稳定表达的“环境钝感QTL”, 这些QTL能够在多个环境中被检测到, 具有较小的环境互作效应, 这与前人的研究结果相一致[14,33]。其原因可能 是: (1)解释表型变异较大的QTL, 由于其自身的效应值较大, 与环境互作的效应相对较小, 而效应值较小的QTL与环境的互作, 由于目前统计方法的限制被忽略[34]; (2)株高和穗位高拥有较高的遗传力, 具有相对较小的环境互作效应[35]。QTL与环境的互作在一定程度上影响着性状的表现, 多环境下表达且效应较大的QTL可以作为今后候选基因克隆和分子标记辅助选择的优先位点。本研究检测到的重要环境钝感QTL对进一步的研究具有重要的参考价值。
玉米株高、穗位高是遗传基础较为复杂的农艺性状, 由主效及微效QTL共同控制, 同时存在着广泛的QTL与环境互作和上位性互作。检测到21个株高主效QTL (其中5个为“环境钝感QTL”)和15个穗位高主效QTL (其中4个为“环境钝感QTL”); 挖掘到5个重要的株高和穗位高候选基因区域, 这些区域对玉米株高、穗位高相关QTL的精细定位和克隆有重要参考价值。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|

