α-淀粉酶活性是影响大麦种子发芽和麦芽制作及啤酒酿造的重要性状,
α-淀粉酶属α-1,4-D-葡聚糖水解酶, 作用于淀粉及相关多聚和寡聚糖的1,4-糖苷键, 广泛存在于植物、动物、细菌和真菌中。在大麦中, 根据等电点高低, 将α-淀粉酶分为低等电点Amy1 (p I 4.7~5.0)和高等电点Amy2 (p I 5.9~6.4)两类; 相应地, α-淀粉酶的编码基因也依据其编码酶蛋白的等电点高低分为 Amy1和 Amy2两大基因家族, 分别位于第7和第6染色体上[ 1]。
Amy6-4基因是编码 Amy2的基因家族成员之一, 主要在发芽种子的糊粉层中合成。1985年, 为研究赤霉素(GA)对α-淀粉酶基因表达的影响, Rogers[ 2]根据高等电点α-淀粉酶的cDNA文库和同工酶信号肽序列设计引物, 与大麦糊粉层表达的mRNA杂交, 找出高等电点α-淀粉酶相关mRNA, 经引物延伸克隆出cDNA序列, 通过Southern杂交克隆出全基因序列, 定名为 Amy6-4(GenBank登录号为K02637.1)。
麦芽中的α-淀粉酶是唯一能够最初对天然淀粉颗粒起作用的酶, 其活性直接影响到淀粉的降解, 在麦芽加工和啤酒生产中起重要作用, 因此成为麦芽质量和啤酒大麦品种选育的重要指标。
通过功能基因重测序揭示不同品种或基因型之间的单倍型变异, 进而对等位基因多态性与表型性状进行关联分析, 不仅能寻找功能基因的优良等位变异, 为分子标记选择开发标记, 而且可能发现多功能基因以及用以鉴定多个表型性状的多功能分子标记。2001年, Thornsberry等[ 3]利用92个自交系材料对 dwarf8基因的多态性研究和关联分析发现, 该基因不但影响玉米的株高, 更重要的是有的等位变异还与开花期有关。受这一研究启发, 利用已知基因序列进行基因多样性和功能预测及验证的报道越来越多[ 4, 5]。本研究选用58份大麦种质资源, 根据发表的 Amy6-4基因序列设计引物, 对该基因进行重测序, 并进行单核苷酸多样性和单倍型分析, 试图通过该基因的等位变异与α-淀粉酶活性之间的关联分析, 发现 Amy6-4基因的优良等位变异(突变), 为开发功能标记奠定基础。
58份供试大麦种质中, 56份来自中国的22个省、自治区, 代表不同的生态类型, 包括46份地方品种、5份野生型大麦和5份育成品种。其余2份国外种质来自叙利亚和伊朗(表1)。全部材料由中国农业科学院作物科学所国家农作物种质资源保藏中心提供。
选取每份材料10粒种子, 种于温室, 种子出苗2周后, 剪取嫩叶, 用CTAB法[ 6]提取基因组DNA, 1%的琼脂糖凝胶电泳检测DNA质量, 紫外分光光度计检测DNA浓度。
选取每份材料100粒种子, 置3个铺有湿润滤纸的培养皿中, 于培养箱中15℃暗培养5 d后, 随机取20粒种子, 去除根和芽。采用3,5-二硝基水杨酸法[ 7]测定α-淀粉酶活性。
1.3.1 引物设计 根据NCBI (http://www.ncbi. nlm.nih.gov/nuccore/166994)公布的 Amy6-4基因组序列(GenBank登录号为K02637.1), 与大麦中其他编码α-淀粉酶的同源基因序列比对结果, 选择在非同源区段, 设计覆盖 Amy6-4基因5′ UTR区、编码区和内含子区以及3′ UTR区的4对引物, 用于全长扩增(表2)。引物由北京赛百盛基因技术有限公司合成。
1.3.2 基因扩增与测序 PCR反应总体积为25 μL, 包含基因组DNA 25 ng、dNTP终浓度各0.25 μmol L-1、引物终浓度各10 μmol L-1、HiFi Taq DNA Polymerase (北京全式金生物技术有限公司) 1 U和1×buffer 25 μL。扩增程序为: 94℃预变性5 min; 94℃变性30 s, 退火30 s (温度因引物而异), 72℃延伸 1 min, 38个循环; 72℃终延伸10 min; 4℃保存。扩增产物经凝胶回收试剂盒(TIANGEN)纯化后, 送生工生物工程(上海)有限公司进行双向测序。利用BLAST工具(http://blast.ncbi.nlm.nih.gov/), 将测序结果与GenBank中的序列比对, 以确认扩增结果的正确性。
1.3.3 数据分析 利用Vector NTI Advance 10.0 (Invitrogen, USA)中的Contig-Express组件, 将相互覆盖的序列片段拼接, 用Clustal W1.83软件[ 8]进行多序列比对, 将完成比对的序列保存为PHYLIP格式文件, 采用DnaSP 4.0软件(http://www.ub.es/dnasp)[ 9]进行序列多样性分析及单倍型分析。分别利用 θ值[ 10]和 π值[ 11]估计核苷酸多样性。作为衡量核苷酸多样性的参数, θ值为衡量群体突变的参数, 与核苷酸变异占序列位点的比率有关。而 π值则反映同一位点不同序列间的差异。
根据Tajima[ 12]提出的TajiIma’s D检验, 作中性检验, 以评价 Amy6-4基因是否经受了选择的影响。
1.3.4 系统发育树的构建 使用ClustalW1.83软件进行序列比对, MEGA4.0软件[ 13]邻接法(neighbour- joining)构建系统发育树。
利用覆盖大麦基因组的41个SSR标记, 使用Structure2.0软件[ 14]对58份样品进行群体结构分析。用DnaSP 4.0软件(
将群体结构分析中各个品种的Q值作为协变量, 使用TASSEL 2.1软件(http://www.maizege netics.net/)的一般线性模型(general linear model, GLM)程序, 对测得的α-淀粉酶活性数据与 Amy6-4序列分析的SNP和单倍型数据进行回归分析。
以覆盖 Amy6-4全基因2902 bp的4对引物, 包括起始密码子前1168 bp的5′ UTR区、编码区以及264 bp 3′ UTR区, 对58份大麦种质进行分段扩增和序列比对(图1)。结果只有位于编码区的第3外显子和3′ UTR区存在单核苷酸多态性位点(SNP), 其中在编码区有4个SNP位点, 3′ UTR上存在3个SNP位点(图2), 构成5种单倍型(表3)。
| 表1 58份供试大麦种质的编号、原产地及特性 Table 1 Accession number, origin, and characteristics of 58 barley entries |
| (续表1) 58份供试大麦种质的编号、原产地及特性 Table 1 Continued Accession number, origin, and characteristics of 58 barley entries |
| 表2 用于扩增 Amy6-4基因不同区段的引物 Table 2 Primers for Amplification of different parts of Amy6-4 gene in barley |
Amy6-4基因的第3外显子和3′ UTR存在多样性(图1和图2)。于第3外显子2442 bp处有一个C/G颠换多态性位点, 为一同义突变, 对应于氨基酸序列的第360位点精氨酸(R); 2477 bp位点为A/G转换SNP位点, 为非同义突变, 导致第372位点的氨基酸由天冬酰胺(N)突变为丝氨酸(S); 2587 bp有一个G/A转换SNP位点, 为同义突变, 对应于第405位点甘氨酸(G); 位于2591 bp出现一个G/C颠换SNP位点, 为非同义突变, 导致第410位点的氨基酸由甘氨酸(G)突变为丙氨酸(A)。该基因的3′ UTR区有3个SNP位点, 分别是位于2655 bp的G/A、2657 bp的G/C和2840 bp的G/A多态性位点。在供试的58份大麦材料中, H_3单倍型的出现频率最高, 达51.7% (30/58); 其次为H_1单倍型, 频率为39.7% (23/58); 其他3种单倍型的频率均较低(表3)。
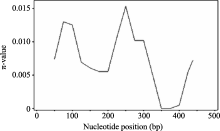
以核苷酸多样性 π值和 θ为指标, 通过对58份大麦材料 Amy6-4基因第3外显子和3′ UTR区的序列多样性研究, 基本明确了相应于 Amy6-4基因全序列, 在长度为475 bp区段内单核苷酸多样性(SNP)分布规律。 π值从2442 bp的0.0075上升至2477 bp的0.001 25, 此后开始下降至0.005, 再从2587 bp到2591 bp迅速升高到最大值0.015 (即在编码区的SNP3), 再次下降; 在3′ UTR区下降到最低值, 接近0后再次升高, 表现为先升后降—再升再降—再升的变化趋势, 而其他区段则相对保守(图3)。
2个多样性参数( θ和 π)的估计值0.005 80和0.007 13, 差异不显著, 表明 θ值和 π值的变化趋势较为一致。
利用 Amy6-4序列中的多态性位点(SNP)的TajiIma’s D检验, 本试验中TajiIma’s D值为0.697 25, 在 P= 0.1水平未达到显著, 说明该基因在进化过程中, 未经受选择作用的影响, 其序列变异为选择中性。
 | 图1 Amy6-4第3外显子和3′ UTR的7个SNP的核苷酸序列(475 bp)比对Fig. 1 Nucleotide alignment with seven SNPs detected in the third exon and 3′ UTR for Amy6-4gene |
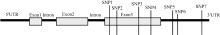 | 图2 Amy6-4结构示意图及在第3外显子和3′ UTR上的多态性位点(SNP)Fig. 2 Schematic structure of Amy6-4 with all detected SNPs, located in the third exon and the 3′ UTR |
| 表3 在58份大麦种质中发现的 Amy6-4的单倍型 Table 3 Haplotypes in Amy6-4 gene among 58 barley entries |
根据58份种质 Amy6-4基因第3外显子和3′ UTR区域SNP变异所构成的5种单倍型, 构建了系统发育树, 发现单倍型H_1与H_4、H_2与H_3、H_5与H_3亲缘关系最近; 其次是单倍型H_1、H_2、H_3和H_4均与H_5较近; H_1和H_4都与H_3较远, 与H_2最远; H_2、H_3和H_5均与H4较远, 与H_1最远(图4)。对各种单倍型品种的来源(表1和表3)分析可知, H_1单倍型的23份种质地域分布较广, 来自中国14个省、自治区; H_2单倍型的3个品种全部是育成品种, 来自中国的浙江和湖北; H_3单倍型的30个品种中, 除2份国外大麦种质外, 其余均为中国青藏高原及其相邻省区的参试品种, 包括西藏8个、四川2个、云南1个、甘肃3个; H_4和H_5单倍型各仅包括1个品种。为分析不同 Amy6-4单倍型大麦的特征特性, 了解5种单倍型在不同类型大麦中的分布, 根据表1数据和图4的聚类结果归纳出表4。可以看出, H_1单倍型包括的23份种质, 全部为皮大麦, 11个春性、12个冬性, 且此单倍型中来自南方的品种, 多为冬性或半冬性品种; H_2单倍型的3份种质也均是皮大麦, 且为育成品种; H_3单倍型的30份材料包括春性23份、冬性7份, 裸大麦17份、皮大麦13份; 单倍型H_4和H_5分别为山东六棱冬大麦(ZDM368)和山西的二棱春大麦品种(ZDM119)。
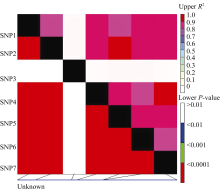
利用58份大麦材料 Amy6-4基因的重测序数据, 分析了该基因碱基序列中7个SNP位点间的连锁不平衡关系, 只有位于第2587 bp的SNP3(G/A)位点与其他6个SNP位点无连锁不平衡(LD)关系(图5)。其余6个SNP位点相互之间, 均存在显著的LD关系( R2=0.82~1.00)。
LD衰减程度对关联分析具有很大的影响。在对 Amy6-4基因第3外显子和3′ UTR区的SNP多态性位点LD分析的基础上, 进一步以 r2作为衡量指标检测该区域的LD衰减程度, 在 Amy6-4基因的多态性区域未检测到明显的LD衰减, r2值在0.8~1.0之间(图6)。表明在该基因的多态性区域内存在较高的锁不平衡度, 适宜进行基于候选基因的关联分析。
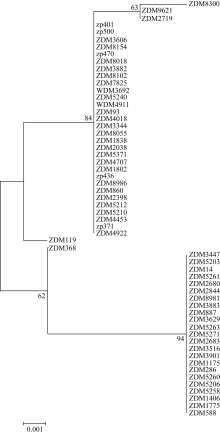 | 图4 依据58份大麦种质 Amy6-4基因SNP多样性的系统发育树Fig. 4 Phylogennetic tree of 58 barley entries according to SNP diversity on Amy6-4locus |
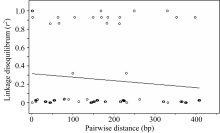 | 图6 Amy6-4基因SNP位点间 r2值随遗传距离衰减散点图Fig. 6 Attenuation of r2values between SNP pairs along with genetic distance on Amy6-4 locus |
| 表4 Amy6-4基因的单倍型及其相应的不同特性样品数统计 Table 4 Haplotype patterns of Amy6-4gene and their affiliation to samples based on different barley traits |
在 Amy6-4基因序列多态性LD及其衰减程度分析的基础上, 使用TASSEL软件的一般线性模型(GLM)程序, 将群体结构分析中 K=6时, 各参试样品Q值作为协变量, 分别对每个SNP和单倍型与实验测定的58份大麦种质的α-淀粉酶活性(表1)进行回归分析。结果显示, 各个SNP位点和各种单倍型, 均与α-淀粉酶活性间无关联性( P>0.05), 表明本研究检测的这些发生在α-淀粉酶编码基因上的等位变异均对酶活性没有影响。
本研究利用58份不同地理来源的大麦种质, 对编码高等电点α-淀粉酶的功能基因 Amy6-4进行了重测序和序列比对分析, 只在该基因编码区的第3外显子和3′ UTR区发现7个SNP位点, 其中4个在编码区, 3个在3′ UTR, 构成5种单倍型, 主要是H_3和H_1单倍型。属于H_1单倍型的23份材料全部为皮大麦, H_3单倍型共30份材料, 多数为中国青藏高原及其邻近省区的大麦种质。据此推测单倍型H3与大麦的皮裸性之间及其与青藏高原大麦之间, 可能存在着某些必然的联系, 值得进一步研究。通过对SNP进行氨基酸序列比对, 发现由外显子上2个同义突变的单核苷酸编码的氨基酸, 分别位于α-淀粉酶蛋白质结构域B的α螺旋(SNP1)及底物结合结构域C (SNP3)。关联分析结果表明, 所发现的7个SNP和5种单倍型, 均与α-淀粉酶活性无关。Robert等[ 15]研究发现, 由品种间单核苷酸变异SNP2或SNP4引发的, α-淀粉酶蛋白质结构域B和结构域C上的氨基酸变化, 对酶蛋白的二级结构并没有影响。本研究结果与Matthies等[ 16]的报道有相近之处, 即位于2487 bp的SNP3和单倍型H_2均与α-淀粉酶活性无关, 但Matthies还发现, SNP3和单倍型H_2与麦芽品质性状, 如浸出率、脆性、可溶性蛋白和黏度等显著关联。根据Bozonnet等[ 17]对蛋白结构的研究结果, SNP3位点突变所编码的同义突变甘氨酸(Gly)残基距离在结合麦芽低聚糖中起关键作用的氨基酸残基(Tyr380)只相隔3个氨基酸残基。本研究中, LD分析结果表明, 只有位于2587 bp的SNP3 (G/A)位点与其他6个SNP位点之间, 没有连锁不平衡关系, 表明该位点处于自由状态。TajiIma’s D检验结果表明, Amy6-4基因序列变异为选择中性, 在进化过程中并未经受选择作用的影响。这一结果与中国20世纪80年代以前大麦育种实践高度相符。本研究绝大多数供试材料为国内种质, 仅有2份国外材料, 在56份国内材料中包括5份育成品种、5份野生大麦和46个地方品种, 其中5个育成品种全部为20世纪60年代中末至70年代初育成。由于α-淀粉酶活性不是直观性选择性状, 且啤酒大麦育种中尚未被提出, 我国早期大麦育种未对该特性加以选择, 因而这5个育成品种与野生大麦和地方品种表现出相同 Amy6-4基因中性选择特点。20世纪80年代以后, 大麦育种目标有了变化, 但由于本试验中未包括近年来育成啤酒大麦品种, 是否存在对 Amy6-4基因的人工选择有待进一步探讨。
在本研究中, 尽管发现大麦品种间α-淀粉酶活性存在较大变异, 但其编码基因 Amy6-4的多态性位点与酶活性间不存在关联性, 说明 Amy6-4基因变异对于α-淀粉酶活性影响较小。这与Yang等[ 18]对限制性糊精酶结构基因的多态性变异与其酶活性之间的关联分析结果一致。β-淀粉酶的形成过程与α-淀粉酶和限制性糊精酶不同, β-淀粉酶主要是在种子发育过程中合成, 而α-淀粉酶和限制性糊精酶均是在种子发芽过程中合成的[ 19]。α-淀粉酶活性作为复杂的数量性状, 具有复杂的遗传机制。在种子发芽过程中, α-淀粉酶基因的表达不仅受到赤霉素(GA)和脱落酸(ABA)的拮抗作用, 而且受到各种调控因子如受赤霉素调控的转录因子GAmyb、ABA诱导的蛋白激酶pKABA以及WRKY38等转录因子的共同作用[ 20, 21, 22, 23]。并且α-淀粉酶在分泌前还要经受一系列的转录后调控, 即α-淀粉酶分泌途径上受到众多因素如GA、细胞内的Ca2+浓度和代谢产生的糖类等因素的调节[ 24, 25, 26, 27]。许多基于双亲杂交后代群体的连锁作图分析和基于LD的全基因组关联分析, 对影响α-淀粉酶活性QTL的研究结果表明, 除大麦3H染色体外, 大麦其他6条染色体上均分布有影响α-淀粉酶活性的QTL[ 28, 29, 30, 31], 进一步表明α-淀粉酶活性是受多基因控制的数量性状。但综合已有对α-淀粉酶活性的定位结果, 认为大麦染色体5H末端的端粒区域可能存在控制α-淀粉酶活性的主效调控基因。可以作为今后研究影响α-淀粉酶活性的主要QTL区域, 进而可能在此区域通过精细定位, 发掘出α-淀粉酶活性的调控因子或主效QTL。
Amy6-4基因的第3外显子和3′ UTR区共存在7个SNP位点, 其中编码区有4个, 3′ UTR区有3个, 共构成5种单倍型。单倍型H_3的出现频率最高, 其次为单倍型H_1, 其余3种频率很低。 Amy6-4基因的7个SNP及其构成的5种单倍型均与α-淀粉酶活性无关。
| 1 |
|
| 2 |
|
| 3 |
|
| 4 |
|
| 5 |
|
| 6 |
|
| 7 |
|
| 8 |
|
| 9 |
|
| 10 |
|
| 11 |
|
| 12 |
|
| 13 |
|
| 14 |
|
| 15 |
|
| 16 |
|
| 17 |
|
| 18 |
|
| 19 |
|
| 20 |
|
| 21 |
|
| 22 |
|
| 23 |
|
| 24 |
|
| 25 |
|
| 26 |
|
| 27 |
|
| 28 |
|
| 29 |
|
| 30 |
|
| 31 |
|