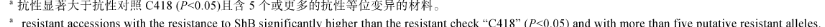采用苗期微室接种鉴定法, 用144个分布于水稻全基因组的多态性标记, 利用TASSEL软件GLM (Q)、MLM (Q+K)和MLM (PCA+K) 3种模型对456份水稻材料组成的自然群体进行纹枯病抗性关联分析。结果发现, 有13个标记位点至少在2种模型中均被检测到与纹枯病抗性显著关联, 单个位点可解释表型变异的1.84%~8.42%; 其中10个标记位点位于以往报道的连锁定位的抗纹枯病QTL附近, 3个标记位点(RM1036、RM5371和RM7585)是未曾报道的新的抗病相关位点。抗性等位变异RM7585-150对纹枯病发病病级减效效应最大; 有259份材料携带抗性等位变异RM5371-129, 占供试材料总数的56.8%, 只有26份材料携带抗性等位变异RM1036-82, 占供试材料总数的5.7%。水稻纹枯病发病病级与其含有的抗性等位变异数量呈极显著的负相关。本研究结果将为水稻抗纹枯病分子标记辅助育种提供理论依据。
To identify and map sheath blight (ShB) resistance loci in rice, we carried out association analysis of 456 rice accessions using 144 genome-wide markers based on GLM (Q), MLM (Q+K), and MLM (PCA+K) models of TASSEL software. The phenotyping were assayed at the seedling stage with a micro-chamber screening method. The results showed that thirteen markers were significantly associated with ShB resistance detected by using at least two models, which explained from 1.84% to 8.42% of the phenotypic variance. In addition, ten of the identified resistant loci were either quite near or within the interval of previously identified QTLs. RM1036, RM5371, and RM7585 were novel resistant loci that had not been previously reported. The resistant allele 150 of RM7585 showed the largest negative effect to ShB rating, allele 129 of RM5371 existed in 259 (56.8%) of 456 rice accessions, and 82 of RM1036 existed in 26 (5.7%) rice accessions. The number of putative resistant alleles presented in rice was highly and significantly correlated with the decrease of ShB rating. The resistant alleles identified in this study are readily available and can be exploited for marker-assisted selection.
水稻纹枯病是由立枯丝核菌( Rhizoctonia solaniKühn)侵染引起的一种世界性水稻病害, 造成水稻巨大的产量损失和品质降低[ 1]。培育和种植抗病品种是防治水稻纹枯病最经济有效的方法之一。水稻对纹枯病的抗性属于典型的数量性状[ 2], 利用传统的遗传分离群体定位水稻纹枯病抗性QTL, 操作繁琐, 耗时耗力。虽然国内外利用传统的遗传分离群体已定位了30多个水稻抗纹枯病QTL[ 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12], 但迄今为止, 仍然没有一个抗纹枯病基因被克隆, 除少数几个抗纹枯病QTL被精细定位外[ 3, 4], 大多数为初步定位结果[ 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12]。未见抗纹枯病QTL定位成果应用于分子标记辅助选择育种。
关联分析(association mapping)是利用不同基因座等位变异间的连锁不平衡(linkage disequilibrium, LD)关系, 分析标记与性状的相关性, 以达到鉴定特定目标性状基因的目的。与连锁分析相比, 关联分析可以利用具有丰富遗传多样性的自然群体为材料, 无需构建专门的作图群体, 并可实现同时检测自然群体同一座位的多个等位基因, 对QTL定位的广度和精度也相对较高[ 13]。鉴于这些优势, 关联分析被广泛应用于发掘植物数量性状基因的研究[ 14, 15, 16], 成为植物遗传学研究的热点。水稻拥有非常丰富的种质资源, 是进行关联分析的理想材料[ 15], 但利用关联分析方法进行中国水稻稻种资源对纹枯病抗性基因定位的研究目前未见报道。
准确的表型鉴定是关联分析的前提。水稻纹枯病的抗性鉴定易受环境温度、湿度及水稻株高和生育期等的影响。至今虽然已研发了一些较有效的大田成株期纹枯病菌接种和抗性鉴定的方法, 但其工作量大、周期长, 难以用于大规模且农艺性状差别大的自然群体的接种鉴定[ 17]。苗期微室接种鉴定法(micro-chamber method, MCM)由Jia等[ 18]于2007年建立, 以水稻幼苗为接种对象, 试验周期短, 环境相对可控, 结果具有更好的重复性和可靠性。目前这一新方法已被成功地应用于水稻纹枯病抗病基因表达和QTL定位研究[ 6, 9]。
本研究采用苗期微室接种鉴定法, 对456份水稻材料组成的自然群体进行纹枯病抗性关联分析, 以期定位水稻抗纹枯病相关的基因位点, 发掘各关联位点的抗性等位变异及优异的抗病种质材料, 为水稻抗纹枯病分子标记辅助育种提供理论依据。
456份水稻材料中, 235份是来自中国23个省(自治区)的地方品种, 193份是中国常规育成品种包括现有生产上主要的恢复系材料等, 28份来自菲律宾、美国、澳大利亚、日本、印度、越南、泰国7个国家。C418和特青是抗病对照, Lemont是感病对照, 武育粳3号较感纹枯病[ 4, 17, 19]。
供试水稻纹枯病菌为强致病力菌株GD118, 由江西省农业科学院植物保护研究所提供。将稻谷、稻壳按1∶2混合, 浸泡24 h后分装培养皿。灭菌后接种纹枯病菌, 28℃培养5~7 d, 待谷粒表面长满菌丝后用作接种物备用。参考Jia等[ 18]的苗期微室法接种鉴定。为了消除因品种间秧苗发育进程不同而造成的抗病性差异, 根据前期不同品种发芽和秧苗发育进程试验结果, 将456份供试水稻材料分4批浸种催芽, 待种子出芽后, 从中选择芽长一致的点播到装有灭菌土的塑料盆(直径13 cm)中生长。待各品种的大部分植株处于3~4叶时, 通过间苗去除叶龄有差异的单株和弱株, 保留生长势和叶龄基本一致的3株植株。然后用镊子将带菌谷粒2粒置于紧挨植株基部两侧, 确保带菌谷粒与幼苗茎基部紧密接触, 保持土表湿润不积水。接种后马上将去除底部的2 L透明塑料瓶罩在幼苗上, 底部入土, 上口去掉瓶盖, 形成微室保湿。控制室内温度为26~30℃, 相对湿度为80%~85%。接种后5~7 d, 待感病对照Lemont的大多数植株死亡时调查各单株病级。病级=病斑高度/株高×9级[ 17]。其中病斑高度为最高病斑的上界距土表的高度, 株高为秧苗最高叶尖距土表的高度。试验重复4次。用软件SPSS 19进行各品种病级差异显著性单因素方差分析和LSD法多重比较。
提取水稻材料叶片DNA。从520对SSR标记中挑选多态性较好且均匀分布于12条染色体上的142个SSR标记和2个InDel标记, 对456份水稻材料进行PCR扩增。扩增产物经8%的聚丙烯酰胺凝胶电泳检测, 银染显色。
用POWERMARKER3.25软件[ 20]统计标记位点的等位变异及其频率和多态信息含量PIC。用NTsyspc2.2 软件完成供试材料的主成分(PCA)分析。用STRUCTURE2.2软件[ 21]完成供试材料的遗传结构分析。用SPAGeDi软件[ 22]计算个体间亲缘关系系数Kinship。使用TASSEL2.1软件[ 23]的GLM (general linear model)和MLM (mixed linear model)模型, 分别以各材料Q值、Q+Kinship值和PCA+ Kinship值作为协变量进行关联分析。参考Breseghello等[ 14]和文自翔等[ 24]的方法计算标记位点等位变异的表型效应。在每一个关联位点的等位变异中, 对纹枯病发病病级减效效应最大的等位变异被认为是该位点的“抗性等位变异”[ 16]。
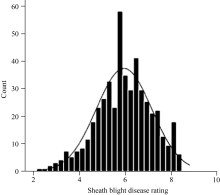
采用环境相对可控的苗期微室法及相对病斑高度进行抗性评价, 能有效地降低环境和形态学因素的干扰影响。结果表明接种纹枯病菌后, 感病对照品种Lemont严重发病, 平均病级8.24±0.12, 武育粳3号为6.89±0.25; 抗病对照品种C418发病轻, 平均病级3.98±0.52, 特青为5.06±0.43, 品种抗感性与前人描述基本一致[ 17, 25]。456份供试水稻材料接种纹枯病后发病病级在各个等级均有分布, 抗病性变异较丰富, 且基本呈正态分布(图1), 适合做纹枯病抗性关联分析。病级最低为2.49 ± 0.45, 最高为8.86±0.29, 平均6.18±0.37。有44份(9.65%)材料的抗性显著大于抗性对照品种特青, 其中18份(3.95%)材料的抗性显著大于抗性对照品种C418 ( P<0.05)。
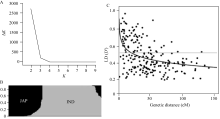
用144个标记共检测到916个等位基因, 变化范围为2~18个, 平均多态信息含量PIC值为0.58。剔除遗传距离较近的标记, 用89个标记分析该自然群体的遗传结构。当 K=2时, Δ K值最大(图2-A), 推断该群体分为2个亚群, 分别包含277份和179份材料(图2-B)。144个标记位点的共线性组合和非共线性组合中都存在着极显著的LD ( P<0.01)。 D'值回归遵循方程 y = -0.0803 ln ( x) + 0.726 ( R2=0.201), 当 D'值取0.5时, LD衰减距离为16.7 cM, 适合做水稻的关联分析(图2-C)。
关联分析中, 群体分层(population stratification)被认为是引起假阳性的最主要因素[ 26], 而加入群体结构和亲缘关系控制的混合线性模型MLM (Q+K)及加入主成分分析和亲缘关系控制的混合线性模型MLM (PCA+K)均能够较好地去除由群体分层引起的假阳性[ 27, 28]。因此本试验采用GLM (Q)、MLM (Q+K)和MLM (PCA+K) 3种模型对144个标记位点与水稻纹枯病病级进行关联分析。结果发现, 有13个标记位点在MLM (Q+K)或MLM (PCA+K)任一模型中被检测到与供试群体纹枯病病级显著关联( P<0.05), 分布于8条染色体上, 其中第6染色体上4个, 第11染色体上3个, 第3、第4、第5、第8、第10和第12染色体上各1个, 单个位点可解释纹枯病病级变异的1.84%~8.42% (表1)。其中11个标记位点在3种模型中均被检测到。RM6917、RM311和RM1233标记在3种模型中均达到极显著水平( P<0.01), 对表型变异解释率分别为3.89%、4.49%和3.63%; 有10个标记位点位于以往报道的连锁定位的抗纹枯病QTL附近(±5 cM)[ 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 16]。
13个与纹枯病抗性显著关联的位点共有54个主要的等位变异(等位变异频率≥0.05), 每个关联位点均表现为既有减效等位变异又有增效等位变异(图3和表1)。减效等位变异减轻纹枯病发病病级即增加抗性, 反之, 增效等位变异增加纹枯病发病病级即降低抗性。在每一个关联位点的等位变异中, 对纹枯病发病病级减效效应最大的等位变异被认为是该位点的“抗性等位变异”[ 16]。其中, 抗性等位变异RM1036-82是13个关联位点的抗性等位变异中平均病级最低的, 其次是RM6297-149; RM 7585-150对纹枯病发病病级减效效应最大, 其次是RM311-162和RM224-118; 有259份材料携带抗性等位变异RM5371-129, 占供试材料总数的56.8%; 有255份材料携带抗性等位变异RM7585-150, 占供试材料总数的55.9%; 携带抗性等位变异RM267-159或RM1036-82的材料数量较少, 只有29份和26份, 分别占供试材料总数的6.4%和5.7%。
| 表1 与纹枯病抗性显著相关的标记位点及各位点的主要等位变异 Table 1 Marker loci significantly associated with sheath blight resistance and their major alleles |
水稻材料纹枯病病级与其含有的抗性等位变异数量呈极显著的负相关 ( r = -0.502, P<0.0001) (表2)。水稻品种霸王鞭和大叶棵同时含有9个抗性等位变异, 是含有抗性等位变异最多的材料。456份供试水稻材料中, 有27份含有5个或更多的抗性等位变异, 而且抗性显著大于抗性对照特青( P<0.05), 占材料总数的5.9%; 其中10份水稻材料抗性还显著大于抗性对照C418, 占材料总数的2.2%。这些水稻材料是优异的抗病种质材料。13个抗性等位变异在10份含5个或更多的抗性等位变异且抗性显著大于抗性对照C418的水稻材料中的分布见表3。
目前被精细定位的水稻抗纹枯病QTL仅有2个。Zuo等[ 4]利用染色体片段置换系将 qSB-11LEQTL精细定位于74 kb区间内, Channamallikarjuna等[ 3]利用重组自交系群体将 qSBR- 11-1精细定位于850 kb区间内, 其余抗纹枯病QTL均为初步定位结果。在将定位结果应用于育种实践以前, 必须验证QTL真实性。由于传统的遗传分离群体的局限性, 研究者通常只能通过设置重复或在不同年份/地点多次试验, 根据结果的重演性来判断QTL的真实性, 费时费力。
本研究采用144个分布于全基因组的多态性标记, 对456份水稻材料组成的自然群体进行纹枯病抗性关联分析。10个抗病标记位点与以往报道的抗病QTL的侧翼标记相同或位于以往报道的抗病QTL区间内, 包括RM267[ 2, 5, 6]、RM190[ 6, 16]、RM6917[ 2, 13]、RM209[ 3, 7]、RM1233[ 3, 8, 16]、RM224[ 3, 8]、RM6297[ 2, 11, 12]、RM6838[ 2, 8]、RM8242[ 2]、RM311[ 7], 进一步验证了这些QTL的真实性, 暗示这10个抗病标记位点在不同的遗传背景或环境中能够稳定表达, 育种利用价值可能更高。
本研究在第11染色体上关联到3个标记, 其中RM1233和RM224之间0.4 Mb, 已精细定位的 qSB-11LEQTL位于该区间内[ 3, 8]; 在第6染色体上关联到4个标记, 其中RM190和RM6917两者之间2.3 Mb, RM190为以往已报道的抗病QTL[ 6, 16]的侧翼标记, RM6917位于抗病QTL区间内[ 2, 13], 说明抗病标记RM190和RM6917真实性较高, 可能是精细定位水稻抗纹枯病基因的目标区域。
3个抗病位点RM1036、RM5371和RM7585是未曾报道的新的抗病基因位点。RM1036距离RG241a-RZ397区间的抗纹枯病QTL[ 12]约3.0 Mb, RM5371距离RM30-RM439区间的抗纹枯病QTL[ 7]约1.4 Mb, RM7585距离Y1065Le-RZ69x区间的抗纹枯病QTL[ 2]约1.8 Mb。这3个标记经过进一步验证将有可能成为水稻抗病性分子辅助选择新的有效标记。
| 表2 13个抗性等位变异在456份水稻种质资源中的分布 Table 2 Distribution of the 13 putative resistant alleles among 456 rice accessions |
| 表3 10份优异的抗病种质材料纹枯病病级及含有的抗性等位变异 Table 3 Distribution of sheath blight disease ratings and putative resistant alleles among the ten resistant accessions |
采用传统育种手段对由数量基因控制的病害的抗性改良, 难度较大。分子标记辅助育种已被认为是抗病育种的一个可行途径[ 29]。但迄今未见抗纹枯病QTL定位成果应用于分子辅助选择育种。本研究通过对13个与纹枯病抗性显著相关的标记位点的等位变异分析发现, 每个位点上有多个等位变异, 每个等位变异效应不同。极显著相关性表明( r = -0.502, P<0.0001), 含有的抗性等位变异数量越多, 供试水稻材料对纹枯病的抗性也越高。这些结果表明, 利用分子标记辅助选择, 聚合不同位点的抗性等位变异, 可以提高水稻纹枯病抗性。因此, 聚合不同的遗传背景中能稳定表达的RM267-159、RM190-118、RM6917-154、RM209-153、RM1233-160、RM224-120、RM6297-149、RM6838-104抗性等位变异, 可能是提高水稻纹枯病抗性水平的一个有效途径。
不同水稻品种对纹枯病的抗性存在明显的差异, 但至今未发现免疫和高抗的品种。目前研究中的抗纹枯病水稻亲本多局限于少数几个公认的抗病材料如“特青”或“Jasmine 85”等。而事实证明, 育种中对少数抗性亲本材料的广泛使用, 造成推广品种遗传背景狭窄、作物病虫害种类演变, 重要病虫害持续发生或流行[ 30]。因此迫切需要挖掘新的抗病种质材料, 发现新的抗病基因或QTL, 以拓宽水稻纹枯病抗性遗传背景。本研究发现27份水稻材料含有5个或更多的抗性等位变异且抗性显著大于抗性对照特青, 它们是优异的抗病种质材料, 可以作为水稻抗病育种的供体亲本加以利用。
发现13个标记位点与纹枯病抗性显著关联, 单个位点可解释表型变异的1.84%~8.42%; 附近, 3个标记位点(RM1036、RM5371和RM7585)是未曾报道的新的抗病相关位点。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|