* 通讯作者(Corresponding author): 禹山林, E-mail:yshanlin1956@163.com
第一作者联系方式: E-mail:chenna7948@163.com, Tel: 0532-87626672
以花生品种花育33为试材, 根据cDNA文库中已知的果糖-1,6-二磷酸醛缩酶(fructose-1,6-bisphosphate aldolase, FBA) 基因全长序列设计引物, 通过RT-PCR克隆到该基因, 命名为
In this article, a fructose-1,6-bisphosphate aldolase (FBA) gene was cloned from the leaf of peanut (
可溶性糖是高等植物叶片光合作用的产物, 它不仅可以作为新陈代谢的能源, 还可以作为调节物质参与植物生长发育相关的多种信号[ 1, 2]。有研究表明可溶性糖的增加可以提高植物对干旱、高盐和低温等的抗性[ 3]。在适应多变的环境, 满足代谢和能量的要求中, 植物进化出一系列机制去特异的感知多种“糖信号”。通过拟南芥糖信号突变体的研究, 目前发现植物体内存在己糖激酶信号途径、依赖己糖但不依赖己糖激酶的信号途径、蔗糖信号途径(包括蔗糖载体的信号途径)、依赖膜的信号途径和果糖信号途径等糖信号途径[ 4, 5]。
果糖1,6-二磷酸醛缩酶既存在于糖酵解也存在于糖异生的途径中, 同时还存在于磷酸戊糖循环中, 因此果糖-1,6-二磷酸醛缩酶对细胞生命活动起着至关重要的作用。近几年研究表明, 植物的果糖-1,6-二磷酸醛缩酶与多种胁迫具有密切相关的联系。Yamada等[ 6]发现在一种耐盐的烟草中, 存在2种核编码的叶绿体定位果糖-1,6-二磷酸醛缩酶(NpAldP1和NpAldP2), 在高盐胁迫时, 后者在植物体内发生积累而前者有轻微下降。对拟南芥盐胁迫处理后取其根部, 进行蛋白质组学研究, 发现果糖-1,6-二磷酸醛缩酶的蛋白含量明显增加[ 7]; 利用拟南芥悬浮细胞系也可以得到类似的结果[ 8]。Provart等[ 9]利用12个低温敏感突变体进行表型鉴定及基因芯片检测, 其中3个低温致死突变体有类似的表达谱, 它们的果糖-1,6-二磷酸醛缩酶基因的表达量都明显上调。Lu等[ 10]分析拟南芥中编码果糖-1,6-二磷酸醛缩酶的基因, 找到8个该家族基因( AtFBA1-8), 并证明它们对ABA、NaCl、重金属镉、温度和干旱胁迫等具有不同的响应模式, 除 AtFBA3外, 其余基因在根中均对干旱胁迫有明显响应。非生物刺激如海水、氯化钠、ABA和PEG, 在2~12 h内可能诱导海马齿根中 spFBA基因表达量显著增加; 过表达 SpFBA基因能够增强转基因大肠杆菌的耐盐性, 这些结果表明, SpFBA在对盐胁迫的反应和非生物刺激方面起着非常重要的作用[ 11]。此外, Chen等[ 12]研究指出拟南芥果糖-1,6-二磷酸醛缩酶基因的表达与硼胁迫相关。以上研究结果表明, 果糖-1,6-二磷酸醛缩酶基因的表达在转录水平或翻译水平上受多种非生物胁迫的调控, 研究其在各种非生物胁迫下的表达对于揭示果糖-1,6-二磷酸醛缩酶基因的功能具有重要意义。
不利的生长环境对花生产量有很大的影响。然而有关花生中非生物胁迫调控的分子生物学研究较少, 处于刚刚起步的阶段。目前一些基因已经被证明可能参与花生非生物胁迫的调控并得到克隆[ 13, 14, 15, 16, 17]。Wan等[ 18]和Wang等[ 19]以花生基因转化拟南芥证明花生中泛素连接酶和Germin-like家族蛋白可能参与拟南芥对干旱或高盐胁迫的调控。此外, 未见有关花生非生物胁迫分子机制研究的更深入报道。本研究通过序列分析从花生已有cDNA文库中找到一个编码果糖-1,6-二磷酸醛缩酶的基因, 根据该基因已知序列通过RT-PCR方法从花生品种花育33中克隆到全长基因。拟通过该基因功能的分析, 进一步了解其对非生物胁迫的响应, 为花生抗性分子调控研究提供参考。
选用花生品种花育33, 将种子播在营养土与蛭石(质量比2︰1)的混合土中, 萌发后在光照16 h /8 h黑暗(28/22℃)的培养箱中生长, 待幼苗生长至三叶期后, 置4℃光照培养箱进行低温处理; 另将花生幼苗从土中拔出, 小心冲洗干净根部土后直接浸泡到200 mmol L-1 NaCl、20% PEG-6000或100 μmol L-1 ABA溶液中。在各处理0、1、3、6、12、24、48和72 h后分别取叶片和根, 液氮冷冻保存, 作为后续试验材料。
总RNA提取试剂盒、TOP10感受态购自天根生化科技有限公司。LA Taq DNA聚合酶, pMD18-T载体, 荧光定量PCR用SYBR Premix Ex Taq聚合酶购自大连宝生物。M-MLV反转录酶购自Promega。凝胶回收试剂盒、质粒提取试剂盒购自Omega公司。
用天根的RNeasy Mini Kit, 参考其使用说明提取样品总RNA。合成cDNA前要将提取的RNA用DNaseI处理以去除DNA污染。用M-MLV反转录酶进行cDNA的合成, 每25 μL反应体系中加入2 μg RNA; 反转录反应体系在42℃进行1 h, 之后置冰上冷却5 min。
根据cDNA文库中已知的花生蔗糖合成酶的全长序列, 设计扩增该基因全长的引物, 引物序列为: 5°-AGGGGAT AAGATTCTTGTGT-3°和5°-TAGCCTTCCATTCCATCTTG TT-3°。以反转录cDNA为模板, 通过普通RT-PCR扩增该基因。PCR程序为: 94℃ 2 min; 94℃ 30 s, 55℃ 30 s, 72℃ 1 min, 35个循环; 72℃ 10 min。
将所测得的序列利用NCBI网站(http://www.ncbi.nlm. nih.gov/)上的BLAST工具进行基因序列的相似性及同源性查找, 并利用这些序列进行基因同源性的比较, 用http://www.ncbi.nlm.nih.gov/网站ORF Finder在线分析cDNA全长序列ORF。根据所测得基因的cDNA序列推导其氨基酸序列, 并利用序列分析工具对其进行分析: 采用Protparam (http://web.expasy.org/protparam/)预测蛋白的基本物理化学性质; 在http://pfam.sanger.ac.uk/上分析蛋白的结构功能域; 采用PredictProtein (http://www.predictp-rotein.org/)预测蛋白的二级结构; 利用ProtComp (http:// nux1.softberry.com/berry.phtml?topic=protcomppl&group= programs&subgroup=proloc)分析蛋白质的亚细胞定位情况。
选取GenBank中不同物种的果糖-1,6-二磷酸醛缩酶(FBA)蛋白, 采用DNAMAN软件的Multiple Alignment进行多序列比对分析, 创建一个不同来源的FBA多序列的比对结果; 在比对的基础上用MEGA 4.0软件完成系统发生和进化分析, 采用Neighbour-Joining方法构建系统树。
进行荧光定量PCR时, 先将cDNA样品稀释到8 ng μL-1, 每反应体系中加2 μL稀释后的cDNA。采用Roche的LightCycler 2.0荧光定量PCR仪。反应条件为 95℃ 10 s; 95℃ 5 s, 60℃ 30 s, 72℃ 10 s; 40个循环; 绘制溶解曲线, 温度每10 s升高0.5℃。内参基因为 Actin11, 每个样品重复3次, 取平均值, 采用delta-delta Cp方法分析数据, 误差线为3次重复的标准偏差。荧光定量所用 Actin11引物为5°-TTGGAATGGGTCAGAAGGATGC-3°和5°-AGTGGT GCCTCAGTAAGAAGC-3°; AhFBA1引物为5°-TTGATAG TGGTTATGGATATGA-3°和5°-TTAAGTTGTAGAGGTGA AAGT-3°。
通过序列分析从已有cDNA文库中获得花生果糖-1,6-二磷酸醛缩酶的基因全长, 根据已知序列设计引物, 通过RT-PCR扩增得到该基因。该基因全长为1489 bp, 开放阅读框ORF为1200 bp, 编码400个氨基酸(图1)。在NCBI网站上对该基因编码的蛋白进行Blast分析, 发现该蛋白与大豆( Glycin max)、苜蓿( Medicago truncatula)、可可树( Theobroma cacao)和葡萄( Vitis vinifera)中的果糖-1,6-二磷酸醛缩酶蛋白同源性达95%以上(图2)。将该基因命名为 AhFBA1 ( A rachis h ypogaea F ructose-1, 6- b is - phosphate a ldolase), 并登录于NCBI, 登录号为KF773841。
利用ProtParam分析显示, AhFBA1的理论分子量为43.03 kD, 理论等电点为8.25, 平均亲水系数(grand ave-rage of hydropathicity, GRAVY)是-0.173; 通过蛋白质亚细胞定位工具预测, 这个蛋白可能定位于叶绿体中, 其中N端1~33个氨基酸为叶绿体定位信号肽。蛋白质二级结构预测结果显示, AhFBA1中α螺旋占42.25%, β折叠占12.50%, 无规则卷曲占53.04%; 保守结构域预测结果表明该蛋白有Glycolytic (fructose-bisphosphate aldolase class-I)保守结构域。
根据花生、大豆、葡萄、可可树、苜蓿和水稻中的果糖-1,6-二磷酸醛缩酶蛋白氨基酸序列比对, 绘制了系统进化树。图3表明, 果糖-1,6-二磷酸醛缩酶的进化基本符合植物进化分类, 花生AhFBA1与豆科植物包括大豆、菜豆、鹰嘴豆、红车轴草及苜蓿中的果糖-1,6-二磷酸醛缩酶聚在一起, 说明它们具有较近的亲缘关系; 与其他双子叶植物如葡萄、番茄、蓖麻、可可树、拟南芥等的FBA蛋白则相距较远。与单子叶植物水稻、小麦、二穗短柄草和谷子的果糖-1,6-二磷酸醛缩酶相距更远。相近物种或同一物种的同一家族蛋白在进化上亲缘关系更近, 例如紫花苜蓿和蒺藜苜蓿中果糖-1,6-二磷酸醛缩酶, 大豆中的果糖-1,6-二磷酸醛缩酶FBA1和FBA2以及拟南芥中的果糖-1,6-二磷酸醛缩酶FBA1和FBA2。单子叶植物的果糖-1,6-二磷酸醛缩酶聚在一起, 双子叶植物的果糖-1,6-二磷酸醛缩酶聚在一起, 反应了同源果糖-1,6-二磷酸醛缩酶系统发生上的密切关系。
通过荧光定量PCR分别检测了花生根和叶中该基因在低温、高盐及干旱胁迫下的表达情况(图4)。
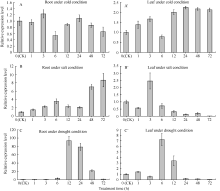 | 图4 低温、高盐和干旱胁迫下 AhFBA1基因在花生根和叶中在的表达模式分析Fig. 4 Expression analysis of AhFBA1in root and leaf under abiotic stresses |
从图4可以看出, AhFBA1在转录水平上的表达对低温、高盐及干旱胁迫均有响应, 但响应模式及响应组织有所差异。 AhFBA1的表达在花生根中对低温没有响应, 随着低温处理时间的延长, 其表达量基本不变(图4-A); 在花生叶中 AhFBA1对低温略有响应, 在低温处理24 h后表达量上调2倍以上(图4-A’)。 AhFBA1在花生根中对高盐胁迫有明显响应, 在花生根中盐处理3 h后表达量即上调2倍以上, 72 h表达量达最高, 最高上调倍数在8倍以上(图4-B); 在花生叶中 AhFBA1在盐处理3 h时上调, 上调倍数约2.5倍, 但随着处理时间的延长表达量开始下降, 72 h表达量下调了近100倍(图4-B’)。干旱胁迫下 AhFBA1在花生叶和根中均有明显响应, 在花生根中在干旱胁迫处理6 h后开始明显上调, 12 h表达量达到最高, 最高上调倍数为90倍以上, 72 h表达量基本恢复对照水平(图4-C); 在花生叶片中 AhFBA1的表达也受到干旱胁迫的明显诱导, 在干旱胁迫处理6 h时表达量最高, 与对照相比, 表达倍数在7倍以上(图4-C’)。
总的来说, AhFBA1在花生根和叶中对低温胁迫响应均不很明显, 在花生根中对高盐和干旱胁迫响应明显, 尤其是在干旱胁迫下, 上调倍数非常高。
研究表明, 胁迫响应基因可能通过ABA-dependent和ABA-independent两种信号途径调控。为了研究非生物胁迫诱导的 AhFBA1表达与ABA的关系, 我们用外源ABA分别对花生根和叶进行处理后通过荧光定量PCR检测 AhFBA1基因的表达。结果表明, AhFBA1的表达在花生根和叶中均受ABA的诱导(图5)。在花生根中 AhFBA1在ABA处理3 h后表达量明显上调, 至48 h上调倍数近30倍, 72 h表达量仍维持在较高水平(图5-A)。在花生叶片中, AhFBA1的表达也受ABA处理的诱导, 且在处理3 h后表达量就最高, 处理至12 h和24 h后略有降低, 此后又有所增高(图5-B)。
低温、高盐和干旱等非生物胁迫可以影响花生萌发、生长、开花、产量等[ 20, 21]。鉴定参与花生非生物胁迫调控的关键基因, 是通过基因工程技术培育高产抗逆花生种质的基础。目前, 花生基因组测序工作尚未完成, 序列拼接与组装工作尚未开始, 仍未获得大量功能基因全长序列, 因此功能基因的克隆与鉴定仍是目前花生中非生物胁迫研究工作的重要组成部分。
果糖-1,6-二磷酸醛羧酶催化果糖-1,6-二磷酸、二羟丙酮磷酸和三磷酸甘油醛的可逆反应, 是卡尔文循环中固定CO2后第1个催化C3化合物转化为C6化合物的酶, 处于第1个分支点上, 同时它也是控制光合作用速率的重要酶之一[ 22]。果糖-1,6-二磷酸醛缩酶对细胞生命活动起着至关重要的作用[ 23]。本研究表明, 花生中的果糖-1,6-二磷酸醛缩酶与大豆、葡萄和苜蓿等植物中的果糖-1,6-二磷酸醛缩酶同源性非常高(图2), 在进化树中与豆科植物聚在一起(图3), 表明该蛋白在植物进化过程中非常保守, 同时也暗示了其在功能上的保守性。
亚细胞定位预测结果显示, 花生果糖-1,6-二磷酸醛缩酶基因定位于叶绿体中, 其N端1~33个氨基酸为叶绿体定位信号肽。在植物细胞中至少存在2种果糖-1,6-二磷酸醛缩酶的异构形式[ 24]。他们由部分同源的基因编码, 因此他们的结构不同并且定位在不同的亚细胞部位, 分别是细胞质和叶绿体[ 25, 26]。前人研究表明, 水稻中细胞质定位的果糖-1,6-二磷酸醛缩酶基因包含2个外显子, 而叶绿体定位的果糖-1,6-二磷酸醛缩酶基因包含6个外显子, 与我们的预测结果类似, 水稻叶绿体定位的果糖-1,6-二磷酸醛缩酶N端编码一个叶绿体定位的信号肽(46个氨基酸)[ 25, 26]。细胞质中的果糖-1,6-二磷酸醛缩酶主要催化1,6-二磷酸果糖生成磷酸二羟丙酮和3-磷酸甘油醛反应, 提高糖酵解途径, 因此在ATP的生成方面具有重要功能, 并且此过程不需要氧气的参与[ 27, 28, 29, 30, 31]。与之相反的是, 质体定位的醛缩酶主要是催化磷酸二羟丙酮和3-磷酸甘油醛生成1,6-二磷酸果糖。这个反应是光合碳还原循环中的重要步骤, 同时受光的调节, 因此在光合作用碳同化方面具有重要功能[ 6, 32]。可见, 对于果糖-1,6-二磷酸醛缩酶基因来说, 亚细胞定位对于其功能起着决定性作用, 下一步还需要通过瞬时表达系统对AhFBA1的确切亚细胞定位情况验证。
研究表明果糖-1,6-二磷酸醛缩酶基因尤其是叶绿体定位的果糖-1,6-二磷酸醛缩酶在植物非生物胁迫抗性调控方面起着关键作用。在盐胁迫的小麦中果糖二磷酸醛缩酶蛋白含量明显增加[ 33], 然而用重金属镉离子处理拟南芥后, 发现植物体内的果糖-1,6-二磷酸醛缩酶蛋白含量明显减少[ 34]。在烟草中过表达叶绿体型果糖-1,6-二磷酸醛羧酶基因可提高其耐盐性, 脯氨酸含量也会增加[ 11, 35]。Purev等[ 36]克隆到一种果糖-1,6-二磷酸醛缩酶基因 ClAldC, 在缺氧、过氧化氢、冰冻条件下表达量升高, 但在盐胁迫条件下表达量变化不明显。Yamada等[ 6]在一种耐盐烟草中分离到2种果糖-1,6-二磷酸醛缩酶, 其氨基酸序列相似性高达91%, 但在盐胁迫条件下其中一种果糖-1,6-二磷酸醛缩酶基因的表达量明显升高, 另一种的表达量略有降低; 在另外2种烟草中发现这2种果糖-1,6-二磷酸醛缩酶基因在盐胁迫下表达量都降低。Zhang等[ 37]在对盐藻的研究中发现其果糖-1,6-二磷酸醛缩酶基因在高盐胁迫条件下表达量升高, 其机制与果糖-1,6-二磷酸醛缩酶参与糖酵解并合成甘油来调节盐藻细胞内渗透压有关。可见, 果糖-1,6-二磷酸醛缩酶家族成员对不同的非生物胁迫有不同的响应方式。研究其在各种非生物胁迫下的表达模式有助于阐明基因功能, 为后续研究奠定重要的分子基础。荧光定量PCR结果表明, 花生果糖-1,6-二磷酸醛缩酶 AhFBA1基因在花生叶片中对低温略有响应, 在花生根中对盐胁迫有明显响应, 在花生根和叶片中对干旱胁迫均有明显响应(图4)。根据该结果, 我们推测花生 AhFBA1可能参与花生对非生物胁迫尤其是高盐和干旱胁迫的抗性调控。同时我们还发现对于低温胁迫, AhFBA1的表达在叶片中的响应强于根中; 而对于干旱和高盐胁迫, A h FBA1的表达在根中的响应强于叶片中。推测出现这种结果的原因可能是因为低温胁迫中叶片直接与外界环境接触, 而在NaCl和PEG6000处理中, 根直接与处理液接触。
此外, 植物对非生物胁迫的逆境适应性调控路径有ABA-dependent的方式和ABA-independent的方式[ 38], 说明果糖-1,6-二磷酸醛缩酶的表达受ABA的调控。Osakabe等[ 39]发现拟南芥 RPK1T-DNA插入突变体在种子萌发、幼苗生长和气孔关闭等方面表现出对ABA不敏感的表型, 进一步研究表明大多受到ABA上调的基因在该突变体中表达量下降, 其中包括果糖-1,6-二磷酸醛缩酶基因表达量也下调; Abe等[ 40]发现超表达 AtMYC2和 AtMYB2的拟南芥转基因植株对ABA更加敏感, 可能是通过上调 rd22基因, 同时很多ABA响应基因在转基因植株中也上调, 其中包括了果糖-1,6-二磷酸醛缩酶基因。本研究证明, 花生 AhFBA1的表达在花生根和叶片中均受外源ABA的诱导, 花生果糖-1,6-二磷酸醛缩酶可能是以ABA-dependent的方式调控花生对非生物胁迫的抗性。
本研究初步证明了花生果糖-1,6-二磷酸醛缩酶基因的表达受各种非生物胁迫的诱导, 可能参与花生非生物胁迫抗性调控过程。下一步将通过转基因手段将该基因转入花生或拟南芥中, 详细分析该基因在花生对非生物胁迫适应性中的功能, 进一步研究其可能的作用机制。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|






