植物叶色变化对叶绿体发育和叶绿素生物合成等光合系统结构和调控机制的研究有着重要的理论意义。水稻叶缘白化突变体
The research on the color change of plant leaf is very important to clarify the structure and mechanism of photosynthetic system, such as chloroplast development and chlorophyll biosynthesis. A novel rice mutant
叶片是植物最主要的光合器官, 光合作用主要发生在叶绿体上, 叶绿体是植物细胞所特有的半自主性细胞器, 也是叶绿素、脂类、淀粉和氨基酸的合成场所[ 1, 2]。叶绿素是参与光合作用的重要色素, 其含量的显著变化会导致叶色的变化, 所以叶绿素缺陷突变体也称为叶色突变体[ 3]。叶色突变的主要特点是叶色表型变异, 其突变来源于控制叶绿素生物合成和叶绿体发育的重要基因沉默或失活, 直接或间接影响叶绿素合成和降解, 改变叶绿素含量[ 2, 4, 5, 6, 7, 8]。近年来, 大量研究结果显示, 叶色突变体具易于观察的突变性状, 是开展光合系统的结构和功能、叶绿素生物合成及其调控机制研究的理想材料[ 9, 10]。同时, 叶色可作为标记性状应用于良种繁育和杂交育种, 也可作为观赏稻应用于休闲观光农业[ 11]。
基于叶色的变化, 水稻叶色突变可分为白化、黄化、浅绿、白翠、绿白、黄绿、绿黄、条纹等类型。其中, 白化是植物叶色突变体的常见类型之一, 其最典型特征是叶绿体不能正常发育, 叶绿素合成受阻。关于水稻叶色突变体的基因定位报道最早见于1975年[ 12]。随着分子标记技术的发展和广泛应用, 目前水稻中被定位的叶色基因至少有80多个, 遍布于除第12染色体外的所有染色体(http://www.gramene.org/)。但迄今为止, 只有部分基因被成功克隆。例如, 编码Mg2+螯合酶3个亚基的 OsChlH、 OsChlD和 OsChlI[ 8, 9, 10, 11, 12, 13], 编码叶绿素酸脂 a加氧酶的 OsCAO1和 OsCAO2[ 14], 编码叶绿素合酶的 YGL1[ 15], 编码核糖核苷酸还原酶大亚基蛋白RNRL1和小亚基蛋白RNRS1的 Virescent3和 Stripe1[ 16], 鸟苷酸激酶基因 Virescent2[ 17], 叶绿素 b还原酶基因 NYC1[ 18], 持绿突变体基因 sgr[ 19], 联乙烯还原酶基因 OsDVR[ 20]。最近也报道了2个水稻叶色相关基因 FGL[ 21]和 VYL[ 22], 分别编码原叶绿素酸脂氧化还原酶B和叶绿体Clp亚基, 参与叶绿体的发育和叶绿素的生物合成。
本研究利用EMS诱变恢复系缙恢10号, 从其后代中获得一份稳定遗传的叶缘白化突变体 mal( marginal albino leaf), 该突变体在整个生育期过程中, 叶片边缘均呈白化表型。遗传分析表明该突变体受隐性核基因控制, 利用BSA法最终将其定位在第8染色体, SSR标记M22和InDel标记ID27之间, 物理距离为171 kb。
mal来自EMS诱变恢复系缙恢10号( Oryza sativa L. ssp. indica), 经过多代自交, 突变性状稳定遗传。2011年, 用表型正常的不育系材料西农1A与突变体 mal杂交, 同年在海南种植F1, 并收获F2种子, 于2012年在西南大学水稻研究所分别种植亲本和F2群体。
上午9:00, 在种植小区中间随机选择长势相对一致的野生型和突变体各5个单株, 分别测定苗期和抽穗期的光合色素含量。称取0.01 g叶片剪碎装入离心管, 加25 mL丙酮﹕无水乙醇(1∶1, v /v)混合液封口暗处理24~48 h, 其间经常摇动, 直到叶片完全变白为止, 重复3次。参考Lichtenthaler的方法计算[ 23]。
在植株成熟后, 分别选择缙恢10号和 mal突变体小区中间10株, 考查株高、有效穗、每穗粒数、每穗实粒数、千粒重、结实率等主要农艺性状, 同时测量各节间的长度。
在抽穗期, 随机选取长势相对一致的单株各5株, 在天气晴朗的上午8:30开始, 利用LI-6400型便携式光合作用测定仪测定突变体的光合特性、叶绿素荧光动力学参数, 重复3次, 取平均值, 以缙恢10号为对照。叶绿素荧光动力学参数包括初始荧光( Fo)、PSII光适应下的最大量子产额 Fv°/ Fm°、非光化学淬灭系数( qN)、表观光合电子传递速率(ETR)[ 24], Fv′/ Fm′=( Fm′- Fo′)/ Fm′, qN=( Fm- Fm ')/ Fm, ETR= ΦPSII× PAR×0.84×0.5。
参照何瑞峰等[ 25]的方法用电镜观察突变体和野生型叶片的细胞结构。以戊二醛和锇酸双重固定后, 利用不同梯度的乙醇逐级脱水, 再置换和包埋, 制超薄切片, 以醋酸双氧铀和柠檬酸铅液双重染色, H600型透射电镜观察并照相。
采用BSA[ 26]法定位目标基因, 即根据F2植株表型, 分别选取10株正常单株和10株突变单株, 剪取等量叶片, 构建正常基因池和突变基因池。按CTAB法[ 27]提取亲本和基因池DNA, 采用碱煮法提取F2群体DNA[ 28]。参照http://www.gramene.org/microsat/ SSR引物序列, 根据西农1A和和缙恢10号的序列差异比对设计InDel引物, 均由上海英骏技术公司合成。PCR反应总体系为25 μL, 包括2.5 μL 10×PCR buffer, 1.3 μL 25 mmol L-1MgC12, 1.0 μL 2.5 mmol L-1 dNTPs, 16.0 μL ddH2O, 2.0 μL 10 μmol L-1引物, 2.0 μL模板DNA, 0.2 μL 5 U μL-1 Taq DNA聚合酶。PCR程序为94℃预变性5 min; 94℃变性30 s, 55℃退火30 s, 72℃复性1 min, 35个循环; 72℃延伸 10 min。PCR产物经10%的非变性聚丙烯酰胺凝胶电泳, 快速银染后观察[ 29]。
F2定位群体中, 将具有西农1A带型的单株记为A, 具有 mal突变亲本带型的单株记为B, 具有杂合带型的单株记为H。根据公式[( H+2 A)/2 n]×100计算遗传距离并构建连锁图谱, 其中 H表示定位群体中出现杂合体带型单株的数量, A表示出现西农1A正常带型的单株数, n表示用于定位的隐性群体总株数。
突变体 mal从苗期开始叶片边缘就呈现白化, 一直持续到成熟(图1-A~E), 抽穗后, mal的3片功能叶的白化面积自上而下依次增大, 即剑叶白化部分
最少。与野生型相比, mal突变体植株显著变矮, 株高仅为野生型的76.26% (表1), 各节间长度都显著减小(图1-F~G)。 mal突变体叶片也明显变窄, 剑叶、倒二叶、倒三叶的宽度分别仅为对照的82.16%、47.16%和76.51% (表2), 倒二叶和倒三叶的长度也显著变短。此外, mal突变体的有效穗数、每穗粒数、每穗实粒数、千粒重与对照相比均显著降低, 分别降低了31.58%、55.75%、26.82%和4.10%, 而结实率和主穗长则没有明显变化(表1)。
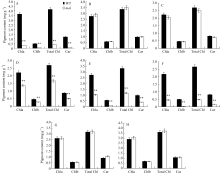
在苗期, 白化比较严重, mal突变体与野生型相比, 叶绿素a、叶绿素b、总叶绿素、类胡萝卜素含
| 表1 野生型(WT)与突变体( mal)的农艺性状 Table 1 Performance of agronomic traits in the wild type (WT) and mutant ( mal) |
量均极显著降低。抽穗期, 由于 mal三片功能叶自上而下白化部分面积逐渐增多, 剑叶和倒二叶白化部分相对较少, 因此与野生型相比, mal突变体剑叶和倒二叶各光合色素含量无明显差异, 倒三叶白化部分相对较多, 从而导致各光合色素含量极显著降低。与野生型相比, mal突变体倒二叶边缘、倒三叶边缘各光合色素含量均极显著降低, 而倒二叶中部、倒三叶中部无显著差异(图2)。
| 表2 野生型(WT)与突变体( mal)的叶片形态分析 Table 2 Leaf phenotypic traits in the wild type (WT) and mutant ( mal) |
与野生型相比, 突变体 mal的初始荧光( Fo)、表观光合电子传递速率(ETR)没有差异, 光适应下的最大量子产额 Fv′/ Fm′、非光化学猝灭效率( qN)均极显著高于野生型(表3), 表明突变体PSII反应中心光能转换效率和原初光能捕获效率较高, 但是其天线色素吸收的光能大部分以热形式散失导致其光能利用率较低。
光合特性比较表明, 突变体 mal的净光合速率( Pn)、气孔导度( Gs)极显著低于野生型, 而细胞间CO2浓度( Ci)极显著高于野生型, 蒸腾速率与野生型无差异(表4)。 mal光合色素均极显著降低可能导致净光合速率降低, 引起光合作用原料CO2和H2O积累, 从而最终导致细胞间CO2浓度升高。
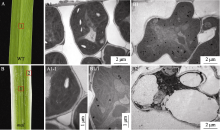
野生型和 mal突变体叶片绿色部分叶肉细胞发育完全, 细胞器均匀分布, 叶绿体规则地贴壁分布, 完整地被膜包裹, 基质浓厚, 基质片层有序排列(图3-A1, B1, A1-1, B1-1)。而 mal突变体叶片白色部分的叶肉细胞发育严重不良, 大部分中空, 无明显完整的细胞器, 叶绿体内部基本完全降解(图3-B2)。说明 MAL基因突变后, 叶片绿色部分的叶肉细胞发育及其叶绿体的结构并未被影响, 而白色部分叶肉细胞及叶绿体发育受到严重抑制。
| 表3 野生型(WT)与突变体( mal)叶片的叶绿素荧光动力学参数 Table 3 Chlorophyll fluorescence kinetic parameters of the wild type (WT) and the mutant ( mal) |
| 表4 野生型(WT)与突变体( mal)叶片的光合特性 Table 4 Photosynthetic characteristics of the wild type (WT) and the mutant ( mal) |
用表型正常的不育系西农1A与 mal杂交, F1表型正常, 说明该突变体受隐性基因控制。F2代群体中出现明显的分离, 分别表现双亲性状, 其中正常单株4100株, mal突变单株1325株。经卡方测验, 正常株∶突变株符合3∶1分离比(χ2=0.69<χ20.05=3.84), 表明 mal突变体受隐性单基因控制。
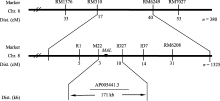
选用西农1A× mal杂交的F2群体作为定位群体, 共获得1325个突变单株, 用于基因定位。在F2代群体中分别选取10株正常株和10株突变株构建正常基因池和突变基因池。选用400个均匀分布于12条染色体上的SSR标记对亲本西农1A和 mal进行多态性分析, 进一步利用在两亲本间表现出多态性的引物, 扩增正常基因池和突变基因池, 并用在基因池间检测到的多态性引物对F2代定位群体中随机选取出的380株突变单株进行单株验证, 结果位于第8染色体的SSR标记RM1376、RM310、RM6429和RM7027与 mal突变位点表现连锁, 分别有33个交
换株、17个交换株、40个交换株及53个交换株, RM1376交换株包含RM310交换株, RM7027交换株包含RM6429交换株, 且RM1376、RM310交换株与RM6429、RM7027交换株互不重叠, 因此将 MAL基因定位在标记RM310与RM6429之间。在两标记间进一步设计了4对SSR引物和30对InDel引物, 其中M22和ID27在两亲本间表现出多态性, 分别有3个和10个交换株, 且互不重叠, 最终将 MAL定位在SSR标记M22和InDel标记ID27之间, 遗传距离分别为0.22 cM和0.73 cM, 物理距离为171 kb (图4)。该区间共有预测基因33个, 其中有7个F-box结构域蛋白, 6个假定蛋白, 3个表达蛋白, 3个逆转录转座子蛋白, 1个生长素输出载体, 1个三角状五肽, 1个水合酶蛋白, 1个淀粉合成酶III, 1个磷酸核糖甘氨酸连接酶, 1个自噬相关蛋白, 1个乙二醛酶家族蛋白, 1个tRNA合成酶, 1个聚异戊二烯合成酶, 1个溴区包含蛋白, 1个赤霉素敏感基因 gar 2, 1个YL1类蛋白, 1个抗病蛋白, 1个突触后蛋白CRIPT。
| 表5 第8染色体上的4个连锁标记 Table 5 Four polymorphic markers used in the fine mapping on chromosome 8 of rice |
国内外针对自然突变或诱变的水稻叶绿素合成
缺陷突变体, 从叶绿素含量与组成、光合能力、叶绿素荧光特性、叶绿体前体物质、叶绿体超微结构、类囊体膜蛋白、突变性状的遗传、突变基因的定位以及突变机理等方面进行了大量的研究, 但研究结果往往不尽相同[ 15, 30, 31, 32, 33]。据不完全统计, 目前报道的水稻叶色突变相关基因已经超过80个, 其中部分基因已进行了染色体定位, 在被定位的基因中只有少数基因被克隆。在各种叶色变异中, 白化突变体较容易发生, 其叶绿体发育往往受阻, 不能进行有效的光合作用, 直至死亡。近年来对白化突变体及其控制基因的研究取得了很大进展。日本学者Iwata等[ 34]早在1975年就报道了11份白化突变体, 分别命名为 al1-al11。通过三体法, 将 al1定位在第6染色体上, al2、 al3及 al6在第5染色体上, al5和 al7在第4染色体上, al4和 al8在第1染色体上, al10在第3染色体上。随着分子标记技术的发展, 人们开展了一系列基因分子图谱定位研究, 程世超等[ 35]将白化致死突变体 abl4定位在第4染色体上, 余庆波等[ 36]将白化突变体 alb21定位在第3染色体上。目前定位在第8染色体上的叶色突变体有失绿突变体 chl8和 chl9[ 37], 表现为整个生育期叶片均呈黄绿色; 白化转绿突变体 v8[ 38], 苗期叶片几乎完全白化, 移栽后转白绿色, 中脉和长出的穗子也出现白斑纹; 斑马叶突变体 z4[ 38], 苗期叶片出现白色或黄色斑马纹, 呈不规则黄带; 温度钝感型淡绿叶突变体 pgl2[ 39], 从移栽到抽穗期, 叶片呈苍白的绿色, 半矮化, 分蘖少, 生育期延迟; 返绿突变体 al12[ 40], 在24℃以下三叶期之前呈白化, 三叶期后返绿。 mal的定位区间为171 kb, 与这些定位在第8染色体上的叶色突变基因不在同一区间。另外, 通常情况下, 大部分白化苗缺乏叶绿素, 不能正常进行光合作用, 幼苗依靠种子中的胚乳营养而生长, 当胚乳耗尽时植株就会死亡, 表现出苗期致死效应[ 41]。而还有一些白化相关突变体的突变性状只在苗期表达, 后期可以恢复正常, 光合色素含量的变化和叶色变化相一致, 因而其突变对主要农艺性状无显著影响[ 42]。本文报道的 mal突变体整个生育期都边缘白化, 绿色部分叶绿素含量与野生型一致, 白化部分叶绿素含量显著低于野生型, 叶片绿色部分细胞结构完整, 叶绿体发育完全, 但其白化部分细胞中空, 叶绿体内部几乎完全降解, 叶肉细胞及其叶绿体发育严重受阻, 最终导致植株生长缓慢, 植株变矮, 有效穗数、每穗粒数、每穗实粒数、千粒重等农艺性状显著降低。因此, 不管是表型分析结果还是基因定位结果均表明 mal是一个新的叶色突变体。
目前已分离的与叶片白化相关的基因较少。白化转绿基因 GRA( t)[ 43]定位在第2染色体上, 通过精细定位和基因测序, 发现该基因编码叶绿体蛋白合成延伸因子Tu。低温失绿基因 CISC( t)[ 44]定位在第9染色体, 编码一个具有三角状五肽重复区(PPR)蛋白, 突变体在该基因的第60位碱基处缺失了8个碱基, 造成移码突变而失去正常功能。 V2属于核基因, 编码鸟苷酸激酶, 催化质体和线粒体内鸟苷酸代谢途径中GMP转化为GDP, 基因突变后叶绿体早期发育受阻。 virescent 2是温敏型突变体, 在20℃条件下, 叶片呈现出病态白化; 在30℃条件下, 叶片几乎恢复正常[ 17]。 V3编码核糖核苷酸还原酶的大亚基RNRL1, 在 virescent 3突变体中, 幼苗期的第1~3叶呈正常绿色, 之后生长的叶片几乎完全白化, 抽穗后叶片转为正常绿色, 同时, 该突变体白化表型受温度影响明显[ 16]。 mal突变体被定位在第8染色体M22和ID27标记之间, 物理距离为171 kb。在该定位区间内, 共有33个候选基因, 并未发现已知的叶色基因存在。我们也对其中可能参与植物生长和叶色变化的三角状五肽基因和生长素基因测序, 发现这两个基因的编码框和启动子在序列上并未发生改变, 推测 mal白化表型可能是由其他基因突变所致。因此, mal属于一个新的水稻叶色突变体, 可能受一新基因调控, 该基因的克隆将为水稻叶色机理研究奠定基础, 同时 mal突变体也可作为作物遗传育种的优良种质资源。
mal突变体在整个生育期叶片均呈边缘白化, 光合色素含量和净光合速率显著降低, 叶片变窄, 株高、有效穗数、每穗粒数、每穗实粒数与千粒重极显著降低。 mal突变体叶片绿色部分细胞及其叶绿体发育完全, 而白化部分细胞结构严重缺陷, 叶绿体结构降解。该突变性状受1对隐性核基因控制, 该基因被定位在第8染色体SSR标记M22和InDel标记ID27之间, 遗传距离分别为0.22 cM和0.73 cM, 物理距离为171 kb, 是一个新的叶色突变基因。
**
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|