通过化学诱变获得遗传稳定的水稻黄绿叶突变体
A yellow-green leaf mutant
植物叶色是叶绿体中各种色素的综合表现, 正常叶片中叶绿素占优势, 通常表现为绿色[ 1]。自然或人为诱变导致叶绿素合成直接或间接相关的基因发生突变, 造成叶绿素合成或降解受阻的一类突变体被称为叶色突变体。大多数突变基因在苗期开始表达, 通常表现黄化、白化、浅绿、条纹、斑点等症状。在大麦[ 2]、小麦[ 3]、拟南芥[ 4]、水稻[ 5]、油菜[ 6]等植物中均见到叶色突变, 其诱变频率较高且易于鉴定。利用这类突变体, 可以帮助人们更好地了解植物光合作用、叶绿素生物合成、光化学反应调节, 以及光合系统构建等相关机制。此外, 还可以考虑将其作为标记性状应用于遗传育种。
水稻是世界重要的粮食作物之一, 也是单子叶模式植物。至今, 水稻中已报道80多个与叶色突变相关的基因(http://www.shigen.nig.ac.jp/rice/oryzabase/ genes/traitGeneClasses.jsp)。叶色突变的机制主要包括叶绿素生物合成途径相关基因的突变, 血红素生物合成途径中的基因突变, 编码其他叶绿体蛋白的基因突变, 与光合系统无直接关系的基因突变等。从谷氨酰-tRNA到叶绿素a和叶绿素b的整个生物合成过程需要15步反应, 涉及15种酶, 在拟南芥中已成功克隆了编码这15种酶的27个基因[ 7, 8]。在水稻中, 已克隆了编码参与叶绿素生物合成的9个基因, 即第3染色体上编码Mg2+-螯合酶3个亚基的 OsChlH、 OsChlD和 OsChlI基因[ 9, 10]以及编码联乙烯还原酶的 OsDVR基因[ 11], 第4染色体上编码原叶绿素酸酯氧化还原酶A的 OsPORA基因[ 12], 第5染色体上编码叶绿素合酶的 YGL1基因[ 13], 第10染色体上编码叶绿素酸酯a加氧酶的 OsCAO1和 OsCAO2基因[ 14]以及编码原叶绿素酸酯氧化还原酶B的 OsPORB基因[ 12]。
本研究通过EMS诱变获得1份水稻黄绿叶突变体 ygl80, 其整个生育期均表现为黄绿叶, 利用( ygl80/浙辐802) F2群体实现了该黄绿叶突变基因的分子标记定位。精细定位和测序结果表明, 编码叶绿素合酶的 YGL1 ( LOC_Os05g28200)基因在编码区第5027碱基处(位于第14外显子), 碱基C突变为碱基T, 使编码蛋白的氨基酸序列第348位的脯氨酸(Pro)突变成亮氨酸(Leu), 推测该基因是造成 ygl80黄绿叶突变性状的候选基因。
10079为来自于粳稻品种日本晴( Oryza sativa L. subsp. japonica, cv. Nipponbare)的一个迟熟突变品系, 由中国科学院遗传与发育生物学研究所储成才研究员和方军博士提供。用化学诱变剂EMS处理10079, 获得一份黄绿叶突变体 ygl80, 经过多代自交, 突变体表型稳定遗传。将 ygl80分别与野生型亲本10079和籼稻品种浙辐802杂交, 在四川成都分别播种亲本、F1和F2, 从苗期开始观察叶色表现。成熟期调查突变体 ygl80和野生型亲本10079的株高、有效穗、穗长、每穗着粒数、结实率、千粒重等主要农艺性状, 每个样本3次重复(小区), 调查样本数15株。
参照Lichtenthaler[ 15]的方法, 在大田苗期和孕穗期, 分别取 ygl80突变体及其野生型亲本的上部叶片, 每个材料重复3次, 每个重复从同样的5个植株上混合取0.2 g叶片, 剪碎浸泡在15 mL的80%丙酮溶液中, 于4℃避光浸提48 h, 中间震荡数次, 最后定容至25 mL。用紫外分光光度计(UV-1700)测定提取液在663、646和470 nm波长下的吸光值。叶绿素a含量(mg g-1) = (12.21A663 - 2.81A646) V / 1000 W, 叶绿素b含量(mg g-1) = (20.13A646 - 5.03A663) V/ 1000 W, 类胡萝卜素含量(mg g-1) = (1000A470 - 3.27Ca - 104Cb) V / (229 × 1000 W), 式中, V为叶绿素提取液总体积(mL), W为叶片鲜重(g), Ca = 12.21A663 - 2.81A646, Cb = 20.13A646 - 5.03A663。
根据叶色调查与分析结果, 从 ygl80与浙辐802杂交所得F2群体中, 选取20株正常绿苗和627株黄苗构建成定位群体, 分单株提取总DNA。从F2植株中随机选取10株正常单株和10株突变单株剪取等量叶片, 将正常绿苗和黄绿苗单株叶片分别混合, 构建正常基因池和突变基因池。用McCouch等[ 16]的方法提取水稻叶片中的DNA。
参照http://www.gramene.org/microsat的SSR引物标记, 并运用Primer5.0软件自行设计插入缺失
(InDel)标记。SSR和InDel引物由北京六合华大基因科技股份有限公司合成。参照Panaud等[ 17]的PCR反应体系, 扩增产物经3.0%~4.0%琼脂糖凝胶电泳和溴化乙锭染色后, 用凝胶扫描成像系统记录电泳分离结果。用MapMaker3.0软件进行遗传作图, 用Kosambi函数将重组率转化为遗传距离(cM), 构建目标基因所在区域的分子标记连锁图谱。
利用水稻基因组注释数据库数据(http://rice. plantbiology.msu.edu/), 根据 ygl80突变体的突变性状, 以及分子标记与目标基因的遗传距离和物理距离, 在突变基因精细定位的染色体区段上遴选 ygl80突变体的候选基因。设计预测基因的引物序列, 分别对野生型和突变体基因组中预测基因序列进行扩增、测序和序列比对, 最终确定突变位点和候选基因。
ygl80突变体生育期均为黄绿色, 明显区别于其野生型亲本10079 (图1)。在成熟期, 与其野生型亲
 | 图1 突变体 ygl80(右)与其野生型亲本10079(左)在苗期(A)及拔节期(B)植株形态Fig. 1 Plant phenotype of ygl80(right) and its wild-type parent 10079 (left) at the seedling (A) and elongation (B) stages |
本相比, ygl80突变体的株高、每株有效穗数、每穗着粒数、穗长和千粒重分别减少14.8%、16.5%、21.3%、9.1%和7.4%, 其他农艺性状差异不显著(表1)。
ygl80在苗期和孕穗期的叶绿素a含量分别为0.89 mg g-1和0.81 mg g-1, 分别比野生型亲本降低72.36%和49.69%; 叶绿素b含量分别为0.07 mg g-1和0.13 mg g-1, 分别降低92.13%和71.74%; 类胡萝卜素含量分别为0.42 mg g-1和0.30 mg g-1, 分别降低53.85%和41.18% (表2), 表明 ygl80的突变性状主要由光合色素含量下降引起。
| 表1 突变体 ygl80与其野生型亲本10079主要农艺性状的比较 Table 1 Comparison of major agronomic traits between the ygl80 mutant and its wild-type parent 10079 |
| 表2 苗期和孕穗期 ygl80突变体与其野生型亲本10079叶片光合色素含量的比较 Table 2 Comparison of photosynthetic pigment contents between the ygl80 mutant and its wild-type parent 10079 during seedling and booting stages |
将 ygl80分别与野生亲本10079和籼稻品种浙辐802杂交, F1植株均表现为正常绿叶, F2代群体中正常绿苗与黄绿苗分离明显, 分离比例经卡方(χ2)测验符合3∶1 (表3), 表明 ygl80的突变性状由1对隐性核基因控制。
以 ygl80与浙辐802杂交F2群体为定位群体, 用350对较均匀分布于水稻12条染色体上的SSR引物对亲本浙辐802和 ygl80的DNA片段进行扩增, 然后用在两亲本间表现出多态的引物扩增正常绿苗和黄绿苗植株近等基因池, 再用在池中检测到多态的引物分别扩增F2植株。结果发现, 位于水稻第5染色体长臂的SSR标记RM5140和RM430与目标基因存在连锁关系, 遗传距离分别为8.4 cM和11.9 cM (图2-A)。为了进一步定位目标基因, 根据粳稻日本晴和籼稻9311序列差异进一步设计多对InDel标记(表4), 最终将 ygl80定位在InDel标记C2和C3之间, 遗传距离分别为0.24 cM和0.39 cM (图2-B), 物理距离约为90 kb (图2-C)。
| 表3 突变体 ygl80与正常绿色品系杂交F2的叶色分离 Table 3 Segregation of leaf color in F2 population from the crosses between ygl80mutant and green leaf varieties |
| 表4 本研究开发的多态性InDel标记 Table 4 Developed polymorphic InDel markers in this study |
叶绿素由一个含Mg的卟啉环和一条长的碳侧链(叶绿醇)组成, 以蛋白复合物的形式存在于叶绿体内, 在光捕获和反应中心的能量转移中扮演着重要角色[ 18, 19, 20]。高等植物的叶绿素分为两类。它们唯一的区别在卟啉D环上, 叶绿素a是一个甲基而叶绿素b是一个醛基[ 21]。通过叶绿素循环叶绿素a和叶绿素b可以相互转化, 一方面叶绿素酸酯a由叶绿素酸酯a加氧酶和叶绿素合酶依次催化形成叶绿素b, 另一方面叶绿素b由叶绿素b还原酶和7-羟甲基-叶绿素a还原酶依次催化形成叶绿素a[ 21, 22, 23, 24, 25]。在叶绿素循环中, 叶绿素合酶可以利用叶绿素酸酯、牛
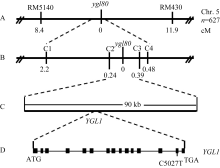 | 图2 ygl80黄绿叶基因在水稻第5染色体长臂上的分子连锁图Fig. 2 Linkage map of the ygl80yellow-green leaf gene on the long arm of rice chromosome 5 |
| 表5 水稻第5染色体定位区间内基因及其推测功能 Table 5 Genes and their putative functions in the target interval |
儿基牛儿基二磷酸或植基焦磷酸作为底物, 催化叶绿素酸酯形成叶绿素a或叶绿素b, 但不能利用细菌叶绿素酸酯作为底物, 这可能与叶绿素合酶独特的基序同底物叶绿素酸酯的结合有关[ 13, 26, 27]。
在水稻、燕麦和拟南芥等高等植物中, 编码叶绿素合酶的基因已被鉴定[ 13, 28, 29]。在水稻中, 编码叶绿素合酶的 YGL1基因被定位于第5染色体长臂上, 是一个单拷贝基因, 全长约5.8 kb, 有15个外显子和14个内含子, cDNA全长1672 bp, 编码由376个氨基酸组成的酶蛋白。 YGL1基因相应的突变体 ygl1是籼稻品种镇恢249的一个自然突变体, 苗期叶片黄化, 中期慢慢转绿, 后期叶色以及叶绿素和类胡萝卜素的含量接近野生型[ 13, 30]。 ygl1的单碱基突变发生在第8外显子上, 由碱基T突变为碱基C, 造成编码的氨基酸序列在198位上的脯氨酸(Pro)突变成丝氨酸(Ser)[ 13, 30]。本研究表明, 经EMS化学诱变而来的 ygl80突变体在整个生长发育期均表现为黄绿叶, 该突变体在编码叶绿素合酶的 YGL1 ( LOC_Os05g 28200)基因的编码区第5027碱基处(位于第14外显子), 碱基C突变为碱基T, 使编码蛋白的氨基酸序列第348位的脯氨酸(Pro)突变成亮氨酸(Leu), 故突变基因是已报道的编码叶绿素合酶的 YGL1基因的等位基因。
通过比对水稻叶绿素合酶与不同物种叶绿素合酶AtCHLG、AsCHLG、SCHLG、RcbchG、RsbchG、HmbchG和CabchG的氨基酸序列, 发现第348位的脯氨酸位点在所有物种中是非常保守的, 而第198位的脯氨酸位点在水稻、燕麦和拟南芥等高等植物中是保守的, 但对所有物种, 相比而言较为不保守[ 13]。因此, ygl80突变体和 ygl1突变体表型的差异, 可能是由于 YGL1基因序列突变位点不同, 引起所编码的叶绿素酶活性降低程度不同造成的。
水稻黄绿叶 ygl80突变体与野生型相比, 叶片叶绿素含量下降, 株高、每株有效穗数、每穗着粒数、穗长和千粒重减少。 ygl80的突变性状由1对隐性核基因控制。该突变基因被定位在第5染色体长臂上90 kb区间内, 是由编码叶绿素合酶的 YGL1 ( LOC_Os05g28200)基因编码区第5027碱基突变而来。该基因是已报道的水稻 ygl1黄绿叶突变基因的等位基因, 不同的突变位点导致 ygl80和 ygl1存在部分表型差异。 ygl80的发现对丰富和深入理解水稻叶色形成和调控的机制, 及丰富水稻特异种质资源具有一定的意义。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|


