作者联系方式: E-mail: panyinghong@caas.cn, Tel: 010-82105849
植物表型分析是理解植物基因功能及环境效应的关键环节, 随着植物功能基因组学和作物分子育种研究的深入, 传统的表型观测已经成为制约其发展的主要瓶颈, 而高通量的植物表型组分析技术和植物表型组学研究是解决这一困境的有效途径。虽然植物表型组分析正在成为国内外研究的热点, 相关概念仍然较为模糊, 阻碍了这一新兴学科的发展。本文分析了植物表型组和植物表型组学的相关概念和范畴, 引入了准表型组、可辨识性状、映射性状、植物表型的遗传和环境包容性等新概念, 将植物表型组定义为“受基因组和环境因素决定或影响的, 反映植物结构及组成、植物生长发育过程及结果的全部物理、生理、生化特征和性状”, 将植物表型组学定义为“对植物表型组信息及相关环境参数的综合控制、完整采集和系统分析”, 并提出了植物表型组学的研究范围、研究方向和顶层设计原则。
Plant phenotyping is a key link in understanding gene function and environmental effects, and with development of plant function genomics and crop molecular breeding, the traditional phenotypic observation has become the main bottleneck. High-throughput plant phenome analysis technology and plant phenomics study is an effective way to solve this problem. Although plant phenome analysis is becoming a hot spot at home and abroad, relevant concepts are still relatively fuzzy, and this situation hinders the development of this emerging discipline. In this paper, the relevant concepts and categories of plant phenome and plant phenomics were analyzed, and the new concepts such as quasi-phenome, identifiable traits, mapped traits, and tolerance of plant phenotype to the changes of inheritance and environment, were introduced. And, plant phenome was defined as “all of physical, physiological and biochemical characteristics and traits which are decided or influenced by genome and environments, and can reflect the plant structures and compositions, or reflect the processes and results of plant growth and development”, and plant phenomics as “the comprehensive controls, complete collections and systematic analyses of plant phenome informations and related environmental parameters”. The scopes, directions, and top design principles of plant phenomics research, were also discussed.
认识植物的表型特征和性状是生物学研究的一个重要命题, 缺乏详尽的表型数据, 将无法深刻理解基因组和环境因素对植物表型的复杂作用。另一方面, 确保全球粮食安全是人类面临的主要挑战之一, 解决粮食安全问题, 除了保障耕种面积和维护生态环境, 最有效的措施是开发利用优良的作物品种和先进的栽培技术, 而与之相关的作物品种资源鉴定、遗传育种、栽培生理、植物保护、功能基因组学和植物生物学等方面的研究基本都涉及到对大量植株的各种特征和性状即表型的鉴别与分析, 以及对复杂的植物生长环境的监测与控制。遗憾的是, 传统的植物表型分析规模小(涉及的样本和性状类别少)、效率低(基本上手工操作)、误差大(难以排除人为和环境因素干扰)、适用性弱(难以跨物种参考分析方法和数据), 已成为制约植物功能基因组学和分子设计育种发展的瓶颈。随着科研需求的增长和技术方法的发展, 进行高通量、精准和经济的植物多表型数据测定和环境参数监控的必要性和可行性已经具备, 植物表型组学(plant phenomics)应运而生。
“ 夫欲行其实者, 必先正其名” , 对新概念的形成和发展及概念间的联系和转化的分析理解, 是开展新学科研究的关键基础。植物表型组学是系统研究植物表型信息的一门新兴学科, 其概念和范畴已远远超越了传统意义上的植物表型研究, 为了推进植物表型组学研究的发展, 有必要对相关的概念与研究范畴进行一番仔细梳理分析。
表型组学(phenomics)概念在1997年首次出现在正式文献中, 特指作为基因组研究重要补充的复杂疾病性状研究[1], 但表型组(phenome)一词最早出现于1949年, 作为与基因组对应的概念, 被描述为细胞质或细胞核中基因和自体繁殖部分之外的总和, 是生物表现型的物性反映或物质基础(the material basis of the phenotype)[2]。1960年代中期以来, 在分类学和进化生物学范畴里还出现了表型学(phenetics)概念, 表型组被表述为生物的全部表型(the phenotype as a whole)[3]。1980年代末, 表型组与基因组在进化过程中的对应关系开始受到关注[4], 随后在1990年代中后期出现与表型组学相关的现代表型组概念, 用于表述与基因组对应的和受表观遗传调控的复杂疾病表型[5, 6, 7]。
基因组(genome)概念最早出现于1920年, 1980年代该概念被重新定义并因一篇关于人类基因组计划(human genome project, HGP)的文章[8]引起社会广泛关注。1990年代后, 陆续启动了各种生物的基因组测序工作, 最终奠定了当今生物学研究最重要的科学基础。基因组学(genomics)以基因组研究为范畴, 是与基因组测序和基因组功能分析相伴产生的学科。正是由于现代基因组概念的传播和基因组学的兴起, 20世纪90 年代中后期以来衍生出了一系列以-ome、-omics为词尾的新词汇或赋予了原有词汇新的含义, 催生了表型组学等一些以基因组研究为基础的新学科。因此, 表型组和表型组学概念的形成和发展与基因组和基因组学紧密相关。
在当前的组学研究中, 基因组学的重点是基因组序列分析和基因注释, 其他组学在基因组研究的基础上, 分别研究不同时空条件下的转录组、蛋白组、代谢组和表型组, 其中转录组学的重点是全长cDNA克隆、微阵列基因芯片分析和ESTs分析, 蛋白质组学的重点是分析蛋白质的表达和修饰水平、相互作用和亚细胞定位, 代谢组学的重点是分析代谢物组成和代谢网络, 而表型组学的定义和研究范畴仍在变化和发展中(表1)。
| 表1 表型组学定义汇总 Table 1 Definition list of phenomics |
尽管表型组学概念发端于医学领域, 在作物学领域, 表型组学在育种中的作用已得到普遍重视, 植物表型组研究正在成为植物基因组功能分析和分子育种的重要支撑[22, 23, 24, 25, 26]。
2.1.1 表型(phenotype) 在植物学和遗传学领域, 特别是在作物育种领域, 表型是一个广泛使用的术语。该术语于1911年被定义为基因型(genotype)和所处环境决定的形状、结构、大小、颜色等全部可测的生物体外在表现[27], 随后该概念范畴被扩大到生物化学[28, 29]、分子生物学[30, 31]和行为学[32]等领域, 但在很多科学文献中, 表型还被用于特指鉴别突变体的一种或一系列特征, 如矮化表型(dwarf phenotype)[33]、多枝表型(bushy phenotype)[34]、抗性表型(resistance phenotype)[35]等。由于一般的表型定义在相当大程度上涵盖了当前表型组的概念, 而表型组作为表型组学中的关键词汇已被广泛使用, 为了不引起混淆, 可缩小表型的定义范畴, 特指一个基因型与环境互作的全部或部分表现, 即表型是一个基因型与环境互作产生的全部或部分可辨识特征和性状。
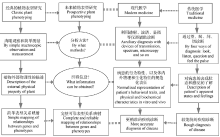
2.1.2 植物表型研究(plant phenotyping) 经典的植物表型研究主要局限于对植物外部物理性的描述, 基本未涉及内部及生化的特征和性状, 而现代医学已经越来越多地采用透射、波谱、显微等仪器检测技术和方法进行更为精准的疾病诊断, 所使用的表型概念也包括了行为特性、以及体内和体表的物理和生化特征(图1)。因此, 一般认为, 随着技术的发展, 植物表型的研究范围应不仅限于外观物理状态[36, 37], 确切地讲, 为了完整可靠地分析基因和表型的关系, 未来的植物表型研究应充分借助各种先进的仪器分析方法。
2.1.3 植物表型(plant phenotype) 过去仅依赖肉眼观察和简单测量获得的植物特征和性状十分有限, 也缺乏规范化的表征, 无法满足作物基因组功能研究和作物育种的实际需求。事实上, 当前已经有很多分析监测技术可用于植物表型研究, 能够精确获得涵盖从分子到群体的各种植物特征和性状。如全基因组关联分析(genome-wide association study, GWAS)获得的单核苷酸多态性(SNP), 即可视为在基因水平上的表型特征。考虑到植物表型研究的现状, 同时参考表型概念的定义及本文对植物表型的层次和范畴的划分(表2), 可将植物表型定义为能够反映植物结构和组成的, 或能反映植物生长发育过程和结果的, 由一个基因型与一类环境互作产生的部分或全部可辨识的植物物理、生理和生化特征及性状。利用这些特征和性状, 应该能够将不同植物基因型和环境的决定作用或影响区分开来。
| 表2 植物表型的层次和范畴 Table 2 Levels and categories of plant phenotype |
2.2.1 植物表型组(plant phenome) 植物表型组可以简单定义为全部的植物表型, 但概念上极容易出现混淆, 因为这一词汇既可用来表示一个基因型与环境互作产生的全部物理、生理和生化特征和性状, 也可表示一种植物的不同基因型与环境互作产生的全部物理、生理和生化特征和性状, 而且某些特征和性状能否被识别和精准检测还取决于所用的技术手段。因此, 更准确的植物表型组概念应定义为受基因组(可包括多种基因型)和环境因素决定或影响, 并能反映植物结构及组成、植物生长发育过程及结果的全部物理、生理、生化特征和性状, 包括可辨识的且能与基因、环境映射或未能映射的性状, 也包括某些目前尚不可辨识的性状和未表现的性状(表3), 植物的这些特征和特性应该能够将植物个体或群体所受的任何基因型的决定作用或环境因素影响区分开。从信息学的角度看, 表型组也可以定义为生物个体或遗传同质群体响应体内外信号所表现出的能以动态数据流形式呈现的全部特征和性状[38]。
2.2.2 准表型组(quasi-phenome) 回顾基因组研究的历史, 由于发现仅3%~5%的核酸序列可编码蛋白质, 似乎测定非编码序列没有必要, 因此早期基因组研究中曾发生过应进行全基因组测序还是靶向功能区域测序的争论。当前的植物表型组研究也面临类似的情况, 是花功夫进行全表型组分析还是集中力量分析有价值的表型, 似乎很难做出决断[17]。一方面, 尽管表型组的概念是从基因组衍生出来的, 由于植物生长发育的时空差异性和环境变化, 表型组学研究理论上不可能获得类似基因组数据的完整信息, 即表型组信息几乎无法穷尽, 不可能像全基因组测序一样在一定期限内达到目标; 另一方面, 有限的表型数据对基因组功能分析和育种栽培研究的意义不大, 只有得到尽可能多的表型信息, 才可能实现完整可靠的表型组与基因组和环境关系的映射。因此有必要提出一个准表型组概念, 定义为现有技术方法可以检测分析的全部表型, 以区分目前尚不可辨识的性状和未表现的性状(表3)。随着植物表型组研究策略的完善、研究方案顶层设计的完成、仪器设备和技术方法的发展, 高通量、精准和低成本的准表型组分析应该能够实现。
| 表3 植物表型组相关概念定义表 Table 3 Definition of related concepts of plant phenome |
2.2.3 植物表型组构成(composition of plant phenome)
本文定义的植物表型组相关概念见表3, 其构成关系见图2。其中, 可辨识性状/准表型组(identifiable traits/quasi-phenome)为一般意义上的表型组, 是植物表型组数据测定和分析的主体; 映射性状(mapped traits)具有能够清楚反映特定的基因和环境因素对生物本身复杂影响的信息, 即已实现与基因及环境关系的映射, 属于严格意义上的可测表型范畴; 目标性状(target traits)属于在基因功能、栽培技术或作物育种研究中需重点关注的性状, 是常规表型组分析的重点; 非目标性状(non-target traits)和无映射性状(unmapped traits)属于待深入研究的范畴, 是未来表型组学研究的重点; 不可辨识性状(unidentifiable traits)属于广义表型的组成部分, 其研究具有前沿性和探索性; 未鉴定性状(unidentified traits)包括有望与基因或环境映射的可量化的指标和参数, 涉及多学科交叉, 属于植物表型组学研究重点之一; 未知性状(undetectable traits)的分析可能成为某些植物基因功能研究中的关键环节, 其发现有赖于科学的预测推导和相关检测技术方法的发展; 隐性性状(recessive traits)指因极端环境或基因的干涉、诱变、互作、沉默等导致植物丧失或新出现的性状, 多数情况下属于未鉴定或未知性状, 也包括对植物基因功能研究有重要意义的新出现或已丧失的可辨识性状。
 | 图2 植物表型组的构成如表3和本图所示, 严格意义上的植物表型组由不同含义的特征和特性构成, 这些特征和特性应该能够将植物个体或群体所受任何基因型的决定作用或环境因素影响区分开。Fig. 2 The compositions of plant phenomeAs shown in Table 3 and this Figure, the strict sense of the plant phenome may be composed of some types of characteristics and traits that have different meanings, and these characteristics and traits could be used to distinguish the gentype decision or environment influence on a plant individual or a group from any other ones. |
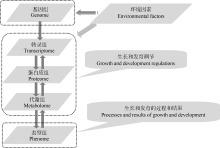
3.1.1 环境参数(environmental parameters) 基因组通过对转录组、蛋白质组和代谢组的控制来决定表型组, 但环境对植物表型与基因关系也有多方面的影响。环境因素可能诱导植物基因组的某些基因突变导致表型改变, 更普遍存在的情况是通过对转录组、蛋白质组和代谢组的干涉来影响植物生长发育的过程和结果, 即通过调节生长发育来影响表型组(图3)。植物的种植期和植株密度, 生长过程中的生物胁迫及光、温、水、肥、气、土等环境因素均对表型有影响, 一般情况下具有同一个基因组的植物在不同的环境中会产生不同的表型, 极端情况下甚至会出现某些完全相反的表型性状, 因此收集和利用表型组数据时必须同步考虑与植物生长过程相对应的环境参数, 即环境参数的全程采集和分析应该是植物表型组研究的组成部分。
3.1.2 环境监控(environmental detecting and control)
表型是基因功能的表现形式, 同时表型的形成也会受到诸多环境因素的影响, 因此表型组学研究实质上以特定的基因组和环境因素为始发点。特别在植物学领域, 特定植物的基因组以个体或同质群体为客观稳定的载体, 基因组学研究和表型组学研究可以独立进行并随后进行映射分析, 而植物生长的环境因素具有非恒定性和易消失特性, 研究中必须提前或同步控制和采集环境参数, 以保证植物表型组数据的可比性[39], 同时, 只有在严格可控条件下的精确表型分析才可能快速(即无需多次重复实验)确定基因型和环境与表型的确切关系。目前自动化表型分析及环境监测控制技术均有长足发展, 整合表型和环境数据的采集在技术上并无大的障碍, 将系统的环境监控列入植物表型组学范畴是合理的, 也是可行的。
3.1.3 植物表型的遗传和环境包容性(tolerance of plant phenotype to the changes of inheritance and environment, CIE) 各种表型性状间可能存在复杂的协同或制约关系, 它们共同决定或影响终极目标性状的产生, 因此植物表型组学研究应该有助于表型性状组合的选择。为了明晰植物表型组学研究与育种的深层次关系, 可引入植物表型的遗传包容性(tolerance to the change of inheritance, TCI)和环境包容性(tolerance to the change of environment, TCE)概念, 同时限定在一定的范围内改变遗传构成和环境因素后, 植物表型的TCI和TCE与主要目标性状的可接受性成正比。特定植物表型的遗传和环境包容性(TCIE)可反映该表型受相关基因和生长条件影响的程度, 育种的目的即是获得表型的遗传包容性和环境包容性高的品种, 也即获得具有多种优良农艺性状和适应性广的品种。这种表型的遗传和环境包容性研究涉及海量数据的采集和分析, 应该成为植物表型组学的一个重要方面。
3.2.1 基本目标(basic goal of plant phenomics)
植物表型组学是直接服务于植物功能基因组学和作物分子育种的新学科, 其研究工作兼具基础性和实用性。当前, 大量的基因分子标记已用于指示与有价值性状相关的染色体区域, 这些DNA或RNA等标记均可视为基因水平的表型特征(表2)。现代育种工作中, 常结合使用外观表型选择和分子标记辅助选择(marker-assisted selection, MAS), 前者能确切反映基因和环境的某些实际作用, 但时效性较差, 后者的优势是可以预测最终表型, 但仍有一定的不确定性。使用全基因组选择(genomic selection, GS), 即在基因组水平上分析计算等位基因效应和全部分子标记, 利用所获得的育种值(derive genomic estimated breeding values, GEBV)指导和辅助育种选择, 也存在不确定性问题。通过植物表型组学研究, 有望获得从基因水平到终极特性水平的各种植物表型信息, 其中生理生化水平上的表型信息应能更好地满足对时效性和确定性的要求。
3.2.2 植物表型组学(plant phenomics) 一项完整的植物表型组学研究, 首先涉及对植物表型组信息及相关环境参数产生条件的综合控制(comprehensive controls), 即为获取有效的表型信息和环境参数, 首先需要精确设计和控制一组实验, 包括基因的操作和组合、植物的繁种和栽培、环境的设置和保障等; 其次涉及对植物表型组及相关环境数据的完整采集(complete collections), 即需要使用一整套技术手段采集各种不同类型的表型及环境信息, 包括不同表型层次的准表型组数据, 光温水气肥等环境数据; 最后, 最重要的环节是对海量的表型及环境数据的系统分析(system analyses), 即需要分析处理海量数据以获得有关表型特征和性状之间的关联(correlation)、基因和环境与表型之间的映射等知识, 包括预先进行海量数据的汇集、储存、整理和呈现, 以及整合其他组学资料特别是基因组资料等。因此, 本文将植物表型组学简洁地定义为对植物表型组信息及相关环境参数的综合控制、完整采集和系统分析(plant phenomics could be concisely defined as the comprehensive controls, complete collections and system analyses of plant phenome informations and related environmental parameters)。
植物表型组学是获取和利用植物表型组信息的综合学科, 从研究目标、手段和层次等方面考虑, 至少可以划分为以下3个方向, 其中构建植物表型组学本体具有十分重要的意义。
3.3.1 检测表型组学(detected phenomics) 包括为实现表型组和环境数据检测开展的相关研究, 以及检测植物的各种基因型或突变型在不同环境条件下的表型组, 即该植物的全部(或部分)发育、生理、生化、形态、结构、品质等特征和性状, 以及对应的环境参数, 可为植物功能基因组分析和环境影响研究提供完整的基础数据。事实上, 目前国际上的植物表型组学研究主要集中在检测大量植物样本中的有限的目标性状(target traits)方面, 而并非进行全表型组性状分析[24, 25, 26]。
3.3.2 定向表型组学(targeted phenomics) 有针对性地分析特定的基因或基因族群在设定的环境中与植物表型特征和特性的关系, 最终比较可靠地实现表型(组)和基因(组)及环境数据之间隐含关系的映射(mapping), 同时分析不同表型层次和范畴之间的关系, 最大限度地实现特定的表型特征和性状之间的关联(correlation)。在整体上考虑不同的表型层次和类别的形成关系, 以及基因组和环境的决定作用或影响程度, 是有效进行植物功能基因组研究和分子育种的关键所在, 也是近年植物表型组学研究的核心[40, 41, 42]。
3.3.3 本体表型组学(ontology-based phenomics)
目前学术界已经使用表型本体(phenotype ontologies, PO)这一术语来表示对种内或种间的基因型和表型关系的明确的、概念化的、规范的说明[43, 44, 45], 但从划分研究层面来考虑, 使用本体表型组学能更恰当地反映构建和应用PO与检测表型组学和定向表型组学的区别。Ontology (本体论)本是一个哲学概念, 一般指通过认识论而得到认识的一切实在的最终本性, 即研究存在的最终本性。随着信息技术的发展, ontology被赋予了一系列新含义, 总结起来, 可以将该术语定义为相关概念及其关系的规范化表述。在生物学领域, 可以将其具体定义为利用计算机技术收集和整理公开发表的生物学数据及知识, 构建和呈现这些可用信息之间相互关系的概念化模型(本体), 以揭示这些数据和知识所能反映出的生物体或生命现象的本质特征。植物本体表型组学研究的最终目是构建和应用具有查询和预测功能的、能反映种内或种间基因组、环境因素和表型组之间系统关系的植物表型组学本体(plant phenomics ontologies, PPOs)。
尽管植物表型组学研究已有长足的发展, 理论基础缺乏和研究的系统性、有效性、共享性差仍是客观现实。笔者认为, 欲实现系统的、有效的、全球信息共享的植物表型组学研究, 需要从如下三方面开展顶层设计:
(1) 确定特定物种的表型特征特性及环境参数的术语类别、检测方案和技术标准, 即构建植物表型的统一术语库和检测标准, 前者属本体表型组学范畴, 后者属检测表型组学范畴。
(2) 确定表型组学数据采集、储存、整理及呈现的技术方法和运行模式, 即综合工程、仪表、计算机、信息学和生物学技术, 构建具有高效率、高效力和经济的数据采集和处理系统, 属检测表型组学和本体表型组学范畴。
(3) 确定各表型信息之间、表型组与基因组及环境之间关系的表示形式和验证程序, 即构建植物表型组学本体和验证模式, 属于本体表型组学和定向表型组学范畴。
规范化的、精准的表型分析在基础研究和分子育种等工作中具有不可替代的作用, 是高效地进行植物功能分析、作物资源筛选、优良品种培育、品种比较和栽培技术开发的重要保证。近十余年来为生命科学和药物筛选开发的分析技术, 如用于分子和细胞分析的质谱(mass spectrometry, MS)、近红外光谱(near-infrared spectrometry, NIR)、核磁共振(nuclear magnetic resonance spectrometry, NMR)、免疫组化(immunohistochemistry, IHC)、免疫荧光(immunofluorescence, IF)、流式细胞(flowcytometry, FC)、分子互作分析、SNPs等技术, 已经逐渐应用于植物基因型与表型映射分析中。同时在植物叶面积、株型、生物量和生理特性等表型组数据的采集中, 更多使用的是一些专门为植物研究开发的自动化图像分析技术, 如二维或三维的三光彩色成像(RGB imaging)[46]、荧光成像(fluorescence imaging)[47]、热成像(thermal imaging)[48]、成像光谱(imaging spectroscopy)[49]等, 这类基于图像转换等原理的自动化表型分析技术具有非侵入式和高通量的特点, 已被Nature Methods杂志列为2013年最值得关注的科学技术之一[50], 其中融合高光谱数据和高分辨率图像的成像光谱技术可能在未来植物表型组学研究中发挥重要作用。
目前一些不同规模和类型的植物表型分析平台已在很多国家安装和使用。依据研究对象、目的和环境的不同, 可以整合各种自动化的植物表型分析技术用于培养箱、温室和大田的单株或群体测定, 对部分精细性状则可采用实验室离体测定。按对植物样本的影响方式, 表型检测方法可划分为无创检测方法(noninvasive methods), 如不损伤植株或器官的光谱分析、图像分析、粒重分析等; 微创检测方法(minimally invasive methods), 如需要痕量取样或轻微损伤植物的SNPs分析、转录特征分析、呼吸特征分析等; 有创检测方法(invasive methods), 如需要较大量取样或较严重损伤植物的蛋白质特征分析、代谢物特征分析、解剖分析等; 以及毁灭性检测方法(destructive methods), 如需要破坏整个植株或器官才能完成的某些表型分析。但总体来讲, 现有的技术和方法无法充分满足高通量和高质量分析植物样本的需求, 开发和使用具有高效率、高效力和经济的性能(efficiency, effectiveness and economy), 同时具备物联网(internet of things)技术特性的植物表型分析系统, 是未来植物表型组学的重要方向之一。换言之, 有必要基于对植物表型组和表型组学概念与范畴的分析, 进一步研发和整合各种实时和自动的样本识别、数据采集、条件控制、信息传输等表型分析与环境监控技术, 构建自动化、高通量和精准的模块化植物表型研究技术平台。
国外较大规模开展植物表型组分析的机构主要是一些国家级的研究单位和大型种业公司的科研部门, 较活跃的学术组织有IPPN、EPPN和UKPPN, 多数大规模开展植物表型组分析的机构均加入了这些组织(表4), 一些专门从事植物表型组分析技术和设备研发的科研机构和公司也十分活跃。
| 表4 开展植物表型组分析的主要机构和组织 Table 4 Main institutions and organizations in the field of plant phenome analysis |
目前国际上知名度较高的植物表型组学学术会议和活动主要有International Plant Phenotyping Symposium (IPPS, http://www.plant-phenotyping.org/); PhenoDays和PhenoDays USA (http://www.pheno-days.com/), International Workshop on Image Analysis Methods for the Plant Sciences和Image Analysis for Biologists (http://www.cpib.ac.uk/outreach/); Plant Phenotyping Workshop (http://www.plant-phenotyping- network.eu/), UK Plant Phenomics Network Meeting, Photo-Physiology Phenotyping Workshop和Image Analysis Methods in the Plant Sciences (http://www. ukppn.org.uk/); European Plant Phenotyping Network Workshop (http://www.epsoweb.org/)。
迄今为止, 植物表型组学研究论文主要发表在一些传统的植物学、遗传学和综合性的期刊上, 未见本学科的专业期刊创刊, 但近几年已有几本相关的重要专著、专辑和学位论文出版, 即High-Throughput Phenotyping in Plants: Methods and Protocols (Normanly主编, 2012), High Throughput Automated Seedling Phenotyping System (Subramanian, 博士学位论文, 2012), Phenomics: A New Tool in the Prediction of Host-specificity in Classical Biological Control of Weeds? (Rapo, 博士学位论文, 2012), Phenotyping for Plant Breeding: Applications of Phenotyping Methods for Crop Improvement (Panguluri和Kumar主编, 2013), OMICS Applications in Crop Science (Barh主编, 2013), The Handbook of Plant Metabolomics (Weckwerth和Kahl主编, 2013), Phenotyping for Plant Breeding-Applications of Phenotyping Methods for Crop Improvement (Saeed和Panguluri主编, 2013)和Phenomics (Hancock主编, 2014)。
植物表型组学是一门正迅速发展的新兴交叉学科, 对相关概念与研究范畴的分析讨论有助于学科的建设和发展。通过对表型组学的起源及其各种定义的了解, 以及对植物表型组和表型组学相关概念间的关系、概念的含义和范畴等方面的分析讨论, 能够初步勾勒出支撑植物表型组学研究的理论框架。首先, 植物表型组应该包括从分子到群体的全部物理、生理、生化特征和性状。其次, 在植物表型组研究中可引入准表型组、可辨识性状、映射性状、目标性状等概念。第三, 精确的环境监控应成为植物表型组学研究的一部分, 可引入植物表型的遗传和环境包容性概念。第四, 植物表型组学可定义为对植物表型组信息及相关环境参数的综合控制、完整采集和系统分析, 进一步可区分为检测表型组学、定向表型组学和本体表型组学, 其基本目标是获得从基因水平到终极特性水平的各种植物表型信息。最后, 尚需仔细考虑植物表型组分析技术和植物表型组学研究的顶层设计原则, 包括相关的术语类别、检测方案和技术标准, 获取和使用相关数据的技术方法和模式, 以及各种信息之间关系的表示形式和验证程序等。
当前植物表型组学领域的热点之一是建设分析技术平台, 但简单地依赖商品化的设备难以完成系统复杂的植物表型组研究。作为一门新兴的交叉学科, 植物表型组学尚有较大的发展空间, 有必要在把握植物表型组和植物表型组学的概念和研究范畴的基础上, 整合植物学、信息学、仪表学、光电子学、自动化学、机械学、环境学、工程学等多领域的研究和技术力量并加大创新资金投入, 建立满足实验室、温室和大田中不同作物表型性状检测和环境监控的模块式集成分析系统。此外, 还有必要注重定向表型组学和本体表型组学的理论研究与实践, 加强植物表型组学人才的培养和引进工作。
表型组信息客观存在于植物中, 有待被系统地发现、定义、分析和应用。深入的植物表型组学研究, 必将极大地促进植物功能基因组学和作物分子育种与高效栽培的进程。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|
| [49] |
|
| [50] |
|