第一作者联系方式: E-mail: malei220@mail.tricaas.com, Tel: 0571-86650417
高等植物叶绿素的生物合成对其正常光合作用起关键作用。本文根据前期芯片杂交结果, 采用RT-PCR和RACE技术克隆了3个茶树叶绿素合成相关基因, 分别为谷氨酸-tRNA还原酶基因( CsGluTR)、叶绿素合酶基因( CsChlS)、叶绿素酸醋氧化酶基因( CsCAO), 对应的GenBank的登录号分别为HQ660371、HQ660370、HQ660369。序列分析表明, CsGluTR基因全长2165 bp, 开放阅读框长1665 bp, 编码554个氨基酸, 推测的蛋白分子量约为60.6 kD, 理论等电点为8.78; CsChlS基因全长1463 bp, 其中开放阅读框长1125 bp, 编码374个氨基酸, 推测的蛋白分子量约为40.5 kD, 理论等电点为8.58; CsCAO基因全长2146 bp, 其中开放阅读框长1611 bp, 编码536个氨基酸, 推测的蛋白分子量约为60.8 kD, 理论等电点为8.03。比对分析表明, 3个基因编码的氨基酸序列与其他植物中同源基因的相似性均在70%以上。利用荧光定量PCR技术检测3个基因在不同白化阶段的表达, 表明 CsChlS和 CsCAO基因具有明显的表达协同性, 它们在叶片中的表达量与叶片的颜色变化高度同步; 而 CsGluTR在白化叶片和正常叶片中的表达差异相对较小, 同时在新生芽叶转绿过程中最先恢复正常表达水平。说明在白化叶片中, 叶绿素的合成机制受到较大影响, 叶绿素合成受阻导致的叶片内色素类物质含量降低或消失是叶片白化的直接原因。
Chlorophyll is one of the main pigments participating in photosynthesis in plant chloroplasts, and its biosynthesis is crucial for higher plant. In this article, we cloned and characterized three important genes involved in the biosynthesis of chlorophyll which were CsGluTR, CsChlS, and CsCAO(GenBank accession number HQ660371, HQ660370, and HQ660369) lead on the results of cDNA microarray hybridization. The full-length cDNA of CsGluTR was 2165 bp, containing a 1665 bp ORF encoding a 554 amino acids protein, and its 3′ untranslated region had an obvious polyadenylation signal. The deduced protein molecular weight was 60.6 kD and its theoretical isoelectric point was 8.78. The obtained cDNA of CsChlS was 1463 bp in length, containing a 1125 bp ORF which encoded 374 amino acid residues. The deduced protein molecular weight was 40.5 kD and its theoretical isoelectric point was 8.58. The full-length of CsCAO was 2146 bp, containing a 1611 bp ORF encoding a 536 amino acids protein. The deduced amino acid sequence of CsGluTR, CsChlS, and CsCAO from tea plant shared high identity with those of other species, for instance the similarity of 79%, 90%, and 77 % with Vitis vinifera, respectively. The result of Real-time RT-PCR analysis showed a coordinated expression of CsChlSand CsCAO, which was corresponded with the change of the albino phenotype. However, there were small changes in the expression level of CsGluTR between the normal and albino leaves. These results implied that the biosynthesis of chlorophyll is completely hindered in albino leaves, causing the decline of pigment content and the albino phenotype.
叶绿素是光合作用的主要色素之一, 也是大多数植物叶片呈现绿色的主要原因。高等植物叶绿体中, 从L-谷氨酞-tRNA开始, 经多步酶促反应最后形成叶绿素a和叶绿素b, 涉及15种酶、20多个基因[1, 2]。一般可将其整个生物合成过程大致分为4个阶段, 由谷氨酸生成δ -氨基酮戊酸、叶绿素合成开始、由δ -氨基酮戊酸合成原卟啉IX、在镁离子螯合酶的作用下原卟啉IX与Mg2+结合生成镁原卟啉IX, 再经过酶促反应合成叶绿素酸酯, 进而生成叶绿素a, 同时进行叶绿素a和叶绿素b的相互转化。在这一复杂而保守的过程中, 任何一个基因的突变都可能严重影响叶绿素合成的效率, 从而表现出不同的叶色变异[3]。叶色变异是高等植物中突变频率较高且易于鉴定的突变性状, 迄今为止, 在水稻、小麦、大麦、玉米、大豆、棉花、烟草、玉米、向日葵、番茄、黄瓜、马铃薯、桑树等几乎所有高等植物中都发现了叶色突变体[4, 5, 6, 7, 8]。常见的叶色突变类型主要表现为黄化、浅绿、深绿、白化、白绿条纹和黄绿条纹等[9]。
通常情况下, 叶绿素含量下降会对作物品质产生负面影响, 有些甚至会导致植株死亡, 但在茶树中, 由于以白化或黄化突变体为原料制作的茶样具有更高的氨基酸含量和更优异的外形, 从而使其经济价值更高, 如白叶1号(安吉白茶)、安吉黄茶、景宁白茶、中黄1号(天台黄)等白化或黄化品种已成为当地茶农增收的重要途径。特别是白叶1号, 在安吉县的种植面积已达6700公顷、产量1300吨、产值14.5亿元[10]。近十多年, 科研人员已从生理学、生物化学、细胞学等多方面对其叶片白化的机制进行了研究, 并取得了很大的进展, 但仍未解开引起突变表型的核心分子机制[11, 12, 13]。本课题组在前期进行白叶1号不同白化阶段的芯片杂交实验时, 筛选到了一批可能与其白化表型相关的差异表达基因, 其中包括多个叶绿素合成途径的催化酶[14]。本研究在此基础上, 采用RT-PCR和RACE技术克隆了其中3个基因的全长序列, 并对其表达情况进行了分析, 该研究结果将为揭示白叶1号的白化机制提供参考。
以种植于国家种质杭州茶树圃的白叶1号为试验材料, 采集其春季不同白化阶段的一芽二叶, 以及一芽五叶的不同部位叶片(一芽一叶、第二叶、第三叶、第四叶和第五叶), 液氮速冻后, 于-80℃冰箱保存备用。随后采用植物总RNA快速提取试剂盒, 按说明书提取白叶1号所有样本的总RNA。用微量紫外检测仪NanoDrop 1000 (Pittsburgh, PA, USA)测定RNA样品浓度, 1.2%变性琼脂糖凝胶电泳检测RNA质量。
利用NCBI上的BlastX软件分别对芯片筛选出的茶树谷氨酸-tRNA还原酶(glutamyl-tRNA reductase, CsGluTR)、叶绿素合酶(chlorophyll synthase, CsChlS)和叶绿素酸醋氧化酶(chlorophyllide a oxygenase, CsCAO) 3个基因片段进行序列同源比对。同其他植物相比, 本实验室通过前期转录组测序获得的3个基因序列均是基因的中间片段, 缺少基因的3′ 端和5′ 端[15]。因此根据基因的已知片段, 设计12条嵌套的基因特异引物(表1), 并参照Clontech公司的SMART RACE cDNA Synthesis Kit试剂盒说明书, 将提取的白叶1号一芽二叶新梢总RNA反转录为cDNA, 以稀释10倍的cDNA为模板进行PCR, 反应体系中10× PCR buffer (Mg2+ plus) 2 μ L, 10 mmol L-1 dNTPs各0.4 μ L, 模板cDNA 0.4 μ L, PCR特异引物和通用引物(10 pmol μ L-1)各0.2 μ L, TaqDNA聚合酶(5 U μ L-1) 0.2 μ L, 加水至终体积20 μ L, 反应在ABI Veriti Thermal Cycler型PCR仪(Applied Biosystems, California, USA)上进行, 反应条件为94℃预变性3 min; 94℃变性45 s, 54℃复性45 s, 72℃延伸45 s, 35个循环, 最后72℃再延伸5 min。PCR产物经蒸馏水稀释10倍, 取0.4 μ L作为模板, 以每个基因的第2轮引物和通用引物再次进行PCR扩增。反应条件除退火温度升至60℃以外, 其他均相同。扩增产物经1.2%琼脂糖凝胶电泳检测, 用多功能DNA纯化回收试剂盒(Axygen Biosciences, California, USA)回收扩增产物, 连接到pTA2载体, 再转化到大肠杆菌DH5α , 蓝白斑筛选, 挑取白色单菌落鉴定, 根据菌落PCR结果, 选择3个阳性克隆进行双向测序。参照马春雷等[16]的方法对测序结果进行生物信息学分析。
| 表1 基因克隆及表达分析所用引物序列 Table 1 Primers used in gene cloning and expression analysis |
根据白叶1号春季不同时期叶色的变化特征, 分别采集不同白化阶段的一芽二叶和第5叶完全白化时的一芽五叶枝条, 将其分部位提取总RNA, 并反转录成cDNA, 稀释5倍作为荧光定量模板。采用相对定量的方法, 以18S rRNA为内标基因(表1), 荧光定量反应体系为SYBR Premix Ex Taq 25 μ L, 10 μ mol L-1上下游引物各2 μ L, ROX Dye II 1 μ L, cDNA 4 μ L, 加水至终体积50 μ L。反应在ABI PRISM 7500实时定量PCR仪(Applied Biosystems, Foster City, CA)上进行, 程序为95℃变性30 s, 94℃变性15 s, 60℃退火延伸34 s, 40个循环, 于60℃收集荧光, 反应结束后分析荧光值变化曲线和熔解曲线, 每个反应重复3次, 采用2-Δ Δ Ct算法分析结果[17]。
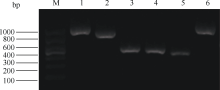
根据已知的CsGluTR基因部分序列设计5′ 端和3′ 端嵌套引物, 经过2轮PCR扩增后, 5′ 端得到一条约900 bp的片段, 3′ 端得到一条约850 bp的片段(图1)。测序结果表明, 5′ 端测序的3个克隆为一条长883 bp序列; 而3′ 端测序的3个克隆则是相似性极高的不同序列, Blast比对分析表明它们均与其他植物的GluTR基因有较高的同源性, 其中最长的为812 bp, 最短的为802 bp。最后将测序得到5′ 端序列、原有的中间片段和最长的3′ 端序列拼接, 得到一条全长2165 bp的cDNA序列, 开放阅读框长1665 bp, 编码554个氨基酸, 推测的蛋白分子量约为60.6 kD, 理论等电点为8.78, 该基因在GenBank的登录号为HQ660371。同时将另外2个3′ 端序列用同样方法与原序列拼接, 并根据其开放阅读框翻译成氨基酸后, 发现他们编码的蛋白质大小一致, 但有7个位点的氨基酸发生了置换, 说明在茶树中, 谷氨酸-tRNA还原酶可能是以基因家族或者多基因家族的形式存在, 其功能是否一致还有待进一步验证。
通过NCBI-CDS在线分析CsGluTR基因的保守结构域[18], 发现其编码的氨基酸序列中存在NADP binding site (GxGxxG)和tRNA binding site (Sx4ExE/ Qx3Q) 2个保守结构域, 它们同属PLN00203多结构域, 是谷氨酸-tRNA还原酶的特征结构。进一步将CsGluTR的全长cDNA进行BlastX同源比对, 结果显示, 它与其他物种中报道的GluTR的相似性均在60%以上, 其中与葡萄(Vitis vinifera)和克莱门柚(Citrus clementina)的相似性最高, 达79%。
按照上述方法分别设计CsChlS基因的5′ 端和3′ 端嵌套引物, 经2轮PCR扩增, 电泳检测均得到一条约400 bp的清晰条带(图1), 与预期目的片段大小相符, 测序结果分别为395 bp和325 bp, 通过DNAstar的SeqMan程序将其与已知序列拼接, 最终得到一条全长1463 bp的cDNA序列, 开放阅读框长1125 bp, 编码374个氨基酸, 推测的蛋白分子量约为40.5 kD, 理论等电点为8.58, 该基因在GenBank的登录号为HQ660370。
序列分析表明, CsChlS编码蛋白具有典型的叶绿素合酶的活性位点NDDDND[aS]D, 即2个富含天冬氨酸(D、143、146、150、272、279)的结构域[19]。将CsChlS的全长cDNA进行BlastX同源比对, 结果显示, 与其相似性较高的序列均为其他物种中的叶绿素合成酶蛋白, 其与葡萄、克莱门柚、黄瓜(Cucumis sativus)、烟草(Nicotiana tabacum)、野草莓(Fragaria vescasubsp. vesca)等的相似性分别为90%、88%、87%、87%和86%。
茶树叶绿素酸醋氧化酶基因按照同样方法设计引物, 经2轮PCR扩增, 5′ RACE和3′ RACE电泳结果显示, 分别在800 bp和400 bp处有2条较清晰的电泳带, 测序结果为774 bp和388 bp, 将其与原有中间片段拼接, 最终得到一条全长2146 bp的cDNA序列, 开放阅读框长1611 bp, 编码536个氨基酸, 推测的蛋白分子量约为60.8 kD, 理论等电点为8.03, 该基因在GenBank的登录号为HQ660369。序列分析表明, CsCAO编码的氨基酸包含氧化酶所必有的2个保守的结合位点, 即Rieske-type (2Fe-2S)蛋白结合位点和单核心铁结合位点[20]。将CsCAO的全长cDNA进行BlastX同源比对, 结果显示, 其与橙(Citrus sinensis)、葡萄、桑树(Morus notabilis)、可可(Theobroma cacao)的CAO蛋白相似性分别为78%、77%、76%和76%。
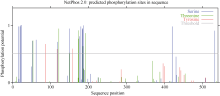
蛋白质磷酸化是蛋白质翻译后最常见和最重要的一种修饰方式, 分析茶树CsCAO编码氨基酸的潜在磷酸化位点, 有助于了解茶树CsCAO蛋白在翻译后水平上的调控方式。本文采用在线软件NetPhoS 2.0对该基因编码蛋白的潜在磷酸化位点进行预测显示, 共存在25个潜在的磷酸化位点(图5), 且主要分布于多肽链的前半部分, 其中丝氨酸、苏氨酸和酪氨酸的磷酸化位点数分别是15、7和3, 说明对于CsCAO蛋白, 磷酸化与否对其发挥催化功能至关重要。
对来自葡萄(Vitis vinifera)、克莱门柚(Citrus clementina)、黄瓜(Cucumis sativus)等16个不同物种的谷氨酸-tRNA还原酶、叶绿素合酶和叶绿素酸醋氧化酶蛋白进行多重比对, 并采用Mega5.0邻近相接法(Neighbor-Joining, NJ)构建系统进化树。分析结果表明(图7), 来自不同物种的共48条序列首先按照功能可清晰地分为3类, 且不同物种的叶绿素合酶和叶绿素酸醋氧化酶在进化上的关系更近; 但在3个不同类群下, 则无明显的物种聚类规律, 如在叶绿素合酶的大分支下, 茶树最先与玄参科猴面花(Mimulus guttatus)和马铃薯(Solanum tuberosum)聚为一类; 在叶绿素酸醋氧化酶的大分支下, 茶树则与葡萄(Vitis vinifera)和马铃薯(Solanum tuberosum)聚为一类; 而在谷氨酸-tRNA还原酶的大分支下, 茶树与玄参科猴面花(Mimulus guttatus)和大豆(Glycine max)等聚为一类。由此说明, 基于单基因的系统进化分析可作为基因功能判定的有力证据, 但并不适合用来研究不同物种之间的进化关系。
本实验室经多年田间观察发现, 白叶1号春季的表型变化可分为如下几个阶段: 春季新发嫩芽的芽头为浅绿色, 展开叶先呈黄绿色, 随着芽叶生长, 叶片逐渐变大, 叶脉呈绿色, 叶面转白, 至一芽三叶期, 叶面全部为白色。随后新叶转绿, 一芽四叶期顶叶为绿色, 其他叶片为白色; 一芽五叶期的一芽一叶转绿, 表现为白绿相间, 其余三叶仍为白色。随后新生叶片恢复至正常绿色叶片, 早期白化叶片则逐渐干枯掉落。从表型看, 并无白化叶返绿过程, 复绿表型主要是由于特定时间的新生叶片转为绿色, 而原一芽三叶期的成熟白叶则慢慢老化脱落, 个别芽头一芽三叶期时的一叶, 即主脉仍为绿色的新叶随后有返绿特征, 而最初两叶很难复绿。根据这一表型变化过程, 我们从白叶1号的一芽二叶期开始, 每周采集其新稍一芽二叶, 至新稍完全复绿; 同时采集其新稍生长至一芽五叶期后的各部位叶片。随后利用荧光定量PCR技术对本试验克隆的3个叶绿素合成途径中的关键催化酶的表达规律分析表明(图8), 在白叶1号到达一芽二叶期后的约2周内, 其叶绿素合成途径已被破坏, 3个叶绿素合成主要结构酶的表达量相继到达最低水平, 其中CsGluTR在第7天和第14天的相对表达量均为0.57, 与对照相比表达量降低了近一半; CsChlS和CsCAO在第7天的相对表达量分别为0.57和0.47, 在第14天的相对表达量分别为0.68和0.63; 对应的表型观测结果表明, 这一时期也是白叶1号新梢处于完全白化的阶段。随后CsGluTR基因的相对表达量在第3周恢复到1.73, 并随着新稍发育逐渐增强; 同时2个基因CsChlS和CsCAO的相对表达量也在3周后显著提高, 分别为0.89和0.76, 这一时期属于白叶1号的一芽四叶期, 也是新生芽叶转绿的阶段; 当新稍到达一芽五叶期后, 新生芽叶中的3个基因的表达都恢复至较高水平, 其相对表达量分别为18.70、16.19和16.94; 表达量最高的CsGluTR基因是其最初一芽二叶的近20倍。
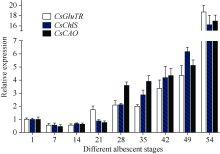 | 图8 茶树CsGluTR、CsChlS和CsCAO基因在白叶1号不同白化阶段的表达Fig. 8 Expression of CsGluTR, CsChlS, and CsCAO genes in the different albescent stages of tea plant variety Baiye 1 |
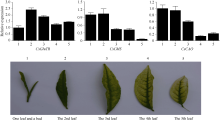
另外, 对3个基因在不同部位叶片的表达检测结果也表明, 3个基因的表达与其表型变化密切相关(图9), 特别是与叶绿素合成直接相关的2个基因CsChlS和CsCAO, 其表达量在第四和第五叶中达到最低水平, 在第四叶中分别为0.42和0.14; 在完全白化的第5叶中分别为0.06和0.22, 其中CsChlS在第五叶中的相对表达量与一芽一叶中的相差约17倍。说明在白化叶片中, 叶绿素合成机制可能已被破坏, 叶绿素合成受阻导致的叶片内色素类物质含量降低或消失是叶片白化的直接原因。
叶片是作物光合作用的主要器官, 叶片的突变对其光合作用以及整个生长过程都有重要的影响。研究叶色突变体, 发掘与叶绿体形态建成以及叶绿素合成代谢相关的新基因, 探究作物光合作用机制, 具有重要的理论意义和应用价值。在高等植物中, 叶色变异种类繁多, 不同突变体的分子机制也较为复杂。总的来说, 突变基因通过不同的调控方式, 直接或间接影响叶绿素的合成及稳定, 最终导致叶色表型变化。根据目前对叶色突变体的研究结果, 叶色突变机制主要有叶绿素生物合成和降解途径中基因突变、血红素代谢途径的基因突变、编码其他叶绿体蛋白基因突变、与光合系统无直接关系基因突变几类[21]。其突变类型可能是数量性状, 也可能是质量性状; 可能是细胞核遗传, 也可能是细胞质遗传。通常情况下, 为了确定突变性状的遗传模式, 需通过正反杂交试验验证, 但由于茶树属于多年生木本植物, 受限于杂交周期、结实率及自交不亲和性, 难以得到像大田作物那样的重组近交系用于遗传作图, 分离群体的规模也受到很大限制。目前为止, 已报道的可用于遗传分析的茶树分离群体还不足10个, 其中没有一个是基于叶色突变建立的[22]。因此, 构建合适的茶树叶色突变分离群体, 将是解答叶色突变机制的重要途径。本实验室通过多年的重复杂交, 已初步建立了一个白化性状在子代中能有效分离的F1群体, 进一步的遗传分析还在进行中。
大量研究表明, 叶绿素的合成是由一系列酶共同作用的生化反应过程。其中任何一个编码酶基因的突变都有可能使相关酶的活力发生改变甚至丧失, 影响相关蛋白质的合成, 从而导致叶绿素合成过程受阻, 使叶色表现出不同程度的变化。在高等植物中, 参与叶绿素合成过程的所有酶编码基因均已被克隆鉴定[23]。但茶树作为重要的多年生叶用经济作物, 有关这一途径的功能基因研究却仍处在起步阶段, 目前仅有4个基因在GenBank登录, 分别为镁离子鳌合酶H亚基基因、谷氨酸-tRNA还原酶基因、叶绿素合酶基因和叶绿素酸醋氧化酶基因[24], 均为本实验室提交。其中谷氨酸tRNA还原酶是叶绿素合成的第一个关键酶, 它催化谷氨酰-tRNA转化为谷氨酰-半醛。研究表明, 该酶是一个具有一个V型折叠结构的二聚蛋白, 主要在叶绿体基质中发挥作用[25]。Kumar和Soll[26]将谷氨酞-tRNA还原酶的反义RNA转入拟南芥后, 转基因植株间即存在明显的叶色差异, 并且叶色变异程度与HEMA基因的表达水平成反比。在大多数植物中, 编码该酶的基因以多基因家族的形式存在[27]。例如在大麦中, 发现了3个不同的克隆能够编码该蛋白, 其中克隆BHA1和BHA87具有一致的开放阅读框, 但3° 非编码区的长度不一样; 而克隆BHA1和BHA13则有87个不同的核苷酸, 从而导致编码蛋白的11个氨基酸置换[28]。在茶树中我们也发现相似的情况, 通过RACE测序, 共克隆了3条序列相似度很高的茶树谷氨酸-tRNA还原酶基因, 序列分析表明它们具有相同的开放阅读框, 但在3° 端存在7个氨基酸置换位点, 这些位点的存在是否对其编码酶活性有影响还需进一步鉴定。叶绿素合酶是叶绿素合成途径中的一种关键酶, 定位在类囊体膜上, 催化叶绿素酸醋植醇化, 生成叶绿素a或叶绿素b, 这一步骤对叶绿素辅基蛋白的翻译和积累, 类囊体膜组分的稳定装配具有重要意义[29, 30]。在大多数高等植物中, 叶绿素合酶多以单拷贝基因的形式存在[31]。Lopez等[32]研究发现, 叶绿素合酶与细菌叶绿素合成酶属于聚异戊烯转移酶家族, 具有典型的聚异戊烯焦磷酸结合位点(domain II)。Schmid等[33]研究了燕麦叶绿素合酶基因异源表达蛋白特性, 揭示半胱氨酸、精氨酸残基和Mg2+是酶催化活性所必需。另外, 通过定向突变叶绿素合酶残基His-197, 能够使其活性丧失, 说明His-197可能与叶绿素酸酯中Mg2+的结合有关[34]。叶绿素a氧化酶是由叶绿素a生成叶绿素b的关键酶, 该酶编码基因的突变会导致叶绿素b的合成受阻, 甚至不能合成叶绿素b[35]。Tanaka等[27]利用插入诱变技术获得6个衣藻叶绿素b缺失突变体, 并从中首次克隆出叶绿素a氧化酶基因。Lee等[36]在水稻基因组分离出2个与CAO高度同源的基因, 并命名为OsCAO1基因和OsCAO2基因, 它们均位于水稻的第10染色体上, OsCAO1在光合组织中表达并受光的诱导, OsCAO1基因的失活将导致植株体内叶绿素b的合成受阻、含量减少和表现出苍白叶表型, 严重时甚至不能合成叶绿素b, 致使植株不能正常进行光合作用而早衰早亡。
白叶1号是浙江省安吉县茶农发现的一个温度敏感型白化突变资源, 因为其特异的外观特征与内在品质, 成为制作高档名优茶的理想原料。已有研究认为, 白叶1号阶段性返白是其内部基因突变的结果, 突变基因在一定温度范围内得以表达或表达加强[12], 其温度阈值被普遍认为在20~22℃之间, 当外界温度超过23℃度时, 白化叶片便开始复绿。但本实验室经多年田间观察发现, 最初白化的叶片基本不能完成复绿过程, 普遍在七月初干枯脱落, 复绿现象只能发生在叶脉尚未白化的新生叶芽, 说明叶片内部结构的破坏程度也是影响叶片复绿的一个重要因素。王新超等[37]利用mRNA差别显示技术研究了其正常叶片和白化叶片的基因表达差异, 结果从58个差异表达的片段中鉴定出12个阳性克隆, 通过Blast比对得到了5个有注释信息的基因片段, 分别为血红素结合蛋白家族中心区域基因、甲硫氨酸合酶基因、转座子基因、3-磷酸甘油醛脱氢酶假基因和ACC合成酶基因。李娟等[38]用SSH技术, 构建了白叶1号全白期与全绿期叶片的正反向消减文库, 并从中筛选到80个差异表达序列, 推测有3个基因片段可能与白叶1号高氨基酸形成相关。本实验室则采用芯片技术分析了白叶1号不同白化阶段的基因表达谱, 筛选到差异表达基因671个, 在所有差异表达基因中包含多个叶绿素合成相关的重要催化酶[14], 本研究通过RACE技术克隆了其中3个主要结构酶的全长序列。表达分析表明, CsChlS和CsCAO基因具有明显的表达协同性, 它们在叶片中的表达量与叶片的颜色变化高度同步; 而谷氨酰tRNA还原酶的荧光定量试验结果显示, 它在白化叶片和正常叶片中的表达差异相对较小, 同时在新生芽叶转绿的过程中, 它的表达最先恢复到正常水平。通过以上结果, 我们可以初步推测叶绿素合成途径受阻是安吉白茶白化表型的直接原因, 但具体是哪种突变引起了该连锁反应还有待进一步研究。
从茶树中克隆了3个编码叶绿素合成途径的重要催化酶基因CsGluTR、CsChlS和CsCAO, 它们均具有典型的结构特征, 且编码的氨基酸序列与其他植物中同源基因的相似性均在70%以上。通过对不同白化程度和不同部位茶树叶片中3个基因的表达进行分析, 发现CsChlS和CsCAO的表达量与叶片颜色变化密切相关。推测叶绿素合成受阻导致的叶片内色素类物质含量降低是安吉白茶白化表型的直接原因。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|