第一作者联系方式: E-mail: renqin12@mails.ucas.ac.cn
由致病疫霉( Phytophthora infestans)引起的晚疫病是最具毁灭性的马铃薯病害。为明确植物介导的RNAi沉默致病疫霉基因的有效性, 本研究采用重叠延伸PCR技术克隆同时与晚疫病菌4个 ces基因均同源的融合基因 C1234, 构建内含子连接的 C1234反向重复序列植物表达载体, 采用农杆菌介导法转化晚疫病易感马铃薯品种大西洋, 经PCR和Southern杂交检测, 获得129个转基因株系。离体叶片接种病原菌后, 有97个转基因株系发病速度明显慢于野生型, 接种6 d后病斑大小和霉层厚度均明显小于对照, 并且叶片感病部位没有出现失绿斑, 而野生型产生了明显的失绿斑。实时定量RT-PCR分析发现, 发病延缓的叶片上致病疫霉4个纤维素合酶基因的表达水平明显低于野生型。本研究表明, 转基因植株中产生的以晚疫病菌 ces基因为靶标的dsRNA能够沉默致病疫霉相应基因表达, 延缓发病进程。
Potato late blight caused by Phytophthora infestans is the most devastating disease in potato. The objective of this study was to test the efficiency of plant-meidated RNAi in silencing genes in P. infestans and to find a new way to breed potato resistant to late blight. Over-lap PCR was employed to amplify a fused-gene C1234 simultaneously homologous to four cellulose synthase genes in P. infenstans. Then, a plant expression vector containing inverted repeat of C1234 was constructed and transferred to Atlantic, a potato variety severely susceptible to late blight by agrobacteria-mediated genetic transformation. A hundred and twenty nine regenerated lines were confirmed to be transgenic plants with PCR and Southern blot. When detached leaves were inoculated with P. infestans, 97 out of 129 transgenic lines delayed disease symptoms with smaller lesions and less hyphae compared to the wild type at six days after inoculation. Chlorotic spots did not appeared on leaves from transgenic lines while developed severely on leaves from the wild type at the same day. mRNA accumulation of the four cellulose synthase genes from P. infestanscolonized on leaves from transgenic plants with delayed symptoms was significantly lower compared to the wild type with Real-time RT-PCR. The results demonstrated that dsRNA of C1234 generated in transgenic potato could induce homologous genes silence and delay the process of infection in intimately touched P. infestans.
致病疫霉[Phytophthora infestans(Mont.) de Bary]引起的晚疫病是马铃薯最具毁灭性的病害, 其防治难度远超水稻稻瘟病和小麦锈病, 是当前世界农业生产中危害最为严重的病害之一。晚疫病的发生和流行给马铃薯生产造成巨大损失, 并严重威胁着世界农业生产及粮食安全[1]。目前, 应对马铃薯晚疫病的策略主要有改进栽培措施、化学控制和使用抗病品种3种途径。栽培措施的改进对晚疫病发生和流行的影响十分有限, 而化学控制在一定程度上可以减轻致病疫霉的危害, 但费用极高, 并且使用的往往是一些非环境友好型农药, 造成巨大的生态风险[1]。使用抗病品种被实践证明是最为经济有效的马铃薯晚疫病控制途径。但由于致病疫霉具有极强的进化潜力[2, 3], 在不到5年时间里相继克服了作为马铃薯抗晚疫病育种主要抗性来源的11个R基因和多个新近开发、并被育种家寄予厚望的持久抗性基因[4, 5, 6]。随着致病疫霉A2交配型的出现及A2与A1交配型间有性繁殖的发生使得致病疫霉的变异速度更快、毒力变化更加复杂, 成为名副其实的抗性基因“ 摧毁者” , 导致马铃薯抗晚疫病育种举步维艰[3, 4]。针对这一现状, 有学者提出了利用抗病基因多样性持久防治晚疫病的策略[7]。尽管这一策略在水稻病害控制中得到成功应用[8, 9], 但马铃薯抗病基因多样化的实现需要导入大量的抗性基因, 以构建晚疫病抗性基因近等混合系。这是一个漫长的过程, 目前开发的马铃薯晚疫病抗病基因资源仍然十分有限, 使得该策略短期内难以应用于抗晚疫病育种实践。所以, 探索新的更有效的马铃薯抗晚疫病育种策略显得尤为重要。
RNA干扰(RNA interference, RNAi)是一种双链RNA(double-stranded, dsRNA)介导的序列特异的基因沉默技术, 能够高频、高效、高度特异地引起与dsRNA同源的基因的沉默[10], 并且RNAi信号可以在密切接触的物种间相互转移[11]。该技术已在增强植物抵抗病毒[12, 13]、细菌[14]、真菌[15, 16, 17]、杂草[18]及虫害[19, 20, 21]研究中得到成功应用, 但在增强植物对卵菌抗性的研究方面尚无成功报道。本研究拟克隆同时与致病疫霉4个纤维素合酶基因同源的融合片段C1234, 转化ihpRNA载体至易感马铃薯品种大西洋, 观察检测转基因植株叶片接种病原菌后的发病情况, 以期探索植物介导的RNAi能否干扰致病疫霉基因的表达。
马铃薯(Solanum tuberosum L.)品种大西洋脱毒试管苗由甘肃农业大学张金文教授提供, 致病疫霉由甘肃省农业科学院植物保护研究所分离, 载体pHANNIBAL和pART27由澳大利亚Peter M Waterhouse博士惠赠[12], 大肠杆菌DH5α 、农杆菌LBA4404由本实验室保存, 植物RNA提取试剂盒Concert Plant RNA Reagent和反转录试剂盒购自Life Tech公司, 克隆载体pGEM-T easy vector购自Promega公司, 限制性内切酶、T4DNA连接酶和高保真DNA聚合酶购自TaKaRa, DNA Marker、凝胶回收试剂盒购自TIANGEN公司, 尼龙膜(nylon membrane, positive geladen)和Southern blot探针标记及检测系统均购自Roche公司。其他试剂均为国产分析纯。
根据GenBank中公布的致病疫霉纤维合成酶编码基因ces1(GenBank登录号为EF563993)、ces2 (GenBank登录号为EF563994)、ces3(GenBank登录号为EF563995)和ces4(GenBank登录号为EF563996)碱基序列, 采用生物学软件Oligo 6设计重叠延伸PCR引物, 为方便后续载体构建, 在ces1干扰片段上游引物和ces4干扰片段下游引物5° 段分别引入酶切位点(表1)。然后, 参照Goodwin等[22], 采用CTAB法提取晚疫病菌基因组DNA, 以其为模板采用重叠延伸PCR技术扩增4个干扰片段及融合基因。具体操作流程为: 分别配置以1u和1l2u、2u1l和2l3u为引物对的PCR反应液, 先进行5个循环的扩增, 然后将2种反应液等体积混合后继续扩增25个循环, 琼脂糖凝胶电泳后切胶回收目的条带, 得到ces1和ces2基因干扰片段的融合基因C12; 分别以3u2l和3l4u、4u3l和4l为引物采用同样方法得到ces3和ces4基因干扰片段的融合基因C34。最后, 以等量的C12和C34为模板, 以1u和4l为引物进行PCR, 琼脂糖凝胶电泳分析PCR产物, 切胶回收目的条带C1234。将C1234与pGEM-T easy vector连接, 连接产物转化大肠杆菌DH5α 感受态细胞, 得到含有4个纤维素合成酶基因干扰片段融合基因的重组质粒pTC1234, 送交基因测序公司进行序列分析。
先用限制性内切酶Xho I/EcoR I消化pTC1234, 琼脂糖凝胶电泳后回收小片段(C1234), 与经同样酶切并回收的载体pHANNIBAL连接, 转化大肠杆菌DH5α , 得到重组质粒pHCS; 再用Xba I/Hind III消化pTC1234并回收小片段(C1234), 与经同样酶切的pHCS载体相连, 得到中间载体pHIVC; 最后, 用Not I消化pHIVC, 回收大片段, 与经同样酶切并回收的pART27载体相连, 得到具有融合基因C1234反向重复序列的植物表达载体pRNAiC。
| 表1 重叠延伸PCR扩增引物 Table 1 Primers of over-lap PCR |
采用直接导入法[23]将pRNAiC导入农杆菌LBA4404, 获得用于进行马铃薯遗传转化的工程菌, 以大西洋试管苗叶片为受体材料参照Gustafson等[24]进行转化。当获得的Kan抗性苗长到2 cm左右时, 将其切下转入附加80 mg L-1 Kan的MS培养基进行生根筛选。
采用CTAB法[25]提取能在生根培养基上形成完整根系、生长旺盛的转化再生苗叶片基因组DNA。以提取的基因组DNA为模板, 1u为正向引物、4l为反向引物进行PCR扩增, 筛选转基因株系, 然后在对PCR检测呈阳性的株系进行Southern杂交, 亦即将PCR检测呈阳性的植株基因组DNA经Not I过夜消化后用0.8%的琼脂糖凝胶分离, 再用20× SSC转移到尼龙膜, 80℃固定2 h后与探针杂交。探针制备以纯化的C1234为模板, 采用随机引物标记法标记, 杂交及检测的其他步骤均按照DIG High Prime DNA Labeling and Detection Starter Kit I所述方法进行。
转基因植株移栽后50 d左右鉴定抗病性。参照Grenville-Briggs等[26]方法接种, 采用悬浮液滴接种法接种离体叶片, 接种3 d后统计病斑生长速率(lesion growth rate, 简称LGR)。即测量接种点的病斑大小, 连测4 d。测量每个病斑最长和最宽处(两者成直角), 其面积A = 1/4× π × 长× 宽, 根据病斑面积, 将病斑划为成功与非成功侵染2种情况。如果病斑大小局限于接种点内, 即0< A< 16 mm2, 则视为非成功侵染; 如果病斑面积在几次测量中都小于接种点面积, 则视为过敏性反应; 如果病斑面积有一次超过16 mm2, 即被视为成功侵染, 对成功侵染的病斑, 根据其面积计算病斑的平均半径, 以时间(d)为横坐标, 以病斑平均半径为纵坐标, 通过线性回归, 估计LGR[27]。
随机选取3株转基因植株进行实时RT-PCR分析纤维素合成酶基因ces1、ces2、ces3和ces4基因mRNA积累水平, 实时RT-PCR采用SYBR Green I 法[Premix Ex Taq (Perfect Real Time)试剂盒], 引物序列、分析方法参照Grenville-Briggs等[28]。
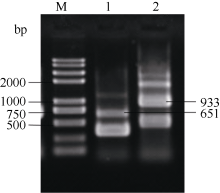
以马铃薯晚疫病菌基因组DNA为模板, 以1u和1l2u、2u1l和2l3u为引物对进行重叠延伸PCR扩增后电泳分析, 在650 bp左右处出现条带(图1, 泳道1), 大小与ces1和ces2干扰片段之和相符, 初步说明成功扩增了ces1和ces2干扰片段的融合基因C12; 以3u2l和3l4u、4u3l和4l为引物对扩增后在940 bp左右处出现电泳条带(图1, 泳道2), 与ces3和ces4干扰片段之和相符, 初步说明成功扩增了ces3和ces4干扰片段的融合基因C34; 以等量C12和C34为模板, 1u和4l为引物扩展后在1600 bp左右处出现电泳条带(图2, 泳道1和泳道2), 初步说明成功扩增了ces1、ces2、ces3和ces4干扰片段融合基因C1234。将C1234与克隆载体pGEM-T easy vector连接后分析碱基序列表明, 组成融合基因的4个片段碱基序列与GenBank中公布的相应序列完全相符, 说明获得了含有同时以4个致病疫霉ces为靶标的干扰片段的载体pTC1234。
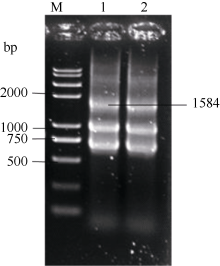 | 图2 C1234重叠延伸PCR扩增M: trans2K plus II DNA marker; 1~2: C1234重叠延伸PCR扩增 产物。Fig. 2 Results of over-lap PCR amplification for C1234M: trans2K plus II DNA marker; 1: over-lap PCR for C1234. |
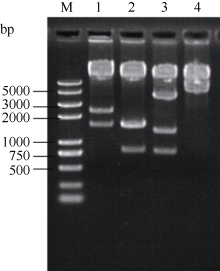
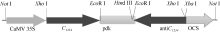
根据构建的pRNAiC载体上C1234反向重复序列表达框架的限制性酶切位点分布特点对其进行酶切分析, 经Xba I/Hind III消化后, 在1600 bp处出现特异条带(图3, 泳道1), 说明C1234反向插入pdk内含子和OCS终止子之间。经Xho I/EcoR I消化后分别在1600 bp和740 bp左右处出现特异条带(图3, 泳道2), 分别与C1234和pdk内含子的大小相符, 并且1600 bp处条带亮度是相邻泳道1600 bp处条带的2倍左右, 说明经该内切酶组合消化, 既切下了正向的C1234, 也切下了反向的的C1234, 也说明pdk内含子两端分别连接了正向和反向的C1234。经Not I/Xho I消化, 分别在4000、1300和740 bp左右处出现特异条带(图3, 泳道3), 其大小与“ C1234+pdk内含子+反向C1234” 、CaMV 35S启动子和OCS终止子大小相符, 进一步说明C1234以正确的方向分别插入到了pdk内含子的上游和下游, 构建了以CaMV 35S为启动子、OCS为终止子的内含子连接的C1234反向重复序列表达框架。经Not I消化, 在6000 bp左右处出现特异条带(图3, 泳道4), 其大小与“ CaMV 35S+ C1234+pdk内含子+反向C1234+OCS终止子” 相符, 进一步说明成功构建了pdk内含子连接的C1234反向重复序列表达框架, 并插入到了植物表达载体pART27的Not I位点之间。C1234反向重复序列表达框架结构如图4所示。
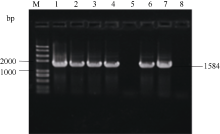
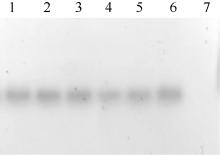
转化大西洋叶片, 获得278株Kan抗性再生苗。将抗性再生苗转接到含Kan的生根筛选培养基上, 有166株形成了完整根系。对这166株植株进行PCR鉴定, 其中有129株在1600 bp左右处出现特异条带, 而作为阴性对照的非转化植株则未出现相应条带(图5), 说明融合基因C1234反向重复序列整合到受体品种基因组中, 进一步对部分PCR检测呈阳性的植株的Southern杂交发现, 基因组DNA经Not I消化后杂交, 所有株系均在相同大小处出现杂交条带, 由于植物表达载体pHIVC左右边界内2个Not I位点之间是C1234反向重复序列表达框架和Kan抗性标记基因表达框架, Southern杂交均出现大小一致的条带, 说明C1234反向重复序列表达框架和Kan抗性标记基因表达框架均整合到了马铃薯基因组中, 获得了转基因植株, 也进一步说明Kan抗性生根筛选具有较强可靠性。
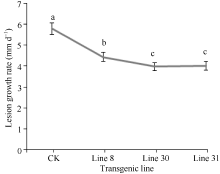
离体叶片接种致病疫霉后, 从第4天开始逐渐在叶片的接种部位及附近出现白色菌丝, 随着时间延长, 产生大量菌丝体, 并且病斑范围不断扩大, 形成不规则的覆盖白色霉层的病斑; 从第5天开始, 叶片的病斑部位及其周围逐渐失绿。与非转基因的野生型对照相比, 转基因植株中有97个株系发病速度明显慢, 占总转基因株系75.2%, 病斑生长速率显著小于
野生型对照(图7)。发病过程中, 菌斑明显小于对照, 霉层也明显比对照薄(图8), 并且失绿斑比对照晚出现2 d以上。说明转基因植株中形成的同时与致病疫霉ces1、ces2、ces3和ces4基因同源的dsRNA诱导了晚疫病菌相应基因表达的下调, 延缓了发病进程。
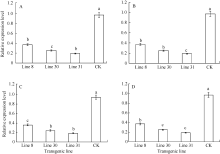
从97个发病延缓的转基因株系中选取3个独立株系采用实时RT-PCR分析纤维素合酶基因ces1、ces2、ces3和ces4的mRNA积累水平(图9), 发现侵染这3个转基因株系的晚疫病菌相应基因的mRNA积累水平显著低于对照, 其中, 侵染Line 8的ces3基因mRNA表达量下调最显著, 侵染Line 30的致病疫霉ces1和ces3基因mRNA表达量也显著下调, 侵染Line 31的ces2和ces4基因的mRNA表达量也下调明显, 并且不同的转基因株系同一基因的mRNA下调幅度水平不同, 同一株转基因株系不同基因mRNA下调幅度水平也不完全一样。
 | 图8 离体叶片接种致病疫霉6 d后病斑和失绿斑A: 转基因株系; B: 野生型。A: transgenic lines; B: wild type.Fig. 8 Lesions and chlorotic spots on detached leaves six days after P. infestans inoculation |
植物介导的有害真核生物RNAi是通过在植物中形成dsRNA或siRNA等RNAi信号分子, 经寄生、取食等途径传递到有害真核生物中, 激发入侵有害真核生物的RNAi机制, 引起相应基因的沉默, 作为一种类似“ 报复” 的行为给予入侵有害生物以精确打击[29, 30, 31], 已经在植物抗虫、抗真菌、抗寄生杂草研究中得到了成功应用。采用该策略沉默入侵生物基因的关键是作为干扰信号的dsRNA或siRNA的在植物中必须有一定的积累, 由于RNAi干扰存在剂量效应, 只有植物细胞中积累的RNAi信号分子量足够多或病原物入侵后寄主细胞仍然能够持续产生RNAi信号, 才能保证传递到入侵真核生物中RNAi信号的量达到触发RNAi的阈值, 所以目前该策略主要成功应用于病原物和寄主长期共存的活体营养型病原菌引起的病害研究[15, 16]。而晚疫病菌为半活性营养型病原菌, 入侵植物后会杀死细胞, 终止RNAi信号的持续产生, 采用该策略实现对致病疫霉基因的沉默目前尚未有报道, 而已有的关于植物介导的RNAi沉默另一种卵菌类病原菌寄生疫霉的研究发现转基因植株产生的dsRNA不足以触发其RNAi机制而引起相应基因的沉默[32]。本研究构建了双子叶植物中的强启动子CaMV 35S启动子驱动的同时与马铃薯晚疫病菌4个纤维素合成酶基因同源的C1234反向重复序列表达载体, 转化马铃薯获得了129个转基因株系, 其中有97个转基因株系接种晚疫病菌后延缓发病, 病斑生长速率显著低于对照, 并且发病延缓的叶片上寄生的晚疫病菌4个纤维素合成酶基因mRNA积累水平也显著低于野生型, 说明对致病疫霉这种半活体营养型病原菌采用植物介导的RNAi能够沉默其基因表达, 并且转基因植株中能够引起致病疫霉基因沉默的株系出现频率较高, 本研究中引起其基因沉默的转基因株系占总转基因株系的75.2%。这说明马铃薯介导的RNAi能够有效沉默入侵致病疫霉基因表达。植物介导RNAi能够引起致病疫霉基因沉默, 而对寄生疫霉却没有作用, 引起这一差异的原因可能是不同卵菌间对环境RNAi信号吸收能力存在差异。
本研究以致病疫霉纤维合酶家族的4个纤维素合成酶基因同时为靶标, 这些基因为非致死基因, 但其沉默会引起致病疫霉细胞壁纤维含量下降, 影响正常附着孢的产生和萌发管的形成, 一定程度上影响了致病疫霉的致病性[28]。由于植物介导的RNAi干扰对基因的沉默是不完全的, 只是一定程度下调了相应基因的表达, 不足以彻底阻止病菌侵染, 而晚疫病菌一旦侵染马铃薯, 纤维合酶基因表达水平就不会对病菌致病能力产生强烈影响。故本研究中转基因株系接种致病疫霉后仅仅延缓了发病进程, 而没有产生强烈抗性。故后续采用植物介导的RNAi增强对晚疫病的抗性研究中, 在靶标基因选择上应借鉴在抗真菌研究中的成功经验[15, 16, 17], 选择致病疫霉致死基因或对其生长发育至关重要的关键基因, 使植物的“ 报复” 行为更有效。
通过在马铃薯中表达与致病疫霉基因同源的dsRNA能够下调入侵晚疫病菌相应基因表达, 赋予马铃薯对入侵病菌特定基因精确打击的能力。通过马铃薯介导的致病疫霉RNAi同时沉默晚疫病菌4个纤维素合酶基因表达能够延缓马铃薯晚疫病发病进程。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|