水稻蔗糖非酵解型蛋白激酶SnRK2, 又称胁迫相关蛋白激酶(stress-activated protein kinase genes in rice, OsSAPKs), 在调控水稻非生物胁迫信号传导中起着重要作用。本研究对 OsSAPK2的结构及其在水稻抗白叶枯病反应中的功能进行了初步研究。结果表明 OsSAPK2被定位于细胞核和细胞质内, 与 OsSAPK1、OsSAPK3同属于Kulik’s II组。 OsSAPK2-RNAi转基因水稻中 OsSAPK2下调表达, 人工接种水稻白叶枯病菌后, 转基因水稻比受体对照的病斑长度显著增长, 抗病相关基因 OsLRR1、OsHIR1表达水平下降, 感病相关基因 OsMAPK5表达水平升高。此外, OsSAPK2具有自激活活性, 可能与 OsMAPK5等胁迫相关蛋白互作。上述结果为进一步研究 OsSAPK2调控水稻抗白叶枯病的分子机制提供了信息。
Sucrose nonfermenting1-related protein kinase2 (SnRK2), also known as stress-activated protein kinase ( OsSAPKs), plays an important role in signal transduction. In this study, we analyzed the structure and function of OsSAPK2 in response to Xanthomonas oryzaepv. oryzae ( Xoo) infection. The result suggested that OsSAPK2 is a member of Kulik’s II group like OsSAPK1, OsSAPK3 and located in nucleus and cytoplasm. OsSAPK2and disease-resistant genes OsLRR1, OsHIR1 were down regulated in OsSAPK2-RNAi transgenic rice, while disease-related gene OsMAPK5 was up regulated. Compared with non-transgenic plants, transgenic plants were more susceptible to Xoo infection. OsSAPK2 could activate itself and interact with several stress-related proteins. These results indicate that OsSAPK2 might be involved in the regulation of resistance response by regulating the expression of OsLRR1, OsHIR1, OsMAPK5 and interacting with stress-related proteins.
植物在整个生长发育过程中受多种外界环境的影响, 包括病原菌的侵染生物胁迫以及干旱、寒冷、高盐等非生物胁迫。经过长期进化, 植物发展出响应不同环境胁迫的适应机制[1], 能够识别外界胁迫信号, 将信号传递到不同的信号元件中, 迅速做出一系列防御反应。其中, 蛋白质的可逆磷酸化(主要通过蛋白激酶和磷酸酶发挥作用)是高等植物重要的信号转导方式之一[2, 3]。
蔗糖非酵解型蛋白激酶(sucrose non-fermenting1- related protein kinase, SnRK)是一类广泛存在于植物中的蛋白激酶, 包括SnRK1、SnRK2和SnRK3三个亚家族, 参与糖代谢途径并对干旱及盐胁迫等多种渗透逆境有应答作用[4]。SnRK2家族是植物中一类特异的Ser/Thr类蛋白激酶, 分为3组, Kulik’ s I组, 激酶不受ABA激活; Kulik’ s II组, 受ABA微弱激活; Kulik’ s III组, ABA能够完全激活该家族成员[5]。通过生物信息学分析与试验相结合, 已从众多物种的基因组数据库中鉴定出大量的SnRK2家族成员。拟南芥、水稻和高粱中均含有10个SnRK2家族成员[6, 7, 8], 玉米中有11个SnRK2家族成员[9], 烟草、小麦、大豆等作物基因组中也鉴定出一些SnRK2基因[10, 11, 12, 13, 14, 15, 16]。Mikoł ajczyk等[16]研究发现烟草BY-2悬浮细胞渗透胁迫可以激活渗透胁迫激活蛋白(NtOSAK)。大豆中的SnRK2成员SPK1和SPK2可以被高渗透逆境诱导激活, 而SPK3和SPK4可被脱水和高盐激活, SPK3还可以被外源ABA激活[17]。水稻基因组中包括SnRK2s家族10个基因, 被命名为OsSAPK1-10 (stress-activated protein kinase genes in rice, OsSAPKs), 这10个SnRK2成员均可被NaCl所诱导激活, 其中SAPK8、SAPK9和SAPK10可以被ABA激活[6]。近年来SnRK2基因家族参与调控植物非生物胁迫反应的报道越来越多[18, 19, 20], 但其在生物胁迫中的功能鲜有报道。
由Xanthomonas oryzaepv. oryzae(Xoo)引起的水稻白叶枯病是我国和亚洲其他国家稻作栽培中一种重要的细菌病害, 在我国华南、华中和华东地区, 以及东南亚稻区和非洲稻区普遍发生[21, 22]。本实验室前期对OsSAPK基因家族的研究中初步发现不同水稻品种接种白叶枯病菌后, OsSAPK2表达水平存在明显差异[23]。本研究利用RNAi技术对OsSAPK2 (LOC_Os07g42940)进行抗病功能验证, 通过蛋白序列分析, 酵母双杂交和荧光定量PCR技术分析OsSAPK2互作蛋白和部分已知抗病相关基因的表达水平, 初步探讨了OsSAPK2参与调控水稻抗白叶枯病的分子机制。
根癌农杆菌(Agrobacterium tumefaciens)菌株EHA105、白叶枯病菌菌株PXO61(P1)、KS6-6(C2)均由本实验室保存。大肠杆菌(Escherichia coli)菌株DH5α 购于北京全式金公司。根瘤农杆菌(Agrobacterium tumefaciens) EHA105、酵母(Saccharomyces cerevisiae)菌株AH109由本实验室保存。实验所用载体pDONR201、pH7GWIWG2(II)购于Invitrogen公司, pGBKT7购于Clontech公司。
根据NCBI在线BLAST对水稻基因LOC_ Os07g42940 (OsSAPK2)序列进行分析, 选择其中705 bp的特异核苷酸序列作为RNAi干扰区段。根据特异核苷酸序列设计引物SAPK2-RNAi-F/SAPK2- RNAi-R (表1), PrimeSTAR PCR扩增OsSAPK2干扰区段, 以Gateway技术构建RNAi抑制表达载体(pH7GWIWG2(II)-OsSAPK2; 图1-A)。
根据参考序列日本晴OsSAPK2序列, 利用Primer 5.0设计引物SAPK2-EcoRI-F/SAPK2-BamHI- R (表1), PrimeSTAR PCR扩增获得OsSAPK2编码区序列1020 bp, 以酶切连接方法构建酵母双杂交诱饵表达载体(pGBKT7-OsSAPK2; 图1-B)。
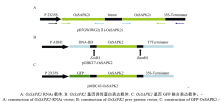 | 图1 OsSAPK2基因RNAi表达载体、酵母双杂交诱饵载体和GFP融合载体示意图Fig. 1 Construction of RNAi, prey protein vector of Y2H, and GFP fused vector of OsSAPK2 |
| 表1 本研究所用引物 Table 1 Primers used in this study |
根据参考序列日本晴OsSAPK2序列, 利用Primer 5.0设计引物SAPK2-CDS-F/SAPK2-CDS-R (表1), PrimeSTAR PCR扩增获得OsSAPK2编码区序列1020 bp, 以Gateway技术构建GFP-OsSAPK2融合表达载体(pMDC43-OsSAPK2; 图1-C)。
水稻、玉米、拟南芥、马铃薯和白杨SnRK2家族成员蛋白序列来自http://www.uniprot.org/, 利用Cluster W进行序列比对, MEGA5进一步构建进化树。
将含有目标基因载体pMDC43-OsSAPK2用基因枪介导法转化至洋葱表皮细胞中, 以不含目的基因的空载体pMDC43为阳性对照, 用激光共聚焦显微镜观察。
将PH7GWIWG2(II)-SAPK2载体转化到农杆菌EHA105, 用农杆菌介导法转化粳稻品种9804的水稻愈伤组织, 以潮霉素(50 mg L-1)筛选阳性愈伤组织, 进而再生获得转基因植株(9804-SAPK2i)。
利用标记基因潮霉素(Hpt)特异引物Hpt-F/R (表1)对9804-SAPK2i的不同转化体T2代转基因株系进行PCR检测, 以质粒pH7GWIWG2(II)为阳性对照, 1%琼脂糖凝胶电泳, 成像观察。
以蔗糖磷酸合成酶基因(SPS)为内源参照基因(单拷贝), 标记基因潮霉素抗性基因(Hpt)作为外源基因设计引物SPS-RT-F/R、Hpt-RT-F/R, 利用qPCR进行拷贝数分析。参照杨立桃等构建校正标准曲线与线性方程[24]。
2014年夏天, 在中国农业学科院作物科学研究所网室进行抗病表型鉴定。采用人工剪叶接种法在植株分蘖盛期进行接种白叶枯病菌代表菌株PXO61(P1)和KS6-6(C2), 2周后测量病斑长度。接种后0、12、24、36、48、60和72 h取样, 液氮速冻后储存于-80℃冰箱备用, 每个样品3个重复, 每个重复5株。用Invitrogen公司的TRIzol试剂提取水稻叶片总RNA, DNase I处理后, 用Promega公司试剂反转录成cDNA, -20℃保存备用。
以水稻Actin (Os03g0718100)作为内参设计引物Actin-F/Actin-R (表1), 用目的基因的特异引物SAPK2-RT-F/SAPK2-RT-R (表1)检测转基因植株中OsSAPK2基因的表达。根据抗病相关基因OsLRR (LOC_01G59440)、OsHIR1 (LOC_08G30790)、MAPK5 (LOC_03G55560)在NCBI中编码区序列分别设计引物01g59440-F/01g59440-R、08g30790-F/08g30790-R、03g55560-F/03g55560-R (表1), PCR扩增检测稳定后用于荧光定量PCR。
用qRT-PCR试剂盒(TransStart Top Green qPCR SuperMix, TransGen)于Bio Rad CFX96 Real time System上进行实时荧光定量PCR分析, 此反应体系为20 μ L [2× TransStart Top Green qPCR SuperMix 10 μ L、Passive Reference Dye (50× ) 0.4 μ L、上下游引物(20 μ mol L-1)各0.2 μ L、cDNA 2.0 μ L、ddH2O 7.2 μ L]。扩增反应程序为94℃, 30 s; 94℃, 5 s; 60℃, 34 s; 40个循环。每个取样点设3个重复, 采用2-Δ Δ Ct法分析确定基因的相对表达量。
依照Clontech (Clontech Laboratories, Inc, USA)酵母实验手册进行酵母转化和酵母双杂交实验。将构建好的诱饵载体(pGBKT7-SAPK2)转化酵母菌株AH109, 涂于色氨酸缺陷的SD平板(-Trp, SD), 在30℃培养箱中生长3~5 d后观察菌落的生长状况, 挑取单菌落于5 mL液体色氨酸缺陷的SD液体培养基中, 30℃条件下220转 min-1摇菌培养16 h, 按100倍、10-1倍、10-2倍、10-3倍、10-4倍、10-5倍梯度稀释, 并点涂在SD缺陷平板上培养(-Trp, -Trp/-His, -Trp/-Ade, -Trp/-His/-Ade, SD), 观察菌落生长状况。
酵母双杂交筛选: pGBKT7-OsSAPK2与pGADT7文库(接种白叶枯病菌的水稻酵母次级文库)质粒, 共转化后将菌液涂在加入定量3-AT营养缺陷培养基平板上(-Trp/-His/-Ade/-Leu, SD), 在30℃培养箱中生长3 d后观察菌落的生长状况, 挑取筛选培养基平板(-Trp/-His/-Ade/-Leu, SD)上的单克隆到筛选液体培养中, 30℃条件下220转 min-1摇菌培养, 并进行PCR检测, 挑取扩增出特异条带菌液测序, 分析目的基因。
测序结果与水稻基因组比对所获得的候选互作蛋白基因, 分别利用Primer 5.0设计引物, 荧光定量PCR进行表达分析。
通过对OsSAPK2基因的cDNA及蛋白质序列的分析, 发现水稻品种9804基因组中OsSAPK2的序列与数据库中报道的日本晴序列完全一致, 具有丝苏氨酸蛋白质激酶(Serine/Threonine protein kinases)和催化结构域(catalytic domain), 此外还含有一个特殊的STYKC结构域[23]。
选取多个植物物种的SnRK2家族成员, 利用Cluster W进行序列比对, MEGA5进一步构建进化树。结果显示, 不同植物SnRK2家族成员均可分为3类, 单子叶的水稻和玉米具有高度的同源性, 双子叶的拟南芥、马铃薯和白杨之间的距离较近(图2-A), 暗示单双子叶的分化位于OsSAPKs基因不同分组后, 可能由于被子植物分化为单子叶植物和双子叶植物过程中, 外界环境的巨变, 主要为非生物胁迫, 其自然选择压力对植物SnRK2家族原始遗传信息进行改变和修饰, 进行不同程度的复制加倍, 使该家族同源基因数目增加, 单双子叶的序列差异增大[25, 26]。OsSAPKs可以分为3组, 其中OsSAPK4、OsSAPK5、OsSAPK6、OsSAPK7属于Kulik’ s I组, OsSAPK1、OsSAPK2、OsSAPK3属于Kulik’ s II组, OsSAPK8、OsSAPK9、OsSAPK10属于Kulik’ s III组(图2-B); 用基因枪将载体pMDC43-OsSAPK2转化至洋葱表皮细胞中, 瞬时表达检测发现GFP-OsSAPK2融合蛋白定位在细胞质和细胞核中(图2-C), OsSAPK2可能与细胞质内相关蛋白和细胞核内相关蛋白都互作, 进而达到信号快速传递的目的。
利用标记基因hpt特异引物对9804-SAPK2i转基因不同独立转化体的基因组DNA进行PCR检测, 琼脂糖凝胶电泳结果显示, 转化体株系大部分能扩增出于质粒载体潮霉素基因一致的特异条带(图3-A标记基因PCR检测电泳图), 表明带有hpt/hyg标记基因的T-DNA已经插入并整合到受体基因组中, 仅有极少数具有潮霉素抗性的转化体呈现假阳性。本实验获得11个9804-SAPK2i独立的转化体。
对9804-SAPK2-RNAi转基因株系目的基因OsSAPK2进行qRT-PCR, 分析各转化体T2代植株OsSAPK2的表达量变化。结果表明4个9804-SAPK2- RNAi转化体的OsSAPK2的表达水平有不同程度的降低, 其中2个转化体目的基因表达水平显著低于对照9804 (图3-B)。
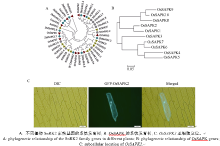 | 图2 SnRK2家族基因的系统发育分析和OsSAPK2亚细胞定位Fig. 2 Phylogenetic analysis of SnRK2 family genes and subcellular location of OsSAPK2 |
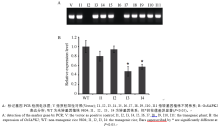 | 图3 OsSAPK2-RNAi转基因水稻PCR鉴定和OsSAPK2表达分析Fig. 3 Identification of OsSAPK2-RNAi transgenic rice plants by PCR and the expression of OsSAPK2 |
根据杨立桃等[24]的方法构建的Hpt和SPS基因的标准曲线如图4所示, Hpt标准曲线的相关系数R2=0.983, 相关性高, 该基因Ct值与起始模板数(Hpt0)之间的相关性方程为Hpt0=10(-0.29CT+10.07); SPS基因标准曲线的相关系数为R2=0.997, 相关性高, 该基因Ct值与起始模板数(SPS0)之间的相关性方程为SPS0=10(-0.314CT+13.149), 利用qPCR分析了9804-SAPK2- RNAi转基因株系(T2)拷贝数(表2), 计算公式为K=2× Hpt0/SPS0。其中, 转基因株系I1、I4的拷贝数为2, I3、I5、I6的拷贝数为1, I2与对照9804的拷贝数为0。结合表达分析发现, 拷贝数对目的基因的表达水平有一定的影响, 但影响程度并不完全一致, 可能与目的基因的插入位置有关。
在分蘖盛期采用剪叶法接种白叶枯病菌代表菌株P1和C2, 连续2周观察发病情况。接种3 d后, 可以观察到感病品种9804、9804-SAPK2i均有明显的暗绿色水浸状线状病斑; 7 d后, 病斑沿叶脉扩展, 变成黄褐色, 二者病斑长度无明显差异; 2周后, 发病情况稳定, 病斑呈现枯白色; 对于不同小种9804-SAPK2i的病斑长度均大于9804, 9804-SAPK2i对白叶枯病菌比9804更为敏感; 相同转基因株系接种菌株C2的病斑均比接种P1时高, 转基因植株对C2的感病性更强, 而对照9804对P1更敏感(图5)。
| 表2 qRT-PCR分析OsSAPK2-RNAi转基因株系中T-DNA插入拷贝数 Table 2 Analysis of OsSAPK2-RNAi (T-DNA) copy number of transgenic lines by qRT-PCR |
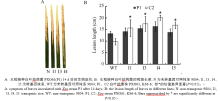 | 图5 OsSAPK2-RNAi转基因水稻接种白叶枯病菌的反应Fig. 5 Reaction of the OsSAPK2-RNAi transgenic rice toXanthomonas oryzae pv. oryzae(Xoo) infection |
SnRK2家族的激酶通常位于胁迫的上游信号转导途径[27, 28, 29]。对3个已知的抗病相关基因进行的表达分析, 抗病相关基因OsLRR1、OsHIR1在OsSAPK2-RNAi转基因植株中表达水平下降, 而感病相关基因OsMAPK5在OsSAPK2-RNAi转基因植株表达水平升高(图6), 推测OsSAPK2可能通过正向调控下游抗病相关基因OsLRR1、OsHIR1, 负向调控感病相关基因OsMAPK5而参与水稻对白叶枯病的抗性反应。
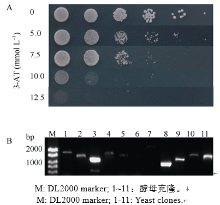
OsSAPK2属于丝氨酸/苏氨酸蛋白激酶。为了研究OsSAPK2是否具有自激活活性, 我们将OsSAPK2插入到pGBKT7, 转入酵母AH109中, 以BD为阴性对照, 结果显示OsSAPK2具有自激活活性。随后涂布在含有0、5、10、12.5 mmol L-1 3-AT的SD缺陷平板上培养(-Trp, -Trp/-His, -Trp/-Ade, -Trp/- His/-Ade, SD), 观察菌落的生长, 结果表明在12.5 mmol L-1 3-AT的SD缺陷平板上, 酵母不能正常生长(图7-A)。
利用诱饵载体(BD-OsSAPK2)进行酵母双杂交互作蛋白的筛选, 共筛选到247个阳性克隆, PCR检测结果如图7-B, 挑取扩增大小为1000 bp及以上的单克隆测序。测序结果与水稻基因组比对获得了多个候选互作蛋白(表3), 包括已知在植物抵御非生物胁迫过程中起作用的OsEREBP1、OsPTF1等。
为进一步深入了解候选蛋白的功能, 对候选蛋白基因进行了表达分析。结果表明, 正常处理下, 候选蛋白基因在OsSAPK2-RNAi转基因植株中表达水平均高于未转基因植株(图8)。接种白叶枯病菌后, LOC_Os09g07570、LOC_Os02g54160、LOC_Os10g 35840、LOC_Os07g32400的表达水平在OsSAPK2-RNAi转基因植株中逐渐降低, 而在未转基因植株表现升高或变化缓慢; LOC_Os07g05820在转基因和未转基因植株中表达均升高; LOC_Os06g09370在转基因植株中表达升高, 未转基因植株中表达变化趋势不明显。推测可能是由于OsSAPK2-RNAi转基因植株中的OsSAPK2基因被敲除, 引起的不同候选蛋白基因的差异表达。
| 表3 酵母双杂交筛选到与OsSAPK2互作的候选蛋白 Table 3 Candidate interaction proteins of OsSAPK2 screened by Y2H |
本研究利用RNAi技术分析了OsSAPK2基因在水稻对白叶枯菌的抗性反应中的作用。植物的免疫反应是一个复杂的调控网络, 网络中有些因子的功能是冗余的, 对这些因子的沉默可能因为其他因子的补偿而无法引起植株表型的变化。如拟南芥的WAK基因间存在功能的冗余, 利用基因特异的反义链转录本单独沉默AtWAK1或AtWAK2均不能引起拟南芥沉默植株的表型变化[30]。本研究发现接种白叶枯病菌不同菌株后, OsSAPK2基因的下调表达水平不同, RNAi植株接种P1表型变化差异明显小于接种C2白叶枯小种, 可能由于OsSAPK2在响应不同白叶枯菌小种抗性反应中起到的作用是不同的, 也可能是在应对不同病菌生理小种时存在与OsSAPK2基因功能冗余的基因。
番茄细菌性斑点病菌侵染拟南芥时, 类受体蛋白激酶OsLRR1能够减轻病原菌数目、提高防卫反应标记基因(如PR基因)的表达[31]; OsHIR1属于PID (Proliferation, Ion and Death)超家族成员, 在植物受到病原菌袭击的时候参与自发超敏反应坏死斑的形成过程[32]。丝裂原活化蛋白激酶OsMAPK5是介导细胞胁迫响应MAPK级联反应途径的重要信号分子, 既可以响应非生物胁迫又可以响应生物胁迫, 并且呈现负相关功能[33]。OsSAPK2的表达变化可以引起上述几个胁迫应答基因的表达差异, 暗示该基因是水稻抗白叶枯病反应信号传导途径中的一个正调控因子。
此外, 我们利用酵母双杂交筛选到多个可能与OsSAPK2相互作用的蛋白, 这些蛋白可能被OsSAPK2翻译后修饰(磷酸化)或在接收信号等方面起作用。如OsEREBP1基因参与植物抗逆反应[34], OsEREBP1 (LOC_Os02g54160)属于AP2/ERF转录因子家族[35]。BWMK1通过结合和激活OsEREBP1调节病程相关蛋白的表达[36]。我们推测OsSAPK2可能通过与OsEREBP1相互作用, 启动下游病程相关蛋白, 增强水稻抗病性。下一步的实验重点是对候选蛋白进行验证, 深入研究OsSAPK2与互作蛋白的互作机制, 为进一步研究OsSAPK2参与调控水稻抗白叶枯病的分子机制提供信息。
水稻胁迫相关蛋白激酶OsSAPK2被定位于细胞核和细胞质内, 在OsSAPK2-RNAi转基因水稻中OsSAPK2下调表达, 接种水稻白叶枯病菌后, 转基因水稻比受体对照感病性显著提高, 抗病相关基因OsLRR1、OsHIR1表达水平下降, 感病相关基因OsMAPK5表达水平升高。推测OsSAPK2可能通过调节OsLRR1、OsHIR1、OsMAPK5的表达而调控水稻对水稻白叶病的抗病反应。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|