选用陆地棉抗枯萎病品种中棉所12和感枯萎病品种新陆早7号, 利用Solexa高通量测序技术分析棉花幼苗受到枯萎病菌侵染后3 h和6 h根部组织的基因表达谱。中棉所12受病菌侵染后3 h与0 h相比, 6 h与0 h相比以及6 h与3 h相比, 分别鉴定出4447、5481和2559个差异表达基因; 新陆早7号分别鉴定出8615、6727和2078个差异表达基因; 2个品种比较, 在3 h和6 h分别鉴定出1879个和500个差异表达基因。基因功能分析表明, 差异表达基因划分为生物学过程、细胞组分和分子功能注释本体, 并进一步细分为48个功能类别。代谢通路分析显示, 中棉所12和新陆早7号在枯萎病菌侵染后的6 h与0 h相比鉴定出的代谢通路(Pathway)最多, 均有126条; 在枯萎病菌侵染后6 h, 2个品种相比鉴定出的代谢通路最少, 仅有89条。各个比对组的代谢通路涉及的基因可被划分为次生代谢产物的生物合成、多糖的生物合成和代谢、环境适应和免疫系统等13类。在环境适应和免疫系统分类中存在着植物与病原菌相互作用代谢通路, 涉及到996个已知功能的差异表达基因, 有444个基因上调表达, 552个基因下调表达。其中, 差异表达基因最多的是WRKY转录因子家族基因, 其次为丝氨酸/苏氨酸蛋白激酶基因, 而DNA损伤修复/耐受性蛋白, JAZ1、RAR1和RPM1互作蛋白, S位点的特异性糖蛋白S6前体以及钙牵引蛋白等6类基因参与该代谢通路的数目最少。最后随机抽取6个基因进行实时荧光定量PCR验证, 其结果与测序结果一致。
Fusarium Wilt resistant cotton cultivar Zhongmiansuo 12 and susceptible cultivar Xinluzao 7 were used to analyze the gene expression profiling of root tissues at three hours and six hours after the seedlings infection by Fusarium Wilt using Solexa sequencing technology. Compared with no infection, 4447 and 5481 differential genes after three hours and six hours infected by Fusarium Wilt were identified, and compared six hours with three hours, 2559 differential genes were identified in Zhongmiansuo 12; while 8615, 6727, and 2078 was respectively identified in Xinluzao 7. There were 1879 and 500 differential genes in three hours and six hours after infection when the two cultivars were compared. According to the Gene Ontology, these genes were divided into these groups of biological process, cellular component and molecular function; then further subdivided into 48 functional categories. By analyzing the pathways, the most of them were identified in 6 h/0 h after infection between Zhongmiansuo 12 and Xinluzao 7, which were 126 each in the two cultivars; the least of pathways were at 6 h after infection in two cultivars, which was 89 only. However, all the pathways in each comparison group could be classified into 13 categories, such as biosynthesis of other secondary metabolites, glycan biosynthesis and metabolism, environmental adaptation, and immune system. A pathway, named plant-pathogen interaction in the environmental adaptation and immune system category, was involved in 996 differential genes; and the number of up regulated genes was 444 and that of down regulated genes was 552. The most differential genes were in WRKY transcription factor family, the serine/threonine kinase had the medium number of differential genes, while DNA damage-repair/toleration protein, JAZ1, RAR1, RPM1-interacting protein, S locus specific glycoprotein S6 precursor and caltractin had the least genes. Finally, six random genes were subjected to RT-PCR and the results validated the Solexa sequencing conclusions.
棉花枯萎病是一种危害严重的世界性病害。病菌侵染棉花后, 前期往往大量死苗, 后期叶片及蕾铃大量脱落, 甚至造成棉花植株的死亡, 严重影响棉花的品质及产量[1, 2]。在植物体内存在极其复杂和完善的自我防卫系统。当植物受到病原菌侵染, 植物抗病基因编码蛋白与病原无毒基因编码蛋白相互识别和作用, 激活信号传导途径, 导致下游一系列抗病防卫反应基因的启动, 产生快速的抗病代谢, 这一过程涉及大量相关基因表达量的改变。刘坤[3]利用qPCR技术分析棉花受到黄萎病菌侵染后, 29个ERF家族基因的表达变化, 发现除一个基因基本没有变化外, 其余基因均明显改变, 有的基因表达水平甚至增加7倍以上。韩庆典等[4]利用基因芯片技术对水稻细菌性条斑病感抗品种分别进行检测, 共检测到956个差异表达基因, 其中12个基因在2个品种中一致上调, 54个基因在2个品种中一致下调, 20个基因在2个品种中表达变化方向相反。此外, 一些研究发现, 植物受到病原菌侵染后, 同一基因在感抗品种中的表达存在明显差异。何兰兰等[5]发现ERF-B3亚组转录因子基因GhB301, 在受到枯萎病菌诱导后, 其表达量在抗病品种与感病品种中均明显增加, 但在抗病品种中表达量的增加显著高于感病品种; 在抗病品种中, 病菌处理后3 h表达量达到最大, 早于感病品种的6 h。
近年来, 人们在棉花抗病分子机制方面已做了大量研究工作, 并克隆了多个抗病相关基因。但由于陆地棉尚没有完成基因组测序, 对完整的转录组信息了解不足, 前人的研究多是对单个基因相对独立地分析, 难以获得系统的认识。Solexa测序技术通过对样品中数以百万计的mRNA标签进行序列测定, 捕获样品中低表达的基因, 克服了以往DNA微矩阵芯片等技术的不足, 在研究差异表达基因方面显示出强大的优势[6]。Wu等[7]通过Solexa高通量测序对葡萄霜霉病侵染和对照样品进行转录组测序, 发现了大量的差异表达基因, 并找到参与调控与核糖体结构、光合作用、氨基酸和糖代谢途径有关的基因, 初步揭示了葡萄霜霉病病程发展的调控机制。Bai等[8]利用Solexa测序技术研究感抗香蕉枯萎病品种在枯萎病菌诱导后不同时间的基因表达谱, 发现抗病品种对香蕉枯萎病的防御响应更快, 并且在代谢通路(Pathway)分析中发现了大量抗病相关基因, 初步揭示了香蕉抗枯萎病的抗病机制。目前, Solexa高通量测序技术在研究植物的逆境胁迫方面得到广泛应用[9, 10, 11, 12, 13, 14], 本研究利用Solexa高通量测序技术构建感、抗棉花品种在枯萎病菌诱导后不同时间的基因表达谱, 分析枯萎病菌侵染早期不同时间及不同感、抗品种间基因在转录组水平上的差异表达, 旨在为揭示棉花抗枯萎病的分子调控机制及筛选抗枯萎病相关基因奠定基础。
陆地棉抗病品种中棉所12和陆地棉感病品种新陆早7号由石河子大学棉花所提供。挑选经浓硫酸脱绒后的籽粒饱满的种子, 经0.1%氯化汞浸泡15 min消毒, 无菌水冲洗4~5遍, 种植于无菌蛭石, 待棉苗长至3 cm左右移入盛有Hoagland营养液的塑料盒(用带有孔洞的泡沫板漂浮棉苗), 于25℃、光照16 h、黑暗8 h的培养箱中继续培育, 每周换一次培养液[15]。
将生长于PDA培养基的枯萎病菌[Fusarium oxysporumf. sp. vasinfectum(Atk.) Snyder & Hansen]菌落(菌种为枯萎病7号生理小种的强致病力菌系F430, 由石河子大学植保系张莉老师提供)接种到装有已灭菌的查氏培养液中, 28℃振荡培养4 d, 配制成浓度为7× 106个mL-1的孢子悬浮液。将长至二叶一心的棉花幼苗根部浸泡到孢子悬浮液接种病菌, 处理45 min后转入Hoagland营养液中, 采集中棉所12和新陆早7号两品种接菌后0、3和6 h的棉花根部组织, 并分别标记为Z-0、Z-3、Z-6和X-0、X-3、X-6。液氮速冻后保存于-80℃冰箱。
采用改良的CTAB法[16]提取棉花根部组织RNA, 用ND-100 Nanopo spectrophotometer and Agilent 2100 Bioanalyzer检测RNA样品的质量。检测合格的RNA样品, 加热打开二级结构后用带有Oligo(dT)的磁珠富集mRNA, 向得到的mRNA中加入适量打断试剂, 高温条件下使其片段化, 再以片段后的mRNA为模板, 合成cDNA, 利用磁珠纯化双链cDNA。
对所得的双链cDNA经过末端修复、3° 末端加碱基A及加测序接头后, 进行PCR扩增富集测序样本, 从而完成整个文库制备。对构建好的文库用Agilent 2100 Bioanalyzer和ABI Step One Plus Real-time PCR System进行质量和产量检测, 合格后使用Illumina HiSeq 2000测序, 得到Raw reads。
将测序得到的Raw reads, 去除含有接头的序列、未知序列的比例大于10%的以及低质量的reads, 得到clean reads, 以备后续试验。再使用短reads比对软件SOAPalingner/soap 2将clean reads比对到最新雷蒙德氏棉参考基因序列及雷蒙德氏棉参考基因组序列, 最多允许2个碱基错配。对测序结果进行饱和度分析, 随机性分析及在参考基因组上的分布分析, 合格后运用RPKM法(Reads per kb per million reads)计算基因表达量[17]。
基于Audic和Claverie[18]描述的方法, 对表达谱中每个基因进行显著性分析。错配率(false discovery rate, FDR)则被用于确定P值的阈值[19]。符合P< 0.005, FDR≤ 0.001以及│log2 ratio│≥ 1的基因被定义为差异表达基因, 对所有差异表达基因进行GO功能注释及分类, 并利用KEGG (Kyoto Encyclopedia of Genes and Genomes)数据库定位分析Pathway。
从Solexa测序获得的差异表达基因中, 随机挑选6个差异表达明显的基因进行实时荧光定量PCR测定, 采用改良CTAB法提取总RNA。按照Fermentas公司生产的第一链cDNA合成试剂盒说明书上的步骤合成cDNA。依据数字化基因表达谱测序所得序列库对应的代表序列, 使用Primer premier 5.0软件设计实时荧光定量PCR引物, 以棉花的GhUBQ7 (DQ116441)基因作为内参基因(表1)。引物由北京六合华大基因科技有限公司合成, 所用染料为TaKaRa公司的SYBR, 实时荧光定量PCR的反应体系及程序参见TOYOBO公司的SYBR Green Realtime PCR Master Mix (QPK-212)说明书, 设3个重复, 采用2-∆ ∆ Ct法计算基因相对表达量。
| 表1 用于实时荧光定量PCR的引物 Table 1 Primers for real-time PCR |
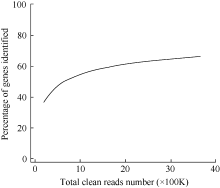
测序饱和度是用来衡量样品测序量的多少是否能够达到分析标准。当测序量(reads数量)增加时, 所能检测到的基因数量也随之增多, 但是当测序量达到某一阈值后, 检测到的基因数量增加的趋势趋于平缓, 证明检测到的基因趋于饱和。以Z-3为例(图1, 其他处理时间的测序饱和度趋势与之类似), 当测序样品的clean reads的数目达到2M时, 检测到的基因数目趋于饱和, 说明测序量已基本覆盖细胞中表达的全部基因, 可用于后续的研究分析。
经Solexa测序后, 对差异表达基因统计分析(表2)表明, 2个品种在枯萎病菌侵染后的3 h和6 h, 分别与对照相比, 差异表达基因中的下调基因均明显多于上调基因, 且感病品种的差异表达基因明显多于抗病品种。在中棉所12中, 与对照(Z-0)相比, 枯萎病菌侵染后3 h (Z-3)差异表达基因数目为4447个。其中上调基因数目为1300个, 下调基因数目为3147个。枯萎病菌处理后6 h (Z-6), 与对照相比, 差异表达基因数目为5481个。其中上调基因数目为1494个, 下调基因数目为3987个。而Z-6与Z-3相比, 差异表达基因的数目较少, 为2559个。其中上调基因数目为1020个, 下调基因数目为1539个。在新陆早7号中, 与对照(X-0)相比, 枯萎病菌侵染后3 h (X-3)的差异表达基因数目为8615个。上调基因数目为2118个, 下调基因数目为6497个。枯萎病菌处理后6 h (X-6)与X-0相比, 差异表达基因数目为6727个。上调基因数目为2038个, 下调基因数目为4689个。而X-6与X-3相比, 差异表达基因的数目为2078个。其中上调基因数目为1010个, 下调基因数目为1068个。
比较2个品种在枯萎病菌侵染同一时间点的差异表达基因(表2)表明, 下调基因数目依然多于上调基因, 但是差异表达基因的总数迅速下降。其中, 在受到枯萎病菌侵染后的3 h (Z-3 vs. X-3), 抗病品种相对于感病品种的差异表达基因数目为1879个。上调基因数目为874个, 下调基因数目为1005个。而在受到枯萎病菌侵染后的6 h (Z-6 vs. X-6), 抗病品种相对于感病品种的差异表达基因数目为500个。上调基因数目为314个, 下调基因数目为186个。
| 表2 差异表达基因 Table 2 Number of differentially expressed genes |
GO分析将104 610个差异表达基因分为3个功能注释本体, 分别描述基因的生物学过程、细胞组分和分子功能。中棉所12比对组Z-3 vs. Z-0、Z-6 vs. Z-0和Z-6 vs. Z-3分别有15 578、21 581和9683个, 而新陆早7号比对组分别有33 462、27 728和7377个unigene至少含有一种功能注释。2个品种相同处理时间的比对组Z-3 vs. X-3和Z-6 vs. X-6分别有7052个和1750个unigene至少含有一种功能注释。生物学过程、细胞组分和分子功能又可被进一步划分为48个类别(表3), 依次为23、14和11个类别。其中催化活性, 代谢过程和蛋白结合注释到的差异表达基因最多, 分别为12 397、12 158和11 678个, 而翻译调控活性类别最少, 仅有1个差异表达基因。
| 表3 差异表达基因的功能分类 Table 3 Functional categorization of the differentially expressed genes |
利用KEGG数据库代谢途径对差异表达基因进行功能注释和分类。通过Solexa测序, 中棉所12和新陆早7号比对组Z-6 vs. Z-0和X-6 vs. X-0鉴定出的Pathway最多, 均有126条, 且名称一致, 注释到的差异表达基因数目分别为5313个和6107个。经对比分析, 发现其余各个比对组的Pathway均少于126条, 且都在126条的基础上缺少一条或多条Pathway。Z-6 vs. X-6的Pathway数量最少, 仅有89条, 注释到的差异表达基因为456个; 缺少37条Pathway, 涉及到脂质代谢、能量代谢、氨基酸代谢、多糖的生物合成和代谢、碳水化合物代谢、辅助因子和维生素的代谢、萜类化合物和多酮类化合物代谢、次生代谢产物的生物合成以及遗传信息处理等9个方面。Z-6 vs. Z-3的Pathway有120条, 注释到的差异表达基因为2405个; 与126条相比缺少6条Pathway, 涉及到辅助因子和维生素的代谢, 氨基酸代谢, 脂质代谢以及遗传信息处理4个方面。X-6 vs. X-3的Pathway有118条, 注释到的差异表达基因为2282个; 与126条相比缺少8条Pathway, 涉及到辅助因子和维生素的代谢, 能量代谢, 次生代谢产物的生物合成以及遗传信息处理。Z-3 vs. X-3的Pathway有117条, 注释到的差异表达基因为2034个; 与126条相比缺少9条Pathway, 涉及到辅助因子和维生素的代谢, 次生代谢产物的生物合成以及遗传信息处理。此外, Z-3 vs. Z-0的Pathway有123条, 注释到的差异表达基因为4060个, X-3 vs. X-0的Pathway有125条, 注释到的差异表达基因为7281个。
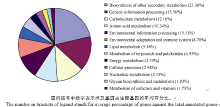
各比对组的Pathway总数不同, 注释到的差异表达基因数目不同, 但是除去无法具体分类的代谢途径Pathway (ko01100)后, 剩余的125条Pathway均可划分为13类别, 将它们在6个比对组中涉及基因占注释基因的平均百分比作图(图2)。次生代谢产物的生物合成涉及基因占注释基因的百分比最高, 平均值为21.36%; 遗传信息处理相关基因所占比例次之, 平均值为15.36%; 而辅助因子和维生素的代谢的相关基因所占比例最少, 其平均值仅为1.73%。除上述代谢相关基因外, 环境信息处理相关基因平均百分比为10.11%; 萜类化合物和多酮类化合物的代谢相关基因平均值仅为4.95%; 还有平均百分比为8.70%的基因参与环境适应和免疫系统; 而这几类Pathway可作为后续研究重点, 分析这些基因在感抗品种对枯萎病菌不同抗性表现中的作用。
在环境适应和免疫系统分类中存在着植物与病原菌相互作用Pathway (ko04626), 并且在Z-3 vs. Z-0; Z-6 vs. Z-0; Z-6 vs. Z-0; X-3 vs. X-0; X-6 vs. X-0; X-6 vs. X-3; Z-3 vs. X-3以及Z-6 vs. X-6比对组中分别涉及到232、290、159、421、323、133、113和34个差异表达基因(含重复), 去除未知、预测及未命名基因后统计汇总(表4)表明, 该Pathway中的996个差异表达基因有444个基因上调表达, 552个基因下调表达; 共涉及到61类差异表达基因, 其中差异表达基因最多的是WRKY转录因子家族基因, 共有162个(79个上调表达, 83个下调表达), 所占比例为16.3%; 其次为丝氨酸/苏氨酸蛋白激酶基因, 共有82个(38个上调表达, 44个下调表达), 所占比例为8.2%; 而DNA损伤修复/耐受性蛋白, JAZ1、RAR1、RPM1互作蛋白, S位点的特异性糖蛋白S6前体以及钙牵引蛋白等6类基因参与该Pathway的数目最少, 分别只有1个, 所占比例仅为0.1%。此外, 值得注意的是, 在这些差异表达基因中有38个与NBS- LRR类抗病蛋白相关, 25个与bHLH转录因子相关, 22个与bZIP转录因子相关, 22个与抗病相关蛋白相关, 7个与抗黄萎病蛋白相关。
 | 图2 13类代谢通路在6个比对组中涉及基因占注释基因的平均百分比Fig. 2 Percentage of genes in the total annotated genes of 13 pathways in six comparison groups |
| 表4 植物-病原菌相互作用Pathway中涉及的差异表达基因 Table 4 Differentially expressed genes of plant-pathogen interaction Pathway |
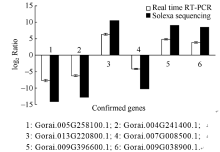
以中棉所12和新陆早7号的在枯萎病菌侵染后3 h和6 h的4个cDNA作为样本, 从Solexa测序中随机挑选的3个上调和3个下调的差异表达基因进行实时荧光定量PCR检测。并根据所得数据以2为底的对数值进行标准化处理后。结果如图3所示, 黑色柱形图为Solexa测序数据结果, 白色柱形图为实时荧光定量PCR验证数据结果。可以看出, 这6个基因相对表达量与Solexa测序的结果趋势是相同的, 说明基因表达谱测序获得的信息具有较高的可靠性。
近年来, Solexa测序技术的迅猛发展给生物基因组学的研究带来了深刻的影响, 为从整体水平研究作物的基因表达变化, 阐明作物对病原菌的抗性机制提供了有力的技术支持。本试验利用Solexa高通量测序技术建立了高抗枯萎病品种中棉所12和高感枯萎病品种新陆早7号在枯萎病菌侵染后3 h和6 h的棉花根部表达谱, 在受到枯萎病菌诱导后, 探索感病品种和抗病品种在转录组水平上的差异。尽管中棉所12和新陆早7号在遗传背景上存在着区别, 但在缺少近等基因系的情况下, 选用2个对枯萎病抗性存在明显差异的陆地棉品种作为研究材料, 利用Solexa测序技术研究不同侵染时间后基因的表达谱差异, 对进一步阐明棉花与枯萎病互作的分子调控机制仍具有指导意义。
通过对感抗品种差异表达基因分析表明, 与对照相比, 2个品种在枯萎病菌侵染后3 h和6 h, 许多基因的表达量发生改变; 其中下调基因的数目明显多于上调基因。这一结果与王刚等的研究类似[20, 21, 22]。此外, 感抗品种在枯萎病菌侵染后相同时间, 其差异表达基因中下调基因仍多于上调基因, 但差异表达基因总数迅速下降。这些差异表达基因是否与这2个品种对于枯萎病表现出不同抗性有关仍需要后续试验验证。另外, 在差异表达基因中有一部分是没有功能注释、没有报道的基因, 推测可能是棉花全基因组测序未完成, 可供参考的数据信息比较有限, 加之很多数据信息来自于棉花纤维发育, 抗病相关信息还非常缺乏, 但它们的重要性不可忽略。
通过GO功能分析发现6个比对组中的差异表达基因在3个注释本体中所占的比例大致相同, 即分子功能约占20%, 生物学过程约占40%, 细胞组分则约占30%。同时大部分基因富集在分子功能本体中的蛋白结合和催化活性, 细胞组分中的的细胞、细胞部分、细胞器上, 并主要参与细胞过程和代谢过程等生物学过程; 这与陈欢等[21]关于大豆籽粒不同发育时期表达谱研究的结果类似。仅从分子功能的转录因子活性类别中就找到了许多目前已证实与抗病相关的bZIP类、WRKY类、AP2/EREBP类、NAC类等以及研究不多的homeodomain和HSF等转录因子调控的基因[23], 这为后续抗病相关基因的挖掘工作指明方向。
Pathway中可以找到许多已证实与抗病相关的萜类、黄酮类, 生物碱类等小分子化合物的合成[24]。如萜类骨干生物合成(ko00900), 单萜的生物合成(ko00902), 倍半萜类和三萜类生物合成(ko00909), 黄酮类的生物合成(ko00941、ko00944等)以及多种生物碱的合成(ko00950、ko00960等)。此外, 已有研究表明, 植物内源激素在植物受到生物胁迫和非生物胁迫时发挥重要作用, 多种植物激素曾被报道参与调控植物对病原菌的抗性[25, 26], 而环境信息处理中恰好存在植物激素信号转导Pathway (ko04075), 其涉及到的差异表达基因也具有重要的研究意义。
在植物与病原菌相互作用Pathway中涉及到996个已知功能的差异表达基因, 其中WRKY、bHLH及bZIP三类转录因子家族分别有162、25和22个差异表达基因参与, 而已经证实它们与植物的抗病反应有关, 其可以与相应基因启动子中的顺式作用元件发生DNA-蛋白的特异性互作, 从而激活防卫反应基因的转录表达[23]。另有82个丝氨酸/苏氨酸蛋白激酶基因, 47个LRR受体丝氨酸/苏氨酸蛋白激酶基因, 38个蛋白激酶基因, 38个NBS-LRR类抗病蛋白, 13个受体丝氨酸-苏氨酸蛋白激酶基因, 11个LRR受体蛋白激酶基因以及3个蛋白磷酸酶基因参与该Pathway。大量研究表明, 蛋白激酶和蛋白磷酸酶对植物-病原菌互作反应起调节作用, 并且与抗病基因也存在联系[27]。Song等[28]曾克隆水稻抗白叶枯病基因Xa21, 其结构与拟南芥中的受体蛋白激酶类似, 属于丝氨酸/苏氨酸-蛋白激酶家族。蛋白质序列分析表明在其氨基端含有LRR区(leucine-rich region), 参与蛋白质相互作用, 表明LRR蛋白与蛋白激酶可以在植物抗病信号传递过程中相互作用, 参与植物的抗病反应[29]。还有22个与抗病相关蛋白基因, 7个抗黄萎病蛋白基因, 上述结果表明植物与病原菌相互作用Pathway中涉及到的差异表达基因具有重要的研究意义, 除重点研究单个基因对棉花枯萎病抗性的功能外, 更应该分析多个基因之间的相互作用以深入了解植物的抗病机制。同时剩余的大量未知、未命名基因也可作为后续研究的重点。
实时荧光定量PCR检测Solexa测序后6个基因的相对表达量, 发现二者上调或者下调的趋势是相同的, 但其结果并不完全一致, 主要由2个技术在检测灵敏性上的差异造成[30], 依然可说明Solexa测序获得的结果具有较高的可靠性。
本研究利用Solexa高通量测序技术对枯萎病菌侵染早期不同时间、不同感抗品种的基因的表达变化进行了全面、深入的检测, 无论是差异基因的筛选, GO功能分析以及Pathway分析等都表明感抗枯萎病品种在枯萎病菌诱导后抗性机制有差别, 而大量的差异表达基因也为筛选抗枯萎病相关基因提供丰富的数据资源, 为进一步研究棉花抗枯萎病的分子调控机制奠定了基础。
选用中棉所12和新陆早7号, 利用Solexa高通量测序技术分析棉花幼苗受到枯萎病菌侵染后3 h和6 h根部组织的基因表达谱, 从中筛选出大量差异表达基因。已注释的差异表达基因依据功能, 可划分为生物学过程、细胞组分和分子功能3个功能注释本体, 进一步细分为48个功能类别。代谢通路分析鉴定出从89条到126条不等的Pathway, 涉及到的基因可以划分为次生代谢产物的生物合成, 多糖的生物合成和代谢, 环境适应和免疫系统等13类。996个已知功能的差异表达基因参与植物与病原菌相互作用Pathway, 涉及差异表达基因最多的是WRKY转录因子家族基因, 其次为丝氨酸/苏氨酸蛋白激酶基因; 而DNA损伤修复/耐受性蛋白、JAZ1及钙牵引蛋白等6类基因参与该Pathway的数目最少。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|