第一作者联系方式: E-mail: 13899525177@163.com, Tel: 13899525177
枯萎病是危害海岛棉生产的重要因素之一, 研究枯萎病抗性分子机制将为培育抗病海岛棉品种、解决枯萎病对海岛棉的危害问题提供坚实的基础。本研究在前期转录组测序的基础上, 对海岛棉枯萎病抗性差异表达基因进行分析(Differentially Expressed Gene, DEG); 以7个抗病性表现不同的海岛棉品种为材料, 利用qRT-PCR方法研究抗病差异表达基因在接种0~40 h的表达量差异, 分析基因表达量与病情指数的相关性。结果表明, DEG分析得出类黄酮代谢通路相关基因与海岛棉枯萎病抗性有关。qRT-PCR分析显示抗病材料中类黄酮代谢通路关键基因的表达量显著高于感病材料。在接菌后多个时间点, 类黄酮代谢通路中的关键基因 TT7、 CHI和 DFR在抗病材料中的表达量显著或极显著高于感病材料, 其中 CHI和 DFR基因的表达量与病情指数呈显著负相关。综上所述, 类黄酮代谢通路相关基因对海岛棉枯萎病抗性均有影响, 且 CHI、 TT7和 DFR基因是关键基因。
Fusarium wilt is one of the important factors threatening the production of island cotton ( Gossypium barbadense L.). Understand the molecular mechanism of Fusarium wilt resistance will facilitate cotton breeding against Fusarium wilt and therefore alleviate the problem in cotton production. On the basis of RNA-seq results in previous studies, differentially expressed genes (DEG) were further analyzed in this study. After inoculating the pathogenic fungus for 0-40 hours, the DEGs expression levels of seven varieties in different wilt resistance were studied using qRT-PCR method. The correlation between gene expression level and disease index was analyzed. The flavonoid biosynthetic pathway genes were found to be related to Fusarium wilt resistance. The qRT-PCR result indicated the significantly higher expression levels of flavonoid metabolic pathway genes in resistant genotypes than those susceptible genotypes. The expression levels of TT7, CHI, and DFR, the key genes in flavonoid metabolic pathways, were significantly higher in resistant genotypes than in susceptible genotypes ( P < 0.05 or P < 0.01) at several time points after inoculation. Particularly, the expression levels of CHIand DFRwere negatively correlated with disease index. Therefore, we conclude that the flavonoid metabolic pathway related genes might be involved in the resistance to Fusarium wilt in cotton, among which CHI, TT7, and DFRare the key genes.
海岛棉(Gossypium barbadenseL.)在棉花栽培种中纤维品质最优[1], 枯萎病的浸染导致其产量和纤维品质降低[2]。研究表明在相同条件下对陆地棉(Gossypium hirsutum L.)和海岛棉接种枯萎病菌, 后者的感病程度较重[3]。陆地棉枯萎病抗性遗传与海岛棉不同, 校百才等[4]研究发现陆地棉的枯萎病抗性以加性效应为主, 而Smith等[5]认为海岛棉的抗性是由2个具有加性效应的显性基因共同决定。随着分子生物学技术的发展, 从分子水平上研究棉花的抗病机制已有诸多报道, Yang等[6]利用基因沉默技术证明GbRVD在海岛棉抗黄萎病过程中扮演重要角色。Gao等[7]研究发现GhNDR1和GhMKK2是影响棉花抗黄萎病性的关键基因。但目前关于海岛棉抗枯萎病性基因研究的报道仍然较少, 挖掘海岛棉抗病基因, 为海岛棉抗病育种提供依据十分关键。
近年来, 越来越多的研究者采用转录组测序(RNA-seq)挖掘功能基因。Xu等[9]以海岛棉抗病品种海7124为材料, 利用RNA-seq技术分析其接种黄萎病菌V991菌株后的转录组变化, 检测到3442个差异表达基因, 并认为木质素代谢通路相关基因在棉花抗病防御反应中扮演重要角色。王春晖[10]对陆地棉抗黄萎病自交系1017012及其接菌后6个时间段的RNA混样进行转录组测序, 基因差异表达分析发现接菌后上调表达基因59 856个, 下调表达基因51 976个, 128条代谢途径相关基因富集表达。
类黄酮化合物作为重要的次生代谢物, 广泛分布于维管植物[11]。研究表明其与植物抗病性密切相关。Malhotra等[12]和贾振华[13]发现类黄酮化合物槲皮素在番茄和拟南芥的抗病方面发挥积极作用。Anna等[14]研究证实, 25种类黄酮物质可抑制黄萎病菌丝的生长。关于类黄酮代谢途径结构基因与植物抗病性的关系已有研究: 转反义DFR基因杨树中抗病物质儿茶素(Catechin)含量明显降低[15], GbCHI蛋白能显著抑制大丽轮枝菌(Verticillium dahliae)孢子的萌发和菌丝的生长[16]。植物在遭受病原微生物浸染后CHI活性会增强, 说明由它调控的类黄酮代谢途径是植物抵抗病原菌的防卫反应之一[17]。虽然类黄酮代谢途径中个别基因对植物抗病的影响已有诸多研究, 但系统性总结并揭示海岛棉类黄酮代谢途径中各基因与海岛棉枯萎病抗性的关系还未见报道。本研究以7个抗病性表现不同的海岛棉品种为材料, 研究RNA-Seq富集到的6个类黄酮代谢途径相关基因及另外2个关键基因FLS和C4H在0 h和其他5个接菌时间点的表达量差异, 并对基因表达量与病情指数进行相关性分析, 以期探究类黄酮代谢途径相关基因影响海岛棉枯萎病抗性的关键时期和关键基因, 为培育海岛棉抗病品种提供依据。
选取海岛棉材料新海14 (母本, 感病), 06-146 (父本, 抗病)及其杂交后的F2:6RIL群体中的抗、感株系RIL10893 (超抗)、RIL10895 (高抗)、RIL10897 (感病)和RIL10796 (易感)共6份材料进行转录组测序。选取海岛棉抗病材料06-146、埃棉2号(抗病)、DJ-07-136 (抗病)、5917 (抗病)、新海14、海92-3 (感病)和埃及棉424 (感病)共7份材料进行相关基因表达分析(表1和表2)。
| 表1 转录组测序材料 Table 1 Variety list of RNA-seq |
| 表2 实时荧光定量材料 Table 2 Variety list of q-PCR |
选取饱满种子, 经75%乙醇冲洗3次, 双氧水浸泡4~5 h后, 放入ddH2O里浸泡催芽24 h左右至露白, 将种子放在铺有湿润滤纸的发芽盒中, 28℃避光恒温培养至芽长3 cm左右, 移至霍格兰氏(Hoagland’ s)营养液中, 在25℃, 光暗周期16 h/8 h的条件下培养。待第一片真叶完全展开时, 接种稀释至1× 106个mL-1的枯萎病菌(生理小种7号)。选取0 h及接菌后40 h的棉株下胚轴, 经液氮速冻处理, -80℃超低温冰箱保存备用。
从0 h及接菌后4、10、18、28和40 h的棉株下胚轴提取RNA进行转录组测序, 得到的Reads用Trinity软件进行无方向组装, 将短的Reads拼装成长的转录本, 用以鉴定转录本的亚型, 对unigene进行RPKM (Reads Per Kilobase per Million mapped reads, 每百万reads中来自某基因每千碱基长度的reads数)处理, 再用DEG进行差异分析(筛选阈值为Q< 0.005且|log2Fold Change|> 1)。使用DAVID (The Database for Annotation, Visualization and Intergrated Discovery)在线平台(https://dauid.ncifcrf.gov/)对这些基因进行功能聚类分析, 明确海岛棉在抗病过程中涉及的相关代谢通路中主要基因, 以及这些基因在不同品种和不同接菌时期的表达差异。
将得到的差异表达序列, 运用NCBI中的ORF finder和BlastX找到序列的保守区域, 利用Primer 5.0设计qRT-PCR引物(表3)。使用天根生化科技有限公司的试剂盒(RNA plant Plus Reagent), 按照产品说明书提取总RNA。参照Thermo Fisher Scientific科技公司RNA纯化试剂盒(Thermo Scientific Gene JET)的具体步骤对总RNA进行纯化, 利用Titertek-Berthold检测系统有限公司(Titertek-Berthold)的超微量分光光度计(Colibri)检测总RNA浓度和质量, RNA浓度大于80 ng μ L-1, 且A260/A280大于1.8, 采用赛默飞世尔(Thermo Fisher Scientific)公司逆转录试剂盒(RevertAid First strand cDNA Synthesis kit)反转录提取后的RNA, 用ddH2O稀释cDNA样品5倍后, 置-80℃备用。使用全式金生物技术(TransGen Biotech)有限公司qRT-PCR试剂盒(TransStart Green qPCR SuperMix), 通过美国ABI公司Prism 7500 Fast System, 以UBQ7为内参, 检测7个品种(3个感病品种, 4个抗病品种), 6个接菌时期(0、4、10、18、28和40 h)的基因表达量变化。每个反应重复3次。根据Ct值, 利用公式2-Δ Δ Ct计算基因的相对表达量。
2-Δ Δ Ct=2-[(Ct目的基因-Ct内参基因)处理组-(Ct目的基因-Ct内参基因)对照组]
| 表3 荧光定量引物 Table 3 Primers for qPCR |
于2014年和2015年4月中旬将材料种植于新疆阿拉尔市新开岭镇枯萎病重病田中, 2次重复。在每年10月5日左右, 选择连续5株棉株, 采用剖杆的方法考察海岛棉的枯萎病抗性, 参照五级分类法[18]判断棉花的发病级别并计算病情指数。
病情指数(DI) = ∑ 级数× 每级病株数/(调查总株数× 最高级值)× 100
转录组测序分析DEG数据得出两亲本差异表达基因755个, 抗病亲本上调表达的基因1185个。利用RIL群体中抗病材料RIL 10895和RIL 10893, 感病材料RIL 10897和RIL 10796作进一步分析(表4)。
| 表4 转录组测序中类黄酮代谢相关基因的表达 Table 4 Expression of related genes in flavonoids by RNA-seq |
接菌后, 类黄酮代谢途径中6个关键基因(CHS、CHI、LDOX、DFR、ANR和TT7)在抗病亲本及RIL高抗, 超抗材料中均显著上调表达(表2)。CHS基因在抗病亲本及RIL超抗材料中上调表达的倍数分别为15.81和23.90, 比感病亲本及RIL感病材料高出7倍和11倍。CHI-2在抗病亲本及RIL超抗材料中的表达量高出感病亲本及RIL感病材料6倍之多。DFR, ANR基因在接菌前后表达量差异倍数的高低同棉花的抗性相一致, DFR-1为RIL10893 (8.35) > RIL10895 (1.38) > RIL10897 (1.18) > RIL10796 (1.46), DFR-2为RIL10893 (9.12) > RIL10895 (3.23) > RIL10897 (1.74) > RIL10796 (1.25); ANR: RIL10893 (4.97) > RIL10895 (2.04) > RIL10897 (1.29) > RIL10796 (0.94)。暗示类黄酮代谢途径相关基因可能与海岛棉不同抗性表型的产生关系密切。
为进一步验证类黄酮代谢途径相关基因与海岛棉枯萎病抗性的关系, 选取7个抗性表现不同的资源材料对类黄酮代谢通路中的8个基因进一步研究表明在接菌后18 h、40 h抗病材料的ANR (花青素还原酶)表达量显著高于感病材料, 且在这2个时间点, 感病材料的变异系数大于抗病材料, 说明该基因在感病材料之间的表达量有差异(图1和表5)。除海92-3外, 另外2个感病材料在4、10和28 h、40 h的表达量均上调, 且4 h和10 h上升幅度明显大于28 h和40 h, 在40 h所有材料的表达量均上调, 但是抗病材料的表达量显著高于感病材料。推测该基因的表达量在感病材料中前期上调明显, 在抗病材料中后期上调明显。
| 表5 类黄酮代谢通路相关基因表达量在抗感材料之间的差异 Table 5 Expression level of flavonoid metabolism pathway related genes in resistant- and susceptible materials |
C4H (肉桂酸-4-羟化酶)(图1和表5), 在接菌后40 h, 在抗病材料中的表达量显著高于感病材料, 在感病材料中表达量的变异系数大于抗病材料, 说明在40 h感病材料之间的表达量也存在较大差异。感病材料中除了在4 h有上调表达外, 在其余接菌时间段均无明显上调。抗病材料除DJ-07-136外, 都在40 h拥有最大值。该基因在接菌后40 h抗病材料的瞬时表达量增加明显。
DFR (二氢黄酮醇还原酶)(图1和表5), 在接菌胁迫后, 在抗病材料中的表达量均显著或极显著高于感病材料。除10 h外, 抗病材料在各时期表达量的变异系数均小于感病材料, 说明该基因的表达量在感病材料之间也存在差异。在感病材料中, 该基因的表达量除埃及棉424在4 h有少量上调外, 其余均为下调。而在抗病材料中, 该基因的表达量在4 h均上调表达, 并且在10 h持续上升, 在18 h全部下调, 在28 h除06-146外其余无明显上调。该基因的表达量在抗病材料和感病材料中有显著差异。在抗病材料中除DJ-07-136外, 其余均有上升下降在上升的趋势, 且均在接菌后期(28 h和40 h)达到峰值, 推测该基因可能在海岛棉抗枯萎病中起着重要作用。
TT7 (类黄酮3’ 羟化酶)(图1和表5), 接菌胁迫后18、28和40 h在抗病材料中的表达量显著高于感病材料, 在28 h和40 h的变异系数小于感病材料, 说明在这2个时间点该基因的表达量在感病材料之间也存在较大差异。从图1可知, 除DJ-07-136外该基因的表达量在4~18 h均无明显上调。在28 h, 新海14 (感病)、06-146 (抗病)、DJ-07-136 (抗病)、埃棉2号(抗病)表达量有大幅上升, 但新海14 (感病)的表达量显著低于另外3种抗病材料, 且在这3个抗病材料中, 该基因的表达量在此时有最大值。该基因在感病材料中无明显变化, 在抗病材料中后期明显上调表达。
CHI (查尔酮异构酶)(图1和表5), 接菌胁迫后10、18、28和40 h, 在抗病材料中的表达量显著高于感病材料, 但其变异系数小于感病材料。说明在感病材料之间该基因的表达水平有差异。抗病材料DJ-07-136和5917的表达量在18 h拥有最大值, 抗病材料06-146, 埃棉2号的表达量在40 h拥有最大值。该基因在感病材料中无明显上调表达。推测该基因可能与海岛棉抗枯萎病有关。
CHS (查尔酮合成酶)(图1和表5), 在接菌后18 h和40 h抗病材料的表达量显著高于感病材料, 变异系数小于感病材料, 可能该基因的表达量在感病材料之间也存在差异。除06-146外, 该基因在抗病材料中4 h和10 h的表达量较18 h和28 h更加活跃。除埃棉2号外, 该基因在抗病材料中的表达量均有上升下降再上升的趋势, 且第1个表达峰值出现在10 h, 第2个表达峰值在40 h。
LDOX (花色素合成酶)(图1和表5), 在接菌后10、28和40 h抗病材料的表达量显著高于感病材料, 且在28 h和40 h抗病材料表达量的变异系数小于感病材料, 说明该基因在接菌后28 h和40 h, 感病品种之间的表达水平也有差异。该基因在抗病材料中4 h和10 h的表达量较18 h和28 h更加活跃。在抗病材料中该基因的表达量均有上升下降再上升的趋势, 第1个表达峰值在4 h和10 h, 第2个表达峰值在40 h。推测该基因在抗病材料中的表达趋势可能是一个达到峰值后逐渐降低又回升的过程, 在感病材料中没有这种趋势。
FLS (黄酮醇合成酶)(图1和表5), 在接菌后10、28和40 h抗病材料的表达量显著高于感病材料, 而其变异系数小于感病材料, 说明该基因的表达量在感病材料之间也存在差异。在接菌后该基因的表达量在感病材料中均有下调的趋势。除DJ-07-136外, 抗病材料相对表达量的变化均有上升下降, 再上升的趋势。
这8个基因在4个抗病材料中的表达量较感病材料上调更加明显, 尤其是在40 h。除4 h外, CHI基因在抗病材料中各个时期的表达量均显著高于感病材料。DFR基因在抗病材料中各时期的相对表达量均显著高于感病材料。TT7的基因表达量在感病材料中无明显变化, 在抗病材料中后期明显上调表达。ANR、C4H、DFR和ANS在抗病材料中4 h和10 h的相对表达量高于18 h和28 h。CHS、FLS、LDOX和DFR的表达量在抗病材料中均有上调后下调又回升的趋势, 但在感病材料中并无这种趋势。C4H、CHI和CHS基因在感病材料接菌后4 h的表达量有明显上调, 但是在4 h之后表达量上调不如抗病材料明显。
接菌40 h后, 8个基因在抗、感材料间均有极显著差异(表6)。在除接菌后4 h以外的时期, CHI基因的表达量在抗、感材料间均呈极显著差异。DFR基因的表达量在抗、感材料之间, 除在4 h和28 h有显著差异外, 在其他时期均有极显著差异。在抗、感材料之间, TT7基因的表达量在28 h和40 h有极显著差异。在方差分析中发现抗病材料内部以及感病材料内部基因表达量的变异系数较大, 但是仍然发现抗、感材料之间的基因表达量有显著差异, 说明抗、感材料之间的差异极显著。
| 表6 抗、感材料间基因表达量方差分析 Table 6 Variance analysis of gene expression between resistant and susceptible varieties |
从表6可看出, 尽管在转录组测序的DEG分析中未检测出C4H和FLS基因, 但是在接菌后40 h时8个基因的表达量在抗、感材料之间均差异极显著, 这说明类黄酮途径中的关键基因与海岛棉枯萎病抗性关系密切。从表6还可看出, 在接菌后多个时间点CHI和DFR基因的表达量在抗、感材料之间均差异显著。
将抗、感共7份材料2014— 2015年的病情指数与类黄酮代谢通路相关基因表达量的相关性分析(表7)表明, 在接菌40 h后, TT7, FLS基因的表达量与病情指数呈极显著负相关, CHI和DFR基因的表达量与病情指数呈显著负相关。接菌28 h后, CHI与LDOX基因的表达量与病情指数呈显著负相关。接菌10 h后DFR基因表达量与病情指数呈显著负相关。从表7可以看出CHI和DFR基因分别在2个时期都被检测出的基因表达量与病情指数之间显著负相关, 说明这2个基因可能在海岛棉枯萎病抗性中起着重要的作用。
| 表7 海岛棉病情指数与类黄酮代谢途径相关基因表达量的相关系数 Table 7 Correlation coefficient between flavonoid metabolism pathway genes and disease index of Gossypium barbadense L. |
因为棉花黄萎病菌孢子萌发并渗透植物表皮细胞的过程发生在真菌感染后的前12 h[19]。复杂的病菌感染反应以及信号的传导和交换通常发生在真菌感染12~40 h [20, 21]。所以本研究选取接菌后6个时间点(0、4、10、18、28和40 h)进行荧光定量分析。由于棉花在接菌40 h后会激发细胞凋亡等生理生化反应[22]。为了避免凋亡基因等影响对抗病基因的判断, 本研究不对40 h以后的材料进行分析。
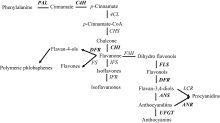
改良海岛棉抗枯萎病性是提高新疆海岛棉生产的根本途径。通过揭示海岛棉的抗病机制可以快速培育抗病品种。体外施加类黄酮化合物可显著提高植物的抗病性[23, 24]。研究者们已通过RNA-seq的方法证实类黄酮代谢相关基因与马铃薯、油棕和向日葵抗病有关[25, 26, 27]。前人对于棉花类黄酮的研究也主要集中在棉纤维颜色方面[28]。而对于类黄酮与海岛棉枯萎病抗性的关系研究甚少。Sun等[29]在对接种黄萎病菌前后陆地棉及海岛棉的转录组数据分析后指出, 在海岛棉中苯丙烷代谢途径相关基因呈上调表达趋势。苯丙烷代谢通路在棉花抵抗病原菌方面的积极作用已被许多研究所证实[11, 30, 31, 32]。类黄酮代谢途径(图2)[9]是苯丙烷代谢途径通过查尔酮合成酶(CHS)延伸的另一个重要分支[13]。本研究RNA-seq分析表明, 在接菌后40 h, 类黄酮代谢途径6个关键基因(CHS、CHI、LDOX、DFR、ANR和TT7)的表达量在抗感材料之间差异显著。其中CHS、CHI、LDOX和TT7基因在抗病亲本接菌前后表达量的差异倍数高达15.81、12.99、13.30和11.54。说明类黄酮代谢途径与海岛棉抗病性存在密切联系。
虽然研究者们通过多种方法证明类黄酮代谢途径与植物抗病有关, 且关键的转录因子以及调控基因也在多种植物中被克隆, 但由于类黄酮代谢途径比较复杂, 相关基因大多数为多基因家族。本研究在RNA-seq的基础上进行DEG分析, 检测到类黄酮代谢通路相关基因与海岛棉枯萎病抗性密切相关, 利用荧光定量的方法对不同抗性资源材料进一步分析表明, 接菌40 h后这些基因在抗病材料中的表达量显著高于感病材料。进一步证实类黄酮代谢通路对海岛棉抵抗枯萎病起积极的作用。
在除40 h外的其他时间点, CHI和DFR基因的表达量在抗、感材料间也存在显著或极显著差异, CHI和DFR基因在接菌后2个时间点的表达量与病情指数有显著或极显著负相关。其中CHI基因的表达量在接菌后的4个时间点均有极显著差异。CHI基因是类黄酮代谢途径中的一个关键酶, 在转基因烟草中CHI活性与总黄酮含量呈正相关[33]。推测在抗病材料中CHI基因受病菌诱导后表达量上调, 使黄酮含量增加, 而类黄酮化合物与相关酚类物质能够保护植物免受微生物浸染[34], 所以黄酮含量的增加可以导致棉花获得抗性。在接菌后各个时间点DFR基因的表达量在抗感材料之间均有显著或者极显著差异。根据马春雷等[35]的研究结果可知, DFR和LAR基因的表达量与茶树儿茶素质量分数呈一定的正相关。DFR基因的高表达可能会导致儿茶素含量增加。前人研究发现, 在枯萎病菌浸染后棉苗组织中儿茶素质量分数明显升高, 且在抗病品种棉苗组织中的儿茶素质量分数高于感病品种, 另外还发现棉花体内的儿茶素可抑制产孢及孢子的萌发[36]。儿茶素的增加可能会抑制枯萎病菌的菌丝生长。以上两基因CHI后续可通过沉默该基因后测定黄酮含量来探究该基因的抗病机制。后续可以结合儿茶素含量测定或者将正反义DFR基因转入棉花进一步研究DFR基因的功能。
Shih等[37]在研究高粱的抗病机制时认为, TT7基因在接菌后24 h表达量开始上调, 在接菌后72 h表达量达到最大值。本研究与其类似的是TT7基因的表达量均在接菌后期上调明显。另外研究结果还指出在接菌后期, TT7基因在抗病材料中的表达量显著高于感病材料。这个表达量差异说明其与抗病密切相关。转TT7烟草与正常烟草相比苯丙氨酸解氨酶(phenylalanineammonialyase, PAL)和过氧化物酶(peroxidase, POD)活性增加, 可减轻病原微生物对烟草自身产生的伤害[38]。另外, TT7基因的过量表达可以导致具有抗氧化和抗菌活性的类黄酮化合物槲皮素(Quercetin)的含量增加[23, 24]。因此推测在抗病材料中TT7的高表达可能导致PAL和 POD活性增加或者槲皮素含量增加, 从而提高海岛棉枯萎病抗性。
关于类黄酮代谢途径相关基因与植物抗病性的关系, 前人的研究主要集中在陆地棉中。Deshika等[39]研究证明棉花在接种枯萎病菌后24 h CHS基因的表达量与对照并无差异。本研究证实CHS基因在接菌后40 h表达量上调明显, 而在28 h大多数品种并无明显上调表达。该基因在海岛棉和陆地棉中的差异表达暗示海岛棉抗枯萎病机制不同。Xu等[9]以接种黄萎病菌前后的海7124为材料进行转录组测序, 并利用qPCR的方法对差异表达基因进行分析, 发现抗病材料中C4H基因和CHS基因在接菌后各个时期(4、12、24和48 h)的表达量均高于感病材料, 且CHS基因的表达量要明显高于C4H基因的表达量。另外Xu等[9]还发现C4H和CHS基因的表达量均在在接菌后24 h达峰值, 在48 h表达量有少量下降。而本研究中这2个基因的表达量都在40 h有最大值。在接种黄萎病菌和枯萎病病菌后C4H和CHS基因的差异表达说明枯萎病与黄萎病的抗性机制也存在差异。
抗病材料中类黄酮代谢通路相关基因的表达量显著高于感病材料。在接菌后40 h, 类黄酮代谢途径8个基因在抗病材料中的表达量均显著高于感病材料。其中CHI、DFR和TT7在抗病材料中其他时间点的表达量也显著高于感病材料。在接菌后的两个时间点, CHI和DFR基因的表达量与病情指数呈显著负相关。在接菌后40 h, TT7基因的表达量与病情指数呈极显著负相关。推测CHI、DFR和TT7可能与海岛棉抗枯萎病有关。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|