利用与大豆产量、品质、抗逆性、适应性等重要性状相关的125对SSR标记对中国大豆主产区东北和黄淮海地区自20世纪40年代以来大面积种植的89个大豆品种进行遗传多样性分析。结果表明, 自北向南大面积种植品种SSR标记的多态性呈逐渐升高的趋势, 黑龙江北部、黑龙江中南部、吉林辽宁地区和黄淮海地区大面积种植品种标记的多态性信息含量(PIC)依次为0.414、0.469、0.522和0.562。除黑龙江北部以外, 其余3个地区20世纪80年代以来大面积种植品种的多态性信息含量均比80年代以前种植的品种高。根据Nei和Li提出的遗传相似系数对供试品种进行聚类分析, 发现除黄淮海北部地区的冀豆12外, 东北和黄淮海地区大面积种植品种各自聚成一类, 而在东北地区品种中, 吉林辽宁地区和黑龙江地区的品种归属不同亚类, 表明同一区域内大面积种植品种的同质化现象相当明显。
The genetic diversity of widely-planted soybean varieties released after 1940s in Northeast China and Yellow-Huai-Hai Rivers Valley (YHH)were analyzed by 125 simple sequence repeats (SSR) markers, which were related to the important traits including yield, quality, stress tolerance and adaptability. The results showed that the average polymorphism information content (PIC) from north part of Heilongjiang province, south and middle parts of Heilongjiang province, Jilin and Liaoning provinces and Yellow-Huai-Hai Rivers Valley were 0.414, 0.469, 0.522, and 0.562, respectively. Except for the varieties from the north part of Heilongjiang province, the varieties released after 1980 in the other three regions had higher PIC than those released before 1980. Based on similarity proposed by Nei and Li, we classified the tested soybean varieties into three major groups by the cluster analysis, corresponding with the original places of the varieties. The varieties from the Northeast and YHH were classified into two major groups respectively, except for Jidou 12, a variety from northern YHH, which was classified into an independent group. In the Northeast, varieties of Heilongjiang were classified into one subgroup and those from Jilin and Liaoning provinces another subgroup, indicating that the widely-planted varieties from the same region were apparently homogenized.
1913年, 中国在世界上率先设立大豆育种项目, 开展大豆遗传研究和品种选育。1923年, 最早的大豆育成品种金大332和黄宝珠问世[1], 至2005年全国累计育成大豆品种1300多个[2]。近年来, 我国大豆品种选育的进度有所加快, 仅2011年就育成品种111个[3], 2012年为98个[4], 估计至2012年底, 通过国家和省级审定的品种总数在1800个以上(含审定制度建立前已推广的品种)。在过去的100年中, 中国大豆品种改良的主要趋势是生育日数更加趋向合理、丰产性不断提高、抗病虫能力增强、化学品质不断改善[5]。从具体性状看, 生育期趋向缩短, 株高降低, 抗倒伏能力明显改善, 分枝数和单株节数减少, 单株荚数增多, 单株粒重提高, 单位面积产量以每年1.5%~2.0%的速度递增[6], 抗病虫和耐逆能力逐步增强, 脂肪和蛋白质含量略有上升[7]。在我国育成的1800个以上大豆品种中, 只有一小部分在生产上得到大面积推广利用, 其中在东北和黄淮海大豆主产区年种植面积达到或超过6.7万公顷的品种有100多个, 占两地区育成品种的10%左右, 这些品种是我国不同时期育种成就的优秀代表。这些大面积种植品种不仅在生产上发挥了重要作用, 而且因其具有综合性状优良、适应性好等特点, 成为育种的骨干亲本[8]。因此, 对这些大面积种植品种在分子水平上的演变趋势和遗传多样性的变化进行分析, 对今后的大豆品种改良工作具有一定的借鉴意义。
在育种过程中, 对农艺、产量、适应性和抗病性等目标性状的选择必然会导致相关基因区域分子标记多态性的改变。因此, 利用与重要育种目标性状连锁的分子标记的多态性信息, 可从基因组水平上揭示我国不同大豆生态区育种选择的倾向性和遗传改良的进度。SSR是目前在大豆上应用最广的分子标记, 已在遗传图谱构建、分子标记辅助选择、遗传多样性及遗传距离分析和品种鉴别等方面得到应用[9]。Li等[10]利用99个SSR标记和554个SNP分析中国303个栽培大豆品种和野生资源, 发现SSR位点的平均等位基因变异数为21.5, Nei’s基因多样性指数为0.77, 而SNP的遗传多样性指数只有0.35。Iquira等[11]利用39个SSR标记分析加拿大100个本地品种和200个外来种质, 发现外来种质比本地材料具有更高的遗传多样性。Mikel等[12]根据系谱关系分析1970—2008年间北美登记的2242个大豆品种, 所得遗传多样性指数为0.89。
目前, 国内外尚缺少对大面积种植大豆品种遗传多样性演变趋势系统评价的报道。本研究利用与大豆产量、品质、抗逆性和适应性等重要性状相关的SSR标记, 分析20世纪40年代以来在我国东北和黄淮海地区大面积种植的大豆品种遗传多样性, 以期揭示不同年代、不同地区大面积种植品种遗传多样性地理分布特点, 为大豆育种中亲本选配和后代选择提供依据。
根据种植面积选取东北和黄淮海地区自20世纪40年代以来在生产上大面积种植的大豆品种共89份, 其中东北地区选育的品种64份, 包括黑龙江省40份, 吉林省15份, 辽宁省9份; 黄淮海地区品种25份, 其中北京市3份, 河北省4份, 山东省12份, 河南省5份, 湖北省1份(在黄淮海地区种植) (表1)。
取苗期大豆幼嫩叶片, 参考Doyle和Doyle[13]的CTAB法提取总DNA, 用1%的琼脂糖凝胶电泳检测DNA质量, 用微量可见分光光度计SMA3000测定DNA浓度, 稀释为工作浓度25 ng µL-1。
选取已发表的或在SoyBase (http://www.soybase.org/)网站登录的与产量及其构成因子[14,15,16]、株高和抗倒伏能力等植株形态性状[17,18,19]以及与植株开花、成熟[20,21]、品质[22,23,24]、抗逆性和适应性等性状相关的150个SSR标记来分析所选取大面积种植品种的遗传多样性。SSR标记的引物序列来源于SoyBase大豆数据库, 由生工生物工程(上海)股份有限公司合成。
PCR反应在Mastercycler Gradient PCR仪上进行。20 µL PCR体系, 包含50 ng基因组DNA、1×PCR缓冲液、0.75 U Taq DNA聚合酶、0.2 mmol L-1 dNTPs、0.15 μmol L-1引物。PCR 程序为94℃ 5 min; 94℃ 30 s, 47℃ 30 s, 72℃ 30 s, 35个循环; 72℃ 5 min; 4℃保存。PCR扩增产物经6%变性聚丙烯酰胺凝胶上电泳分离, 硝酸银染色。
每对SSR引物检测一个位点, 将所得每条多态性带记为1个等位变异, 根据每个SSR标记检测出的等位变异的有无, 分别以0、1记载各品种的SSR等位变异, 在相同迁移率位置上, 有带记为“1”, 无带记为“0”, 缺失记为“9”。 计算每个SSR引物扩增的等位变异的多态性信息含量(PIC),
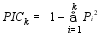
其中 Pi 为第 i 个等位基因在 k 位点上的群体的比例。用NTSYS2.1软件计算群体种质间Nei和Li遗传相似系数[23], 根据所得相似系数, 用非加权配对算术平均法(unweighted pair group method using arithmetic average, UPGMA)[26]绘制树状聚类图, 进行聚类分析。
| 表1 供试品种名称、来源及育成年份 Table 1 Variety name, geographic origin, and release year |
利用150对SSR引物对89份材料的基因组进行扩增, 其中125对引物得到清晰多态性条带, 可用于多态性分析。125对SSR引物共检测出685个等位变异, 平均等位变异数为5.48个, 等位变异的变化范围为1~14个, 以Satt373等位变异数最多(14个), Satt006最少(1个)(表2)
| 表2 部分SSR引物在89个大豆品种中的等位变异情况及PIC值 Table 2 Allele numbers of SSR primers in 89 soybean varieties and their PIC |
在东北地区64份大豆材料中共检测出527个等位变异, 平均等位变异数为4.22个; 在黄淮海地区25份材料中共检测出543个等位变异(表3), 平均等位变异数为4.31个。在这些等位变异中, 有118个为东北地区品种所特有, 其中黑龙江北部品种有5个特有等位变异, 黑龙江中南部品种中有29个特有等位变异, 吉林辽宁地区品种有84个特有等位变异; 黄淮海地区品种有125个特有等位变异。
在20世纪黑龙江北部80年代以前育成的12个大面积种植品种共有293个等位变异, 平均等位变异数为2.34个, 位点多态性信息含量(PIC)变幅为0~0.898, 平均为0.400; 80年代以后育成的13个大面积种植品种共有279个等位变异, 平均等位变异数为2.23个, 位点多态性信息含量(PIC)变幅为0~0.830, 平均为0.340。黑龙江中南部80年代以前的12个大面积种植品种共有333个等位变异, 平均等位变异数为2.660个, 位点多态性信息含量(PIC)变幅为0~0.810, 平均为0.386; 80年代后育成的13个大面积种植品种共有337个等位变异, 平均等位变异数为2.700个, 位点多态性信息含量(PIC)变幅为0~0.847, 平均为0.434。吉林、辽宁地区80年代以前的12个大面积种植品种共有349个等位变异, 平均等位变异数为2.79个, 位点多态性信息含量(PIC)变幅为0~0.833, 平均为0.433; 80年代后育成的13个大面积种植品种共有381个等位变异, 平均等位变异数为3.05个, 位点多态性信息含量(PIC)变幅为0~0.931, 平均为0.474。黄淮海地区80年代以前12个大面积种植品种共有418个等位变异, 平均等位变异数为3.32个, 位点多态性信息含量(PIC)变幅为0~0.875, 平均为0.505; 80年代后育成的13个大面积种植品种共有457个等位变异, 平均等位变异数为3.66个, 位点多态性信息含量(PIC)变幅为0~0.840, 平均为0.533。
| 表3 不同地区大面积种植大豆品种的等位变异 Table 3 Allele of widely-planted soybean varieties from different regions of China |
从以上结果可以看出, 除黑龙江北部外, 其余3个地区80年代后育成的大面积种植品种的平均等位变异数和多态性信息含量均比80年代以前有所提高, 说明这些地区后来育成的大面积种植品种的遗传多样性更加丰富。
20世纪40年代以来, 东北和黄淮海地区大面积种植品种的相似系数在0.244~0.863之间, 其中, 豫豆2号和吉林6号之间的相似系数最小, 集体1号和吉林6号之间的相似系数最大。89个品种在相似系数为0.36时被分为3大类(图1), 东北地区全部品种和黄淮海地区绝大多数品种分属I、II两大类, 黄淮海北部品种冀豆12(序号85)被单独分为一类(III)。第I大类包括所有的东北地区大面积种植品种和2个黄淮海北部地区品种(冀豆7号和鲁豆11)。在相似系数为0.44时, 东北地区品种主要可以分为2个亚类。第一亚类主要为来自吉林辽宁地区的品种和黄淮海北部品种冀豆7号和鲁豆11, 这2个黄淮海品种具有东北地区重要亲本丰地黄和熊岳小黄豆的血缘; 第二亚类主要为来自黑龙江地区的品种和来自吉林辽宁地区的品种九农22和铁丰31。第II大类22个品种全部来自黄淮海地区。在相似系数为0.402时, 第II大类又可以进一步划分为3个亚类, 第一亚类包括爬蔓青和菏泽牛毛黄2个农家品种; 第二亚类包括益都平顶黄、丰收黄、齐黄10号、莒选23、河南早丰1号和文丰7号等, 主要是益都平顶黄和莒选23选育出的后代; 第三亚类包括中豆19、诱变30、科丰6号、中黄13、豫豆2号、豫豆22和邯豆5号等品种, 它们都具有共同的祖先亲本滨海大白花和铜山天鹅蛋。聚类与系谱分析的结果一致, 进一步证明用SSR标记进行聚类分析的可靠性。
东北地区品种之间相似系数平均值为0.458, 其中黑龙江北部品种0.560, 黑龙江中南部品种0.529, 吉林辽宁地区品种为0.495。黄淮海地区品种之间的相似系数平均值为0.417, 黄淮海地区品种与东北地区品种之间的相似系数平均值为0.357。由此可见, 黑龙江北部大面积种植品种之间相似系数最高, 其他依次为黑龙江中南部、吉林辽宁地区和黄淮海地区, 与通过PIC值得到的各地区品种遗传多样性大小相吻合。黑龙江北部地区品种遗传多样性最低的原因可能与该地区育种起步较晚且基本采用本省品种资源做亲本选育品种有关, 而其他3个地区在品种培育过程中均重视选用异地资源做亲本, 品种遗传多样性水平较高。
研究表明, 黑龙江北部、黑龙江中南部、吉林辽宁地区和黄淮海地区大面积种植品种SSR标记的平均多态性信息含量(PIC)依次为0.414、0.469、0.522和0.562, 与通过相似系数计算的结果相符, 说明黄淮海地区大面积种植品种的遗传多样性最高, 这也与我们先前的系谱分析结果一致[8]。黄淮海地区大豆品种多态性信息含量高与该地区大豆的遗传资源比较丰富、品种的祖先亲本较多有关。吉林辽宁地区大面积种植品种遗传多态性居第二, 其次是黑龙江省。黑龙江北部地区20世纪80年代后育成的大面积种植品种的遗传多样性要低于中南部, 且低于20世纪80年代前, 该地区大豆品种遗传多样性降低的趋势值得大豆育种工作者注意。
 | 图1 89个大面积种植品种基于SSR标记的聚类图数字代表的品种名称见表1。The number of variety name corresponds with that given in Table 1.Fig. 1 Cluster analysis of 89 widely-planted varieties based on SSR markers |
栾维江等[27]曾对283份东北大豆进行SSR检测, 认为吉林和辽宁地区品种的遗传多样性高于黑龙江品种, 与本研究结果一致。赵洪锟等[28]对中国不同纬度大豆资源SSR分析发现无论是野生大豆还是栽培大豆, 随地理纬度的增加其遗传距离都有递减的趋势, 表明北方大豆多样性低于南方。20世纪80年代以来, 新育成品种比老品种遗传多样性高(黑龙江北部除外), 这与育种方法和技术的改进是相符的。盖钧镒等[29]认为新近育成的品种比老品种涉及更多的祖先亲本, 遗传基础有所拓宽。
高运来等[30]利用43对SSR引物分析83个黑龙江省主栽的大豆品种, 发现引物的平均多样性指数为0.55, 高于本文中同地区品种多样性指数的高值0.469, 说明大面积种植品种比普通品种的遗传多样性稍低, 可能原因是大面积种植品种必须保留共有的优良性状和基因。此外, 本文选用的是与主要农艺和产量性状相关的SSR标记, 在育种过程中对上述目标性状的选择可能致使其PIC降低。Wang等[31]利用60对SSR标记分析14个在农艺和形态性状上有差异的中国栽培大豆品种的遗传多样性, 得到的多态性信息含量(PIC)为0.50~0.92, 平均为0.78。Wang等[32]利用40对SSR标记分析40份山西大豆育成品种、地方品种和野生资源, 共得到262个等位变异, 平均每个SSR引物的等位变异数为6.55, PIC为0.585~0.850, 平均为0.78, 发现野生大豆、地方品种和育成品种的遗传多样性指数分别为1.5421、1.2864和1.0981。上述两项研究所得PIC数值均高于本研究中的PIC高值0.562, 由此看出大豆在驯化和育种过程中遗传多样性逐渐降低, 地方品种和普通育成品种遗传多样性低于野生大豆, 而大面积种植品种的遗传多样性又低于普通育成品种, 同质化趋势相当明显, 这一方面说明大面积种植品种含有许多共有的优良基因, 是优良基因的聚合体, 另一方面也说明大面积种植品种的遗传基础仍有待拓宽。
Diwan等[33]的研究结果显示, 在北美大豆品种的35个祖先亲本中, 平均每个SSR引物的等位变异数为10.1, PIC为0.64~0.91, 平均为0.8。Chen等[34]分析122份抗大豆胞囊线虫(SCN)资源, 发现每个SSR位点的平均等位变异数为6.70, PIC在0.02~0.50之间变化, 平均为0.35。Hwang等[35]分析包括优良品种在内的87个日本野生大豆种质和栽培品种, 发现377个SSR位点的平均等位变异数为3.7个, PIC均值为0.44。这些研究结果与我们的结果类似, 说明无论在国内还是国外, 育成品种的遗传多样性相近, 而祖先亲本的遗传多样性则丰富得多。
大面积种植品种是众多育成品种的优秀代表, 经过多年多点环境条件的考验, 与普通育成品种相比, 它们稳产性更好、适应性更广泛, 因而得到种植者和育种家的认可。东北和黄淮海地区是我国大豆主产区, 产量占全国的3/4以上, 对这2个地区不同年代大面积种植品种的遗传多样性分析, 能反映我国大豆育成品种在分子水平上的变化趋势, 对于今后的大豆育种工作具有一定指导意义。本研究结果表明, 尽管新近育成的大豆品种遗传多样性有所提高, 但大面积种植品种与普通育成品种相比其遗传多样性仍相对较低。这就要求我们在今后的品种选育中多选用地理远缘、遗传关系较远、含有不同优异等位变异的材料做亲本, 拓宽育成品种的遗传基础, 提高其适应能力, 避免育成品种遗传构成的同质化。黄淮海流域是我国南北大豆产区的交汇地带, 无霜期较长, 品种资源丰富, 是不同来源大豆品种相互组配的理想区域。此外, 随着生育期相关基因的克隆[36,37,38,39,40,41], 通过生物技术手段可改变现有大面积种植品种的开花期和成熟期, 从而拓展其适应区域, 使其在异地种植或做亲本材料, 提高其利用价值。
中国东北和黄淮海地区过去百年来大面积种植的大豆品种在重要性状相关SSR标记的遗传多样性方面表现出明显的地域特征, 自北向南多态性逐渐升高。同一地区不同年代品种的多态性信息含量(PIC)也有区别。黑龙江中南部、吉林辽宁地区和黄淮海地区20世纪80年代以来大面积种植品种的多态性信息含量均比80年代以前种植的品种高, 但黑龙江北部品种的变化趋势相反。同一区域内大面积种植品种的同质化现象相当明显, 育种中应注意利用地理远缘、遗传关系较远、含有不同优异等位变异的材料做亲本。
致谢: 中国农业科学院作物科学研究所常汝镇研究员和黑龙江省农业科学院刘忠堂研究员对品种选择提出宝贵意见, 邱丽娟研究员和刘章雄副研究员提供部分供试品种。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|


