利用分布于小麦全基因组的181对分子标记, 分析264份自然群体的基因型, 采用TASSLE软件的GLM和MLM模型检测与整穗发芽抗性紧密关联的标记位点, 发掘相关位点内的优异等位变异。在2012年和2013年室内整穗发芽率、2013年田间自然降雨整穗发芽率3个环境中, 共关联到20个显著位点(
To improve pre-harvest sprouting (PHS) resistance in wheat breeding, it is important to explore marker loci and elite alleles associated with PHS resistance using intact spikes. In this study, a total of 181 markers were used to 264 genotype materials. General and mixed linear models (GLM and MLM) were used to analyze PHS phenotypic data in three environments (2012-in house, 2013-in house and 2013-in field). The results showed that twenty markers were identified by association analysis, and located on chromosomes 1AS, 2DS, 3AS, 3BL, 4AL, 5AS, 5BL, 6BS, 6DS, 7AL, and 7BL. The markers
小麦收获前遇连阴雨天气或处于湿热环境时间较长, 易发生穗发芽(pre-harvest sprouting, PHS)。穗发芽导致种子内部储藏物质消耗, 千粒重下降和减产, 同时营养和加工品质劣化[ 1]。采用准确、可靠的评价方法筛选抗穗发芽资源, 从中挖掘抗性基因, 开发功能标记, 最终培育抗穗发芽品种是解决这一问题的根本途径。
小麦收获前穗发芽抗性受多种因素共同影响, 如种皮颜色、种子的休眠特性、小麦的穗部形态(有芒或无芒等)、颖壳内发芽抑制物质、籽粒吸水特性、α-淀粉酶活性、脱落酸(ABA)和赤霉素(GA)等因素, 以及外界环境因素如光照、温度和湿度等, 其中种子的休眠性被认为是穗发芽抗性的主要遗传因素[ 2, 3, 4, 5]。目前, 对小麦穗发芽抗性的评价主要有籽粒发芽法、整穗发芽法和α-淀粉酶活性测定法等。籽粒发芽法仅能反映小麦种子本身的休眠状况, 却忽略了颖壳等外部抑制物质的作用。α-淀粉酶活性测定法操作相对较复杂, 不适于批量测定。相对于前两者, 整穗发芽法操作简单, 适合较多试验材料的批量测定, 且更能全面衡量小麦穗子的整体抗性水平, 主要有发芽纸保湿法、塑料袋保湿法、纱布保湿法、模拟降雨法等[ 6]。
通过解析小麦穗发芽抗性分子机制, 发现小麦穗发芽抗性受多基因控制, 存在主效基因。利用DH或RIL等人工作图群体, 控制穗发芽抗性的主效基因被定位在2AL ( Xwmc1045和 Xgwm296), 3AS ( Xfbb370、 Xbarc310、 Xbcd907、 Xbarc57、 Xbarc321、 Xbarc12和 Xbarc310), 3AL ( Xgwm155和 Xwmc153), 4AL ( Xbarc170、 Xgwm397、 Xgwm269、 Xgwm937、 Xgwm894、 Xgwm637、 Xzxq118和 Xhbe03), 5D ( Xcfd40和 Xbarc1097), 6BS ( Xwmc104)及7DL ( XMST101)上[ 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21]。而基于自然群体的关联分析是定位和挖掘数量性状位点的另一条有效途径。利用这一方法, Arif等[ 22]检测出34个与小麦整穗发芽性状相关的位点, 分别位于1A、2A、2D、4A、5B、5D、6A、6B、7A和7D上, 其中2D、5B和7A是携带穗发芽抗性位点的密集区域; Kulwal等[ 23]除发现位于1BS、2BS、2BL、2DS、4AL、6DL和7DS上已报道的QTL外, 还鉴定出位于7BS的新位点。
本试验采用发芽纸保湿法对264份小麦材料进行整穗发芽抗性测定, 采用6%变性聚丙烯酰胺凝胶电泳对其进行基因型分析, 利用TASSLE软件的一般线性模型(GLM)和混合线性模型(MLM)对基因型和表型值进行关联分析, 检测与整穗发芽抗性紧密关联的分子标记位点, 并进一步挖掘优异等位变异及其相应载体材料, 为小麦抗穗发芽分子育种提供参考。
本课题组前期收集并保存410份育种亲本材料, 利用均匀分布于小麦21条染色体上的80对多态性丰富的SSR引物对其进行遗传多样性分析, 从中筛选出亲缘关系相对较远, 且穗发芽抗性不同的264份材料作为本试验材料, 包括推广品种204份、国外引进品种4份、地方品种18份、中间品系和高代材料38份。2011年和2012年, 所有材料均秋播于安徽农业大学合肥大杨店试验站, 每份材料种2行, 行长2 m, 行宽20 cm, 每行40粒, 常规田间管理。
采用整穗发芽率(germination percentage, GP)来评价穗发芽抗性水平, 分为抗穗发芽、中间型和感穗发芽3个抗性等级, 其整穗发芽率分别为0~30%、31%~60%和61%~100%[ 6]。
2012年和2013年分别进行室内鉴定。于小麦蜡熟后期(茎秆、叶片和麦穗全部变黄)收获每份材料10个主茎穗, 5穗为一个重复, 室内风干1 d, 立即存放于-20℃冰箱以维持种子的休眠性, 待收获全部材料后一并进行整穗发芽试验。先把整穗在灭菌水中充分浸泡10 min, 用发芽纸卷裹, 保鲜袋保湿, 置人工气候培养箱中(温度20℃, 光周期14 h昼/10 h夜, 相对湿度80%)培养7 d。于第7天取出并除去保鲜袋, 但保留发芽纸, 于电热恒温干燥箱(150℃)快速烘干, 阻止其继续发芽; 再于阳光下暴晒至籽粒易剥取; 记录每个穗子穗发芽率后取两重复平均值, 以胚部出现中破裂迹象为鉴定标准[ 1]。
2013年小麦收获期遇到连阴雨天气, 于田间进行了重复鉴定。每份材料留10个生理成熟一致主茎穗直接在田间自然发芽, 1周后收回室内, 置于电热恒温干燥箱中(150℃)快速烘干, 手工脱粒, 统计每份材料发芽种子数和总粒数, 计算平均整穗发芽率。
采用SPSS19.0软件分别对3个环境下小麦整穗发芽率进行统计描述和方差分析。
选取15份亲缘关系较远的材料进行SSR引物筛选, 筛选标准为带型清晰、多态性丰富。引物信息来自Graingenes (http://wheat.pw.usda.gov/GG2/ index.shtm/), 由生工生物工程(上海)有限公司合成引物。最终筛选出180对SSR引物, 另外补充小麦穗发芽基因功能标记 Vp1B3[ 6], 总共181个分子标记用于关联分析。
采用SDS饱和酚法提取小麦基因组DNA。在NanoVue Plus微量分光光度计上检测DNA 浓度, 于-20℃冰箱保存。PCR扩增体系为10 μL, 包括10× buffer (含有2.0 mmol L-1 Mg2+) 1.0 μL, 2.5 mmol L-1 dNTPs 0.8 μL, 5 U μL-1 Taq DNA polymerase 0.11 μL, 10 μmol L-1引物各0.4 μL, 2.0 μL模板DNA (50~ 60 ng μL-1), ddH2O 5.29 μL。反应程序为94℃预变性5 min; 35个循环(95℃变性30 s, 48~62℃退火30 s, 72℃延伸30 s); 72℃延伸10 min; 4℃保存。用6%变性聚丙烯酰胺凝胶电泳检测扩增产物。利用软件Gelpro32 (http://www.mediacy.com/)分析带型。
参考Mackay等[ 24]的方法, 从180对SSR引物中选取随机分布于小麦全基因组的105对独立SSR引物对群体遗传结构进行评估。采用STRUCTURE软件[ 25]的混合模型来确定群体结构, K值取1~11, 每个 K值单独运行6次。当 K=4时曲线出现最大拐点, 因此将264份材料分为4个类群。
利用TASSLE软件[ 26]的一般线性模型(general linear model, GLM)和混合线性模型(mixed linear model, MLM), 将群体结构分析所得的 Q值和亲缘关系 K作为协变量, 进行标记与性状的关联分析, 选取两种模型共同关联到的显著( P<0.05)标记位点, 并计算这些标记位点对表型变异的解释率。
参考文自翔等[ 27]描述的方法, 对整穗发芽抗性显著关联的标记位点进一步挖掘其优异等位变异, 明确各等位变异的表型效应及其载体材料。
ai =
式中, ai代表第 i个等位变异的表型效应值, xij为携带第 i个等位变异在第 j份材料中表型值, ni为具有第 i等位变异的材料数, Nk为携带无效等位变异(某一位点中, 若其中一个等位变异被视为“有效”, 则其他等位变异相应地为“无效”)的第 k份料的表型值, nk为具有无效等位变异的材料数。若 ai > 0, 则认为该等位变异为增效等位变异, 反之为减效等位变异。
264份材料在3个环境中均表现出丰富的表型变异, 整穗发芽率变幅分别为0~92%、0~100%和0~96%, 其中2012年室内和2013年田间自然环境下的变异系数均在0.80以上。2013年室内总体平均值最高, 为69%, 2012年室内和2013年田间自然环境下总体平均值较低, 分别为25%和16% (表1)。
| 表1 264份材料在3个环境下的整穗发芽率变异 Table 1 Variation of germination percentage of intact spike in 264 wheat genotypes detected in three environments |
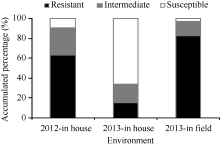
不同穗发芽抗性等级材料的分布在不同环境中表现不一, 2012室内和2013田间环境下分布趋势相似, 均有60%以上的材料分布在0~30%抗性等级中, 分布到61%~100%等级中的材料不满10%。2013室内频率分布不同于前两者, 其分布在0~30%和31%~60%等级中的材料均不满20%, 超过60%的材料分布在61%~100%等级中(图1)。究其原因可能是2013年小麦种子成熟期间的温度以及收获前降雨的影响, 造成整体供试材料穗发芽抗性水平下降。
方差分析(表2)显示, 3种环境下测得供试材料间整穗发芽率差异极显著( P=0.0001), 说明小麦穗发芽抗性既受基因型的控制, 也易受环境影响。供试材料的整穗发芽率在3种环境间的相关性均达极大, 为0.469; 2013室内和2013田间相关系数为0.334。综合分析上述结果说明, 整穗发芽抗性易受环境影响, 但在不同年份间, 以及同一年份内不同环境下, 不同供试材料整穗发芽率表现出相对稳定的趋势。
| 表2 供试264份材料整穗发芽抗性的方差分析 Table 2 ANOVA of sprouting resistance in 264 genotypes used in this study |
显著水平( P<0.01)。其中, 2012室内和 2013室内相关系数最小, 为0.286; 2012室内与2013田间相关系数最
两种模型共同检测到20个标记位点与整穗发芽抗性显著相关( P<0.05)。表3为MLM模型的检测结果。其中与2012室内关联的分子标记位点有10个, 分别位于2DS ( gwm102和 barc1146)、3AS ( barc321和 gwm369)、4AL ( wmc617)、5AS ( barc186)、5BL ( barc156)、6BS ( barc14)和7BL ( barc1181和 barc340), 各标记位点对表型贡献为2.2%~10.2%, 其中6BS上的 barc14对表型贡献最大(10.2%)。与2013室内关联的分子标记位点有11个, 分别位于染色体1AS ( barc28)、2DS ( gwm102)、3BL ( barc344和 Vp1B3)、4AL ( gwm610和 barc78)、5BL ( barc109和 barc156)、6DS ( barc173)、7AL ( barc281)和7BL ( barc340)上, 各标记位点对表型贡献为3.1%~8.0%, 其中4AL上的 barc78对表型贡献最大(8.0%)。与2013田间关联的分子标记位点有9个, 分别位于染色体1AS ( barc28)、2DS ( gwm102和 barc1146)、3AS ( barc321和 barc57)、5AS ( barc186)、5DL ( barc347)和7BL ( barc1181和 barc340)上, 各标记位点对表型贡献为3.5%~15.0%, 其中 barc321和 barc57对表型贡献相对较大(10.6%和15.0%), 均位于3AS染色体区域, 且距离较近。
| 表3 混合线性模型检测到的与穗发芽抗性相关的标记位点( P<0.05)及其对表型贡献 Table 3 Marker loci associated with preharvest sprouting resistance ( P<0.05) detected by mixed linear model and their phenotypic contributions |
3种环境下均检测到与整穗发芽抗性相关的分子标记位点有2个, 分别为 gwm102和 barc340; 2种环境下均检测到的分子标记位点有6个, 分别为 barc28、 barc1146、 barc321、 barc186、 barc156和 barc1181; 其余12个分子标记位点仅在一种环境下被检测到。说明小麦整穗发芽抗性虽然存在效应稳定的QTL, 但受基因型与环境互作的显著影响, 与方差分析结果吻合。
对8个重复检测到的标记作等位变异分析, 共鉴定出10个降低整穗发芽率的优异等位变异(表4), 其载体主要为6份材料, 即茶淀红麦、遂宁坨坨麦、扬麦158、梓潼女儿麦、川麦42和扬麦20。从各等位变异的分布频率来看, barc186-199bp在供试材料中所占比例最大(62.1%, 164/264), 其次是 barc1181- 256bp (38.6%, 102/264), barc28-229bp最小(0.7%, 2/264)。从各等位变异的表型效应来看, barc28- 229bp和 barc28-217bp在3种环境下的穗发芽程度显著低于其他类型, 其来源主要是地方品种, 如遂宁坨坨麦、涪陵须须麦和梓潼女儿麦等, 在小麦育种中难以直接利用; 而广泛分布在推广品种中的 barc186-199bp和 gwm102-142bp虽然效应值相对较小, 但易于直接利用, 应该引起重视。
不同载体材料携带的等位变异及其组合不同, 如遂宁坨坨麦, 其同时携带 barc28-217bp、 barc321- 182/172bp和 barc1181-256bp三种等位变异组合; 扬麦20同时携带 barc186-199bp和 barc340-207bp两种组合; 梓潼女儿麦同时携带 barc1146-197bp和 barc340-194bp两种组合(表4)。这可能对表型起到一种聚合的效应, 从而导致其整穗发芽抗性水平较高。
进一步分析上述10种优异等位变异在不同抗性等级材料中的分布, 发现携带优异等位变异的材料大都表现抗穗发芽特性, 说明它们对整穗发芽抗性有不同程度的贡献(表4)。如携带 barc28-229bp和 barc28-217bp的材料全部表现抗穗发芽; 携带 gwm102-142bp、 barc1146-197bp和 barc321-182/ 172bp的材料中表现抗穗发芽的比率分别为65.2% (45/69)、73.3% (11/15)和66.7% (16/24)。
业已报道, 与小麦穗发芽相关的QTL涉及多条染色体和众多区段[ 12, 13, 15, 16, 19, 22, 23]。本研究采用GLM和MLM模型对264份材料不同环境下的穗发芽表型性状及其基因型进行关联分析, 20个标记位点被共同鉴定出与小麦整穗发芽抗性关联, 分别位于1AS、2DS、3AS、3BL、4AL、5AS、5BL、5DL、6BS、6DS、7AL和7BL上, 这一结果支持上述结论。但本研究鉴定的部分标记位点与相同染色体上已报道的位点距离较远, 如 barc28(1AS)与Mohan等[ 5]定位的QTL(连锁标记为 Xbarc24和 Xbarc119), barc340(7BL)与Liu等[ 17]鉴定的2个QTL(标记区间分别为 Xbarc255- Xgwm43和 Xwmc517- Xbarc278)都较远。本研究是否发现了新位点, 有待进一步验证。今后将着手开发多种标记(SNP、EST-SSR、EST、SCAR和CAPS等)用于加密标记图谱, 同时构建适当的作图群体, 以准确定位这些关联位点。
另有一些已报道的位点在本研究中被重复鉴定, 但其表型的贡献率在不同试验中不尽相同。如3AS上的 barc321和 barc57, 在本研究中其对表型解释效应为5.7%~15.0%, 而在张海萍等[ 14]的试验中为25.6%~48.3%。本研究检测到的 wmc617 (4AL)、 gwm610 (4AL)和 barc347(5DL)与Chen等[ 16]、Torada等[ 21]和Munkvold等[ 28]定位的QTL具有相同标记, 但表型效应较小。说明小麦抗穗发芽性状受群体、遗传背景、环境条件等影响很大, 此外, 基因型与环境互作也很明显, 本研究多数位点(12个)只能在单一环境下被检测到, 方差分析也证实这一点。
本研究检测到若干整穗发芽抗性关联位点在不同环境中较为稳定, 如 gwm102和 barc340在3种环境下均显著关联目标性状, 且与之较近的标记 barc1146和 barc1181也能在2种环境下被检测到, 与Kulwal等[ 8, 23]和Liu等[ 13]的研究结果相似。说明生产上有可能通过分子标记辅助选择技术改良小麦穗发芽抗性。在8个重复检测位点上, barc28-229bp、 barc28-217bp、 gwm102-142bp和 barc340-207bp具有显著提高整穗发芽抗性的作用; 前二者可能是提高整穗发芽抗性的主要优异等位变异, 但其主要由地方品种携带, 无法直接用于小麦育种, 但应重视利用其创制中间材料。另外, 推广品种中存在一些优异等位变异, 便于直接利用, 如本研究发现扬麦158、扬麦20、川麦42均携带控制低穗发芽率(0~30%)等位变异。我们认为, 育种中可以尝试利用分布在育成品种中、效应适中且稳定的等位变异或变异组合(如 gwm102-142bp、 barc156-204bp、 barc340-207bp和 barc340-194bp)来提高穗发芽抗性水平; 在多种环境下表现稳定的QTL, 虽然其效应可能不是最大, 但比在特定环境下表达的QTL更有育种价值[ 29]。
| 表4 与整穗发芽抗性显著关联的分子标记位点优异等位变异的表型效应和相应载体材料 Table 4 Phenotypic effects and carrier materials of elite alleles at loci strongly associated with PHS resistance |
鉴定出与小麦整穗发芽抗性显著关联的分子标记, 证实2DS、3AS和7BL染色体区域含有稳定的抗穗发芽位点, 1AS和7BL等染色体上可能存在新位点。小麦整穗发芽抗性受基因型和环境共同影响。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|