生长素是植物生长发育过程中的关键激素之一,
Auxin, as one of the most important hormones, plays a key role in many processes of plant development. The
生长素(auxin)是人类最早发现的一类植物激素, 在植物向性、细胞分裂、维管组织形成和根毛发育等生长发育过程中发挥重要作用[ 1, 2, 3]。生长素早期诱导表达的原初响应基因主要有 Aux/ IAA、 SAUR( small auxin up RNA)和 GH3( Gretchen Hangen 3)等3类[ 4]。其中, Aux/ IAA家族基因是生长素原初响应基因的典型代表, 通过调控生长素信号通路的下游基因来达到调控植物生长发育的目的[ 5]。此外, Aux/ IAA还可介导生长素信号通路与油菜素内酯[ 6]、茉莉酸[ 7]、乙烯[ 8]等激素信号通路及光信号通路[ 9]之间的相互作用, 是植物生长发育调控网络中的重要调节因子。
Aux/IAA蛋白属于核蛋白, 半衰期较短, 容易降解[ 10]。典型的Aux/IAA蛋白具有4个保守结构域, 其中结构域I和II位于N端, 结构域III和IV位于C端。结构域III和IV与调控生长素响应基因表达的重要转录因子ARF (auxin response factor)的III、IV结构域具有很高同源性, 且能相互作用[ 11, 12]。在弱生长素信号条件下, Aux/IAA与ARF结合, 抑制下游基因转录, 关闭生长素信号通路; 反之, Aux/ IAA在泛素介导下降解, 释放ARF, 开启通路[ 13]。在上述过程中, 结构域I起转录抑制作用, 其对ARF的转录激活具有显著的上位作用[ 14]; 结构域II影响Aux/IAA蛋白的稳定性, Aux/IAA的降解依赖于结构域II与SCFTIR1泛素连接酶复合体的结合[ 15], 结构域II的突变可导致Aux/IAA蛋白降解受阻。
1982年首次在大豆中获得 Aux/ IAA基因[ 16]。随着许多重要物种基因组测序工作的完成, 使全基因组范围内研究 Aux/ IAA基因成为可能。在拟南芥[ 17]、水稻[ 18]、杨树[ 19]、玉米[ 20]、棉花[ 21]、番茄[ 22]、黄瓜[ 23]等植物中, 基于全基因组的分析结果表明 Aux/ IAA基因家族成员具有不同的生物学功能, 如棉花 GhAux8和 GhIAA16可能在棉花纤维早期伸长期发挥重要作用, 小麦 TaIAA1调控根、分蘖、花期和叶型等重要器官的发育[ 24], 许多重要基因可用于作物改良。 Aux/ IAA全基因组分析不仅有助于阐述生长素代谢和信号传导的分子机制, 而且其中重要的功能基因可用于作物的遗传改良。
小麦是世界上主要的粮食作物之一, 属于异源六倍体, 基因组庞大而复杂, 与水稻等模式作物相比, 对小麦株型、产量和非生物逆境等功能基因的研究和利用较落后。小麦A基因组供体乌拉尔图小麦( Triticum urartu)和D基因组供体粗山羊草( Aegilops tauschii)及中国春测序草图的公布极大促进了小麦基因组学研究[ 25, 26, 27]。粗山羊草具有抗病、抗旱、抗逆等优良特性, 其测序工作的完成使基于全基因组的基因功能分析成为可能, 更为小麦基因组学研究提供了重要参考信息。虽然全基因组测序结果显示粗山羊草转录因子可分为58类[ 26], 但对具有重要功能的 Aux/ IAA家族还未分类。
本研究采用生物信息学方法分离了粗山羊草 Aux/ IAA家族基因, 分析了该家族成员的染色体位置、序列特征、基因结构、表达模式和进化等, 相关基因可作为小麦遗传改良的候选基因, 同时本研究也为小麦中 Aux/IAA基因的深入研究与利用提供了有价值的信息。
粗山羊草的全基因组、CDS、表达信息及乌拉尔图小麦的CDS数据均来自AGDB数据库(http://dd.agrinome.org/); 粗山羊草定位序列片段、小麦EST标记信息来自GrainGene2.0数据库(http://wheat.pw.usda.gov/); 拟南芥、水稻、短柄草的Aux/IAA蛋白序列信息来自NCBI (http://www.ncbi.nlm.nih.gov/)数据库; 大麦Aux/IAA蛋白序列信息来自PlantTFDB数据库(http://planttfdb_v1.cbi.pku.edu.cn:9010/)。
从AGDB数据库中下载粗山羊草全基因组数据建立本地BLAST数据库。从Pfam数据库(http://pfam.sanger.ac.uk/)中下载 Aux/ IAA家族(登录号为PF02309)的隐马尔可夫模型文件, 再用HMMEMIT软件[ 28]获得长度为215个氨基酸残基的Aux/IAA蛋白保守基序, 用该保守基序检索本地粗山羊草基因组序列数据库, 检索程序为HMMPfam, E值设为0。
检索结果剔除冗余序列, 利用SMART工具[ 29] (http://smart.embl-hei-delberg.de/)和Pfam中的Sanger工具[ 30]对非冗余序列进行Aux/IAA蛋白保守结构域的检查, E值设为1.0, 其他参数为默认值。
粗山羊草 Aux/ IAA基因的相关信息从AGDB数据库获得, 包括CDS序列、基因组序列、基因组位置。将粗山羊草 Aux/ IAA基因组序列提交GrainGene 2.0数据库, 检索相似性>95%、E值为0的粗山羊草序列片段; 然后查找所得序列片段和附近的EST标记。利用这些序列片段的定位和EST标记信息, 将粗山羊草 Aux/ IAA基因整合到粗山羊草分子图谱[ 31]上。根据染色体位置对粗山羊草 Aux/IAA家族基因命名。采用GSDS软件[ 32] (http://gsds.cbi.pku.edu.cn/)分析 AetIAA的结构。
用DNAstar软件中的Editseq工具将 AetIAA CDS翻译为氨基酸。用NPS在线工具(http://npsa-pbil.ibcp.fr/cgi-bin/secpred_sopma.pl)对AetIAA蛋白进行二级结构预测。用Clustal X软件进行蛋白序列多重比对, 参数为默认值, 用Jalview输出比对结果。采用MEME工具[ 33] (http://meme.nbcr.net/meme4_1/cgi-bin/meme.cgi)分析AetIAA蛋白的基序, 除基序长度范围设为10~300个氨基酸残基, 基序最大发现数目为4个外, 其他均为默认值。
截取 AetIAA家族起始密码子上游2000 bp的基因组序列作为启动子区, 利用PLACE26.0 (http://www.dna.affrc.go.jp/PLACE/signalscan.html)和Plant CARE (http://bioinformatics.pAet.ugent.be/webtools/plantcare/html)数据库分析基因的顺式作用元件。
从AGDB数据库中检索 AetIAA基因在8个组织器官中的EST数据。对数据进行log2转换处理, 利用Mev软件对基因表达数据进行聚类分析, 并显示分析结果。
利用含215个氨基酸残基的Aux/IAA蛋白保守基序在本地数据库进行tBLASTN检索, 对获得的序列进行Aux/IAA保守结构域检测, 获得28个粗山羊草 Aux/ IAA的基因组和CDS序列。根据GrainGene2.0数据库中的定位结果, 将这28条序列( AetIAA1~ AetIAA28)定位于粗山羊草的7条染色体上, 但每条染色体(臂)上 AetIAA基因分布不均, 其中7D和5D上较多, 分别为8个和6个, 4D上最少, 仅1个( 表1和 图1)。
将28个 AetIAA基因整合到粗山羊草分子图谱上, 发现每个 AetIAA都有遗传距离较近的EST标记, 其中5个基因( AetIAA2、 AetIAA3、 AetIAA5、 AetIAA8和 AetIAA25)分别获得了位于同一位点的标记( 图1)。标记BE497375位于 AetIAA25的CDS区。
| 表1 粗山羊草 Aux/ IAA家族基因 Table 1 Aux/ IAA gene family in Ae. tauschii |
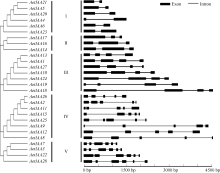
28个 AetIAA的序列长度和内含子(外显子)数目差异较大, 序列最长的为 AetIAA8 (4642 bp), 最短的为 AetIAA21 (310 bp), 每个基因含1~5个内含子( 图2)。根据基因结构, 将这28个 AetIAA基因划分为5组, I组序列普遍较短, III和IV组普遍较长, 内含子长度是组内成员序列长度变异的主要来源( 图2)。由于具有相似的基因结构, 同组不同成员可能具有相似功能。
基序分析和序列多重比对结果表明, 20个AetIAA蛋白(71.4%, 20/28)具有保守结构域I、II、III和IV ( 图3), 这4个保守结构域包含在4个基序中( 图4); 8个AetIAA蛋白(28.6%, 8/28)不具有Motif 1 (Domain I), 4个AetIAA蛋白(14.3%, 4/28)不具有Motif 2 (Domain II); 1个AetIAA蛋白(3.6%, 1/28)不具有Motif 4 (Domain IV); 所有蛋白均含有Motif 3 (Domain III)。
与水稻[ 19]和玉米[ 21]等的Aux/IAA蛋白类似, AetIAA存在一些特征序列。Domain I具有1个两亲性基序LxLxLx, 是Aux/IAA蛋白转录抑制功能所必需的区域[ 12]。Domain II含有Aux/IAA蛋白泛素化降解的靶位点, 其核心序列为VGWPP, 该区域的显性突变会使Aux/IAA蛋白不能够进入泛素化途径而导致稳定性增强[ 36]。Domain III含有1个β折叠与2个α螺旋(α1和α2)构成的βαα基序, 在Aux/IAA蛋白二聚化过程中发挥重要作用[ 12]。大部分粗山羊草Aux/IAA蛋白中还存在2个假定的核定位信号(NLS, nuclear localization signal)[ 37], 一个是由Domain I和Domain II之间的KR与Domain II部分保守序列组成的二分结构(bipartite structure), 另一个是位于Domain IV的SV40-型NLS。这些假定NLS表明AetIAA蛋白可能在细胞核中起作用。此外, Domain II还具有1个光敏色素A的磷酸化位点, 因此推测AetIAA蛋白可能通过光敏色素A的磷酸化作用介导生长素与光信号通路[ 37]。
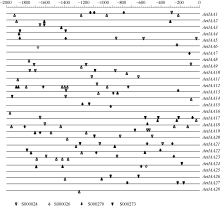
截取起始密码子上游2000 bp的基因组序列, 对 Aux/ IAA启动子区4种参与生长素信号转导的顺式作用元件(PLACE 26.0数据库编号为S000024、S000026、S000270和S000273)进行分析。其中S000026和S000270属于生长素响应元件(ARE, auxin response element), 其作用是在生长素应答中与ARF特异绑定[ 13]。分析结果表明, 28个 AetIAA基因的启动子区均含有与生长素信号转导相关的顺式作用元件, 22个基因有S000024作用元件, 15个含有S000270作用元件, 17个含有S000273作用元件, 2个含有S000026作用元件( 图5)。可见, AetIAA家族各基因受生长素调控表达的模式存在差异。
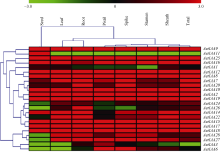
检索AGDB数据库获得23个 AetIAA基因的EST数据。 AetIAA8、AetIAA9、AetIAA17和 AetIAA18等大多数基因(87%, 20/23)在供试组织器官中表达, 这类基因属于组成型表达基因; 少部分基因呈现组织特性表达的特点, 如 AetIAA3、AetIAA11和 AetIAA26分别在雌蕊、种子和根中有较高表达。另外, AetIAA27和 AetIAA28的表达谱极为相似, 即在种子中不表达, 而在其他组织器官中表达( 图6), 推测这两个基因可能具有相似功能。
总体上, AetIAA家族基因在叶鞘中表达量最高(1775.1), 其次是在叶片中(1443.7)和根中(1387.1)。
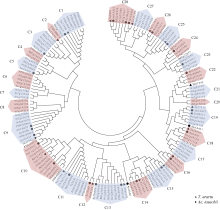
选取155个Aux/IAA蛋白, 来自小麦(1个)、粗山羊草(28个)、乌拉尔图小麦(17个)、大麦(25个)、短柄草(27个)、水稻(28个)和拟南芥(29个)。系统进化分析结果显示, 这些Aux/IAA蛋白可分为28组( 图7), 其中C6、C9、C18和C23组中同时包含拟南芥和禾本科植物的Aux/IAA, 推测其 Aux/ IAA基因的发生在单双子叶植物分化之前, 属直系同源基因; 另外, Aux/ IAA旁系同源基因可能在拟南芥中有28个(97%), 大麦中有12个(48.0%), 推断其在单双子叶植物分化之后按物种特有的方式进行了扩张。
在28个AetIAA蛋白中, 有11对粗山羊草-乌拉尔图小麦、5对粗山羊草-大麦、2对粗山羊草-短柄草和1对粗山羊草-水稻Aux/IAA蛋白直系同源, 未发现处于同一进化分支粗山羊草和拟南芥Aux/IAA蛋白。
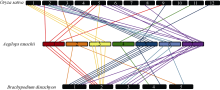
基于AGDB数据库公布的同源基因信息, 对28个 AetIAA基因及其对应的水稻和短柄草基因进行共线性分析表明, 粗山羊草第3染色体上的4个 AetIAA基因全部与短柄草第2染色体上的 BdIAA基因同源, 同时与水稻第1染色体上3个 OsIAA基因同源( 图8)。初步推测, 粗山羊草第3染色体与短柄草第2染色体及水稻第1染色体具有较好的共线性。此外, 粗山羊草第1染色体与短柄草第2染色体和水稻第5染色体、粗山羊草第5染色体与短柄草第1染色体和水稻第3染色体、粗山羊草第6染色体与短柄草第3染色体和水稻第2染色体、粗山羊草第7染色体与短柄草第3染色体和水稻第6染色体均具有较好的共线性( 图8), 这与已报道的研究结果[ 39]一致。
粗山羊草是普通六倍体小麦D基因组的供体, 已从粗山羊草中鉴定和筛选出大量优良基因用于小麦育种[ 40]。粗山羊草及小麦A基因组供体乌拉尔图小麦测序草图的公布极大推动了小麦的基因组学的研究。
生长素是植物生长发育过程中重要的调控因子。 Aux/ IAA基因家族在生长素信号转导途径中发挥重要作用。全基因组测序结果表明, 粗山羊草中包含1489种转录因子, 涉及56个基因家族[ 26]。本文初步研究了粗山羊草中一个新的 Aux/ IAA家族, 共分离出28个 AetIAA基因, 与番茄(26个)、拟南芥(29个)和水稻(31个)等数目相当, 表明 Aux/ IAA家族基因在这些植物的进化过程中具有一定保守性。基于小麦、大麦、水稻和拟南芥等植物Aux/IAA蛋白的系统发育分析表明, C6、C9、C18和C23中同时包含拟南芥和禾本科植物的Aux/IAA序列, 表明这些 Aux/IAA基因的发生在单双子叶植物分化之前; 其他24组仅包含禾本科植物或拟南芥的Aux/IAA蛋白, 推测这些基因发生于单双子叶分化之后或在进化过程中丢失。另外, 粗山羊草中有2对 AetIAA基因位于同一个姐妹群( AetIAA4/AetIAA13和 AetIAA6/ AetIAA20), 推测其功能可能具有冗余性。
分析基因在特定组织的表达丰度是研究基因功能的一个重要方面。本研究分析了23个 AetIAA的表达数据, 大多数(87%, 20/23) AetIAA基因属于组成型表达基因, 在所有检测组织中都表达, 推测 AetIAA基因功能可能存在冗余性; 另外3个基因的表达呈现组织特异性, AetIAA3在雌蕊 、AetIAA11在种子、 AetIAA26在根中有较高表达量, 推测这3个基因可能分别在粗山羊草花器官、种子及根部发育过程中起作用。其中 AetIAA26与水稻 OsIAA23 (Os6g39590)基因直系同源( 图7的C13组), 而 OsIAA23已被证实在水稻根尖静止中心的特征建立中有重要作用, 并显著影响水稻的育性、结实率和产量[ 41, 42]; 同时图谱分析发现, AetIAA26位于一个产量QTL ( QTwt.crc-7D)[ 43]区域内, 距其约7 cM处还有另一个产量QTL ( QGfrmax.nfcri-7D)[ 44], 进一步推断 AetIAA26可能通过调控粗山羊草根的发育来决定育性和产量。
水稻中 OsIAA13也与侧根的发育有关, 然而与其同处一个进化分支的 AetIAA15并未表现根部特异表达, 可能基因在各自进化过程中功能也出现了分化。本研究未检索到 AetIAA4、 AetIAA45、 AetIAA13、 AetIAA21和 AetIAA23的表达数据, 在水稻[ 18]和玉米[ 20]中也有类似情况, 其一可能这些基因具有特殊的时空表达模式, 如水稻 Aux/ IAA基因在光照和黑暗条件下的组织特异性表达模式存在差异[ 18]; 其二粗山羊草 Aux/IAA基因中可能存在假基因, 如缺失部分功能域的 Aux/ IAA基因可能为假基因[ 10]。
Aux/IAA蛋白功能的发挥很大程度依赖于4个保守结构域。本研究中8个AetIAA蛋白(29%, 8/28)缺失Domain I, 4个AetIAA 蛋白(14%, 4/28)缺失Domain II, 几乎所有AetIAA蛋白都具有Domain III和Domain IV。Domain I具有抑制ARF转录活性的作用, 含有LxLxL基序; 番茄Aux/IAA蛋白的抑制活性与LxLxLx基序的存在高度相关[ 45], 本研究中大部分AetIAA 蛋白含有LxLxL基序, 少数蛋白如AetIAA5、AetIAA11等含有LxL基序; Domain II可在较高生长素水平下与TIR1互作导致Aux/IAA蛋白降解[ 16]。研究表明, 缺失Domain II的Aux/IAA蛋白半衰期较正常蛋白更长[ 37]。理论上, 类似 AetIAA6、
AetIAA7、 AetIAA20和 AetIAA21等缺失Domain II的 Aux/ IAA应对IAA处理反应较迟钝; 然而, 番茄 SlIAA16在IAA处理6 h后的转录水平增加9倍[ 22], 在水稻中也发现类似现象[ 18]。因此, 推测这类蛋白的降解可能通过其他途径实现。
本研究中, 28个 Aux/ IAA基因虽然分布在粗山羊草基因组的所有7条染色体上, 但明显聚集在7D和5D上, 占整个基因家族的50%, 且都分布在染色体中部或着丝粒附近; 有3对邻近基因分别位于为5D ( AetIAA14/ AetIAA15)、6D ( AetIAA19/ AetIAA20)和7D ( AetIAA27/ AetIAA28)上, 在拟南芥、水稻和番茄中也存在类似现象[ 17, 18, 22], 这种情况可能由正向或反向的串联重复(tandem duplication)所致, 这也是基因扩增的一种重要方式。将28个 AetIAA基因整合到粗山羊草分子图谱上, 发现 AetIAA2、 AetIAA3、 AetIAA5、 AetIAA8、 AetIAA25分别具有位于同一位点的EST标记, 其中BE497375恰好位于 AetIAA25的CDS区, 这为今后研究该基因的功能提供了极大便利。本研究还发现, AetIAA与模式植物水稻 OsIAA和短柄草 BdIAA存在很高程度的共线性, 因而可以利用已知的水稻 Aux/ IAA功能信息对粗山羊草相应基因进行功能预测, 同时也为 Aux/ IAA基因在不同物种间的进化提供有价值的线索。
从粗山羊草全基因组中分离出28个 Aux/ IAA基因, 分布于7条染色体上, 其中5个基因分别检测到位于同一位点的EST标记。 AetIAA3、 AetIAA11和 AetIAA26分别在雌蕊、种子和根中特异表达, 可能参与调控相应器官的发育和建成。粗山羊草与乌拉尔图小麦亲缘关系最近, 其次是大麦。粗山羊草第3染色体上的 Aux/ IAA基因与短柄草第2染色体及水稻第1染色体上的相关基因共线性较好。本研究结果为小麦中 Aux/ IAA基因研究与利用提供了重要信息。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|