* 通讯作者(Corresponding author): 田静, E-mail:nkytianjing@163.com, Tel: 0311-87670655
第一作者联系方式: E-mail:Liuchangyou2006@yahoo.com.cn
利用28对SSR引物对96份野生小豆资源、小豆近缘野生植物及栽培小豆品种进行遗传多样性分析, 共检测到255个等位变异, 平均每对SSR引物9.10个, 多态信息含量的变异范围从0.374到0.865, 平均为0.722。野生小豆材料和近缘植物
Wild Adzuki bean (
小豆[ Vigna angularisWilld. Ohwi & Ohashi]是豇豆属( Vigna)亚洲豇豆亚属( Ceratotropis)中重要的栽培种之一[ 1], 在我国除个别高寒区外, 在全国各地均有种植, 主产区集中在华北、东北和江淮地区, 是我国重要的出口农产品之一[ 2, 3]。近年来通过杂交选育, 小豆育种工作虽然取得了一定进展, 但一直缺乏突破性的品种[ 4]。种质资源是育种工作的基础, 其中野生资源发挥着重要的作用[ 5]。研究者在野生水稻、野生大豆等野生资源中发掘了许多优异基因, 如水稻抗病基因[ 6], 大豆耐盐基因[ 7]等。国外研究者在野生小豆及小豆近缘植物中也发现了一些优异种质材料。如抗豆象野生小豆材料能够防止豆象危害[ 8], 小豆近缘野生种 Vigna minima可以作为小豆和饭豆间的桥梁亲本[ 9]等。中国是小豆的起源地之一, 野生小豆和小豆近缘野生植物也有大量分布[ 1]。加强对这些资源的搜集、鉴定和利用对小豆育种具有重要意义。
然而目前从国内搜集到的野生小豆及近缘野生植物资源并不多。在已经开展的野生小豆遗传多样性研究中主要利用国外的野生小豆材料。如金文林等[ 10]、陶宛鑫等[ 11]和赵波等[ 12]主要利用日本野生小豆材料作为研究对象, 中国野生小豆材料仅有3份; 宗绪晓等[ 13]在研究栽培小豆和野生变种的演化与地理分布关系时选用了5份国内野生小豆材料。这些现象说明我们对国内野生小豆材料的搜集工作做得不够。依托国家食用豆产业技术体系平台, 我们与中国农业科学院作物科学所的程须珍研究员的课题组一起, 在北京、河北、天津、辽宁、山东等地搜集到野生小豆和小豆近缘野生植物 Vigna minima。对这些材料的评价和分析可以指导我国野生小豆资源的进一步搜集和利用。通过前期工作我们已经验证了这些野生小豆资源以及 Vigna minima与栽培小豆间的杂交可育性(数据未发表)。为了对它们进行遗传评价, 本研究利用Han等[ 14]已经公开发表的小豆SSR标记序列, 分析其遗传多样性, 以期为我国野生小豆及其近缘野生植物资源保存、鉴定、评价及新的优异基因的发掘提供参考。
野生小豆材料69份, 包括中国天津蓟县(TJJX) 32份、辽宁大连(LNDL) 17份、辽宁丹东(LNDD) 5份和日本(Japan) 15份; 小豆近缘野生植物 Vigna minima21份, 包括天津蓟县1片、河北遵化(HBZH) 9份、北京平谷(BJPG) 5份、山东烟台(SDYT) 6份; 小豆近缘野生植物 Vigna nakashimae1份, Vigna riukiuensis 1份; 栽培小豆材料4份(白红99616、冀红352、冀红9218和京农6号)。
从每份试验材料挑选出5粒健康种子播在营养钵内, 采集刚展开的第一片三出复叶, 利用天根公司的植物基因组DNA快速提取试剂盒提取DNA, 用1%的琼脂糖凝胶检测DNA质量。经紫外分光光度计测定浓度后, 加ddH2O稀释至10 ng μL-1, 备用。
从196对小豆SSR引物中筛选中28对扩增条带清晰、多态性好的引物用于后续分析。PCR扩增反应在ABI Applied Biosystems Veriti 96 Well Thermol Cycler扩增仪上进行, 反应体系为10 μL, 包含1×PCR缓冲液, 2 mmol L-1 MgCl2, 0.1 mmol L-1 dNTPs, 0.4 μmol L-1的引物, 20 ng DNA和0.5 U Taq DNA聚合酶。反应程序为95℃预变性3 min; 94℃变性30 s, 退火30 s, 72℃延伸45 s, 循环35次; 72℃延伸7 min。扩增产物经8%的非变性聚丙烯酰胺凝胶电泳分离, 在200 V恒压下电泳, 根据PCR产物分子量大小及差异带型的可辨程度调整电泳时间。将凝胶从玻璃板转移到染色盒, 用蒸馏水洗涤2次, 然后用0.2% AgNO3溶液染色8 min, 蒸馏水快速漂洗2次, 加1.5% NaOH (0.5%甲醛)溶液显影至条带清晰, 再用蒸馏水洗掉凝胶上残留的染色液, 拍照读带。
按照各SSR引物扩增片段迁移位置的不同分别读取数据, 依据不同分析软件需求的差异在Microsoft Excel中相应转换数据格式。用非加权成组配对算术平均法(unweighted pair-group method with arithmeticmean, UPGMA)计算各种质材料间Nei-Li’s遗传相似性系数[ 15], 用NTSYS-pc 2.10t作树状聚类图及主成分分析; 利用PopGen32软件包计算多态信息含量(polymorphism information content, PIC)。
28对SSR引物在96份野生小豆、栽培小豆及其近缘野生植物中共检测到255个等位变异, 每对引物检测到等位变异的数目为4~14个, 平均每SSR位点9.10个(表1)。其中等位变异最丰富(14个)的引物为N27 (连锁群1)、N102 (连锁群5)、N114 (连锁群6)和N163 (连锁群9); 仅检测到4个等位变异的引物共2个, 分别为N60 (连锁群2)和N73 (连锁群3), 占7.14%。等位变异频率从1.04%至76.04%不等, 平均为10.77%。分布频率高于50.0%的等位变异为6个, 占2.35%, 即在6个SSR位点检测出优势等位变异; 分布频率低于5%的等位变异占50.98%。多态信息含量的变异为0.374~0.865, 平均为0.722。其中多态信息含量最高的引物是N111, 最低的是N73, 分别位于小豆的第5和第3连锁群。除N56、N73和N149 3个引物外, 其他SSR引物多态信息含量均高于0.5。
由表2可知, 来自日本的野生小豆材料检测到的等位变异数和特异等位变异数最多, 分别为94个和31个。来自天津蓟县的野生小豆材料虽然检测种质数量(32份)最多, 但等位变异数却与检测种质数仅有5份的辽宁丹东野生小豆材料相同, 均为64个, 表明来自天津蓟县的部分野生小豆材料间遗传差异较小, 可能存在重复取样现象。然而从其具有较多的特异等位变异数(30个)及特异等位变异比最高(44.78%)来看, 来自该地域的野生小豆具有特殊的遗传组成。在来自日本的一份 Vigna nakashimae材料中未检测出特异等位变异, 其等位变异位点均与 Vigna minia的变异位点相同。
| 表1 不同连锁群SSR位点检测的等位变异数及遗传多样性指数 Table 1 Number of alleles and genetic diversity index for SSR loci in each linkage group |
| 表2 不同来源种质的检测数目、等位变异数及特异等位变异 Table 2 Number of accessions, alleles, and specific alleles in different species |
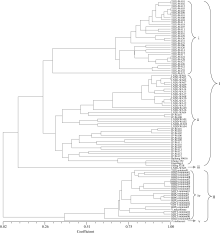
基于UPGMA的聚类分析显示(图1), 利用28对SSR引物, 可以将小豆类群(I)、和小豆近缘植物类群(II)在遗传上完全分开。其中小豆类群(I)又可以分为3个组群, 第一组群(i)均为来自中国天津蓟县的野生小豆材料, 第二组群(ii)包含来自中国辽宁的野生小豆材料和来自日本的野生小豆材料以及4个栽培小豆品种。第三组群(iii)仅有来自中国辽宁大连的1份野生小豆材料。小豆近缘植物类群(II) 也可分为2个组群, 其中第四组群(iv)包括所有的 Vigna minima材料和1份 Vigna nakashimae, 来自不同地域的 Vigna minima基本按照地理来源聚在一起, Vigna nakashimae不能和 Vigna minima区分开。第五组群(v)中 Vigna riukiuensis单独分为一类。
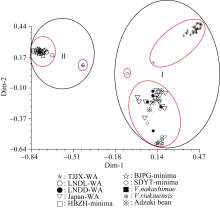
进一步进行主成分分析, 从图2可以更直观的看出小豆类群(I)集中在右侧成群, 而近缘植物类群(II)集中在左侧成群, 其他上述各组群之间界限也非常清楚, 结果显示基于主成分分析的二维坐标图与聚类分析吻合。
以DNA分子标记为手段进行遗传多样性检测被广泛应用到作物种质资源研究中, 成为种质资源鉴定、分类强有力的佐证和检验手段[ 16]。在小豆SSR标记公布之前, 我国研究者主要利用非物种特异性的分子标记如RAPD、AFLP等分析小豆种质资源的遗传多样性, 如金文林等[ 10]利用25个RAPD引物分析了16份野生小豆材料的遗传多样性, 王述民等[ 17]、宗绪晓等[ 13, 18]和粟生群等[ 19]均利用AFLP标记分析了不同小豆种质资源的遗传多样性。在Wang等[ 20]和Han等[ 14]开发的小豆SSR标记公布后, 这些标记成为近年来用于小豆资源遗传多样性分析的主流。徐宁等[ 21]、王丽侠等[ 22, 23, 24]和赵波等[ 12]先后利用小豆SSR标记对国内及部分国外小豆种质资源的遗传多样性进行了分析。本研究利用从Han等[ 14]公开发表的196对小豆SSR标记中筛选出的28对多态性高的标记, 分析近期从国内搜集到的野生小豆资源、小豆近缘野生植物及部分日本野生小豆资源, 28对引物共检测到255个等位变异, 高于王丽侠等[ 23]利用51对SSR引物在145份栽培小豆种质中检测到的等位变异数(222), 这一方面表明小豆SSR标记在野生小豆及小豆近缘野生植物的遗传多样性分析中的有效性, 另一方面也表明野生小豆资源及小豆近缘野生植物中蕴藏着更丰富的遗传变异, 这与Xu等[ 25]的观点一致。
此外, 鉴于本研究中所使用的国内野生小豆材料和小豆近缘野生植物资源均取自我国东北和华北地区, 因此还安排了4份来自东北和华北地区的栽培小豆品种作为野生小豆的对照种。从等位变异位点、聚类分析结果以及主成分分析结果来看, 这些国内育成品种中均含有日本野生小豆材料的遗传背景, 而与国内的野生材料遗传差异更大一些, 这表明中国目前的小豆育种中大量使用了日本小豆材料或具有日本小豆血缘的材料, 另外也从侧面表明中国对国内野生小豆资源的搜集和利用要落后于日本。因此应加强对国内不同地理来源(尤其是本研究尚未涉及的我国南方地区)的小豆野生材料的搜集、鉴定和评价, 为小豆育种提供新的优异基因。
小豆属于短日照植物, 大多数材料对光周期反应比较敏感, 这可能是来自不同地域的野生小豆资源的遗传组成差异的原因之一。王述民等[ 17]、宗绪晓等[ 13, 18]和粟生群等[ 19]利用AFLP分子标记分析小豆资源的遗传多样性中均表明了这一观点; 徐宁等[ 21]和王丽侠等[ 24]利用SSR标记分析栽培小豆种质的遗传多样性时也有同样发现。本研究在对国内不同来源地的野生小豆资源的多样性分析中发现, 野生小豆材料具有大量特异等位变异, 且不同地理来源间明显单独分群。由于野生小豆不像栽培小豆那样可能经过杂交或地域间的交流, 因此更能够说明地理来源和生态环境对小豆的遗传组成具有较大影响, 野生小豆的遗传组成与其地理来源有明显关系。
根据Siriwardhane等[ 9]和Tomooka等[ 26]和我们前期的杂交验证(数据未发表), 小豆与野生小豆、 Vigna minima、Vigna nakashimae、 Vigna riukiuensis间均可以正常杂交结实, 且 Vigna minima、Vigna nakashimae、 Vigna riukiuensis还可以作为小豆与饭豆间的桥梁亲本, 用于小豆与饭豆间遗传物质的融合。这表明小豆与这些种质之间具有较近的遗传关系。从本研究的聚类结果看栽培小豆与野生小豆的亲缘关系最近, 其次是 Vigna minima和 Vigna nakashimae, 与 Vigna riukiuensis的亲缘关系最远。
从聚类结果来看本研究中有一部分相同地理来源的材料遗传相似度较大, 利用28对SSR引物无法将其区分开。如TJJX-WA01与TJJX-WA02间、TJJX-WA03与TJJX-WA05间、TJJX-WA09与TJJX-WA10间、TJJX-WA24与TJJX-WA28间、LNDL-WA02和LNDL-WA03间、LNDL-WA10和LNDL-WA11间。这种现象一方面可能由于我们采用的SSR标记的数目或多态性不足, 另一方面也可能由于我们在野生小豆资源收集过程中存在少量的重复取样。此外, 也可以看出我们在搜集中获得了一些遗传特异材料, 如辽宁的野生小豆LNDL-WA05, 天津蓟县的野生小豆TJJX-WA023, 在以后的保存中可以根据此分析结果去除冗余的材料。
致谢: 感谢唐山市农业科学院刘振兴研究员和辽宁省农业科学院葛维德研究员等人在野生资源搜集过程中提供的帮助。感谢王丽侠博士在数据分析中提供的指导。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|