以甜菜高产低糖型JV34-2和低产高糖型2B023两材料杂交, 构建了200个单株的F2作图群体, 利用所筛选出的56对SRAP引物组合和20对SSR引物, 对F2作图群体进行PCR扩增和遗传连锁分析, 初步构建了一张包含9个连锁群、141个(123个SRAP和18个SSR)标记位点的甜菜遗传连锁图谱。该图谱覆盖长度为1399.88 cM, 平均图距9.92 cM。未进入连锁群的有4个标记。9个连锁群包含3~26个标记不等, 连锁群遗传距离15.69~237.21 cM。连锁群上有20.56%的标记出现偏分离, 主要集中在Chr.3连锁群上, 其余分散在Chr.1、Chr.2、Chr.8和Chr.9中。该图谱是我国甜菜领域利用SRAP和SSR相结合方法, 构建的第一个较精密的分子遗传图谱, 为重要性状的基因定位和优良基因的克隆奠定了基础。
Molecular genetic map is a fundamental tool for genomic research. In this study, a molecular genetic map was constructed using an F2 population consisting of 200 individuals from a cross between sugarbeet JV34-2 (the high beet yield and low sugar content line) and sugarbeet 2B023 (the low beet yield and high sugar content line) and a total of 561 SRAP primers and 114 SSR primers. Approximately 56 out of 561 and 20 out of 114 primers, respectively, showed polymorphisms. Each of these polymorphic primers produced at least one scorable polymorphic DNA band which was visible enough for detection and scoring. The map consisted of nine linkage groups which included 141 (123 SRAP and 18 SSR) markers, and covered 1399.88 cM with an average distance of 9.92 cM. Three markers were still unlinked. All nine linkage groups consisting of 3-26 markers were 15.69-237.21 cM in length. Twenty point five six percent partially segregated markers distributed in the map, were mainly located on Chr.3 linkage groups, and the others were mapped on Chr.1, Chr.2, Chr.8, and Chr.9. This is the first gene map of sugarbeet, constructed by SRAP and SSR markers in China. It will provide the basis for gene mapping of important characters and cloning of excellent gene in sugarbeet.
我国甜菜育种技术相对落后, 近几年国外品种大量进入我国, 我国甜菜育种以及甜菜种子、甜菜糖业存在重大安全隐患。为防止国外品种垄断, 培育出国产高产、高糖、抗病的新品种, 进一步研究优良基因定位和克隆等, 有必要建立我国自主的甜菜分子标记遗传图谱。目前国内尚未建立一套精密的甜菜遗传图谱, 严重影响了我国甜菜分子育种的发展。国外虽早有甜菜遗传图谱的相关报道, 但应用亲本不同, 构建的群体不同, 所得到的遗传信息也不同。而且到目前为止, 已构建的甜菜分子遗传连锁图谱中主要应用的DNA标记是AFLP、RFLP、RAPD和SSR, 尚未见报道应用SRAP。如1996年Hallden等[ 1]利用RFLP构建了包括431个标记, 覆盖620 cM的甜菜遗传图谱, 标记间平均间距1.5 cM; 1995年Barzen[ 2]构建了包含248个RFLP和50个RAPD位点, 全长815 cM的遗传连锁图谱, 并对抗丛根病基因、下胚轴颜色基因和单粒型性状基因进行定位; Uphoff[ 3]构建了包括85个RAPD标记位点的糖甜菜遗传图谱, 定位了下胚轴颜色基因、一年生基因及一个恢复基因; 1999年Nisson等[ 4]用221个AFLP和46个RFLP标记对204个植株群体进行了甜菜叶斑病抗性的QTL分析; 2002年Schneider等[ 5]对甜菜的含糖率、产量和品质进行了QTL分析; 2007年Grimmer和Trybush建立在AFLP、SNP、RAPD标记基础上的甜菜锚定连锁图和抗甜菜坏死黄脉病毒的QTL定位[ 6]。本研究利用SRAP和SSR两种分子标记方法, 以甜菜材料JV34-2 (高产低糖品系)和2B023 (低产高糖品系)为亲本, 构建F2代作图群体, 初步构建甜菜的遗传图谱, 为甜菜QTL定位、标记辅助选择育种等奠定基础。
2010年春季以二倍体甜菜单胚品系JV34-2 (高产低糖型)和二倍体甜菜多胚品系2B023 (低产高糖型)为亲本, 杂交获得F1种子, 同年8月初在山东高密地区南繁, 11月初收获F1母根并运回北方放入地窖内储藏使之通过春化处理(2~4℃), 60 d后即2011年1月初栽植于温室进行F1自交, 2011年5月初获得F2代种子, 播种于呼兰校区试验田, 得到F2作图群体, 从中选用200株作为试验样品。
在苗期每次取50份供试材料的单株幼嫩叶片2片, 用冷冻真空抽干机处理(-50℃左右, 36~48 h), 后研磨成粉末, 用CTAB法提取基因组DNA[ 7]。
SRAP-PCR反应体系含2.5 mmol L-1的dNTPs 1.2 μL, 上下引物各40 ng, 10×buffer (已含Mg2+) 2 μL, TaqDNA聚合酶1 U, DNA 40 ng, 以ddH2O调整至20 μL。反应程序为94℃ 3 min; 94℃ 1 min, 35℃ 1 min, 72℃ 1 min, 5个循环; 94℃ 1 min, 50℃1 min, 72℃ 1 min 35个循环; 72℃ 10 min。20 μL SSR-PCR反应体系含2.5 mmol L-1 dNTPs 0.6 μL, 正反引物各40 ng 10×buffer (已含Mg2+), TaqDNA聚合酶1 U, 模板DNA 60 ng。反应程序为94℃ 4 min; 94℃ 1 min, 50℃ 1 min, 72℃ 1.5 min, 30个循环; 72℃ 10 min。用8%非变性聚丙烯酰胺凝胶电泳检测PCR产物, 电泳缓冲液为0.5×TBE。硝酸银染色观察结果。试验中选用的SSR引物和SRAP引物均来源于已发表的参考文献[ 8, 9, 10], 并由北京华瑞生物技术公司合成, 引物序列和组合见表1、表2和表3。
按点样顺序记录多态性条带: (1)共显性标记中同父本带型相同的记为“1”, 与母本带型相同的记为“0”, 杂合条带记为“2”, 缺失或带型不清的记为“-”(2)显性标记中同父本带型相同的记为“1”, 同母本带型相同的记为“0”, 缺失或带型不清的记为“-”。利用卡方检验检测多态性标记是否符合(3∶1显性, 或者1∶2∶1共显性)分离比例[ 11]。用MapMaker3.0软件划分连锁群、标记各连锁群内的顺序和计算位置等, 用QTL Icimapping 3.1绘制连锁图谱(设置LOD=3.0, 最大距离37.2 cM), 可在
利用亲本DNA从561对SRAP引物组合和114对SSR引物中选出多态性好的SRAP引物组合56对和SSR引物20对, 再利用筛选出的引物对作图群体进行多态性分析。56对SRAP引物组合扩增出126个多态性标记, 平均每对引物组合能扩增出2.25个多态性标记, 标记数为1~5条不等; 20对SSR引物获得19个多态性标记, 有1对SSR引物ms0331在F2群体中未出现多态性。
卡方检验表明, 145个分子标记中(126个SRAP标记, 19个SSR标记), 有31个出现偏分离, 占标记总数的21.38%, 其中SRAP标记28个, 占SRAP总标记的22.22%; SSR标记3个, 占SSR总标记的15.78%。连锁群上共有29个标记偏分离(26个SRAP和3个SSR), 占连锁群标记数的20.56%, 主要集中在Chr.3连锁群上, 有17个偏分离标记, 其余分别分散在Chr.1、Chr.2、Chr.8和Chr.9中, 标记数各为3个。通过对照亲本基因型, 这些偏分离标记中有23个偏向父本, 均为SRAP标记, 占连锁群偏分离标记的79.31%, 其中有20个( P<0.01)表现为极显著偏离父本, 占偏父本标记的86.96%; 有6个偏向母本, 占20.69%, 3个SSR标记和3个SRAP标记, 其中3个标记( P<0.01)表现为极显著偏离母本, 占偏母本标记的50%。偏离度最大的标记是SRAP标记m2e8-4, 卡方值为25.62 (表4和表5), 偏分离标记的分离程度可见图1, 纵坐标越大, 表明偏离度越大。未发生连锁的有4个标记, 其中偏分离标记2个。
| 表1 SRAP引物序列名称 Table 1 SRAP primers and their sequences |
| 表2 56对SRAP引物组合名称 Table 2 Fifty-six pairs of SRAP primer combination |
利用MapMaker 3.0和QTL IciMapping 3.1软件, 取LOD值为3.0, 构建了一张覆盖9个连锁群(用Ch加数字表示连锁群), 包括141个标记位点的分子连锁图谱(123个SRAP和18个SSR), 其中未连锁上的有4个标记位点(3个SRAP和1个SSR), 偏分离标记用*标记(图2)。图谱覆盖基因组总长度为1399.88 cM, 标记间平均间距9.92 cM, 平均标记数15.67个。连锁群的标记位点为3~26个, 长度是15.69~237.21 cM, 其中Chr.1最长, 为237.21 cM, Chr.9最短, 为15.69 cM。每个连锁群间标记的个数和密度有较大差别, Chr.3标记数目最多, 包含26个标记; Chr.9标记数目最少, 只含3个标记; Chr.2标记密度最大, 标记平均间距为4.10 cM; Chr.7标记密度最小, 标记平均间距为19.47 cM (表6)。通过遗传学公式计算得知甜菜遗传图谱的预期长度是1499.15 cM, 框架图谱覆盖率是93.38%。在31个偏分离的标记中有29个表现出连锁遗传关系, 分布于5个不同的连锁群上, 有的形成了偏分离区域(3个或3个以上偏分离标记紧密连锁的区域, SDR)。分布在Chr.3连锁群上的SDR最多, 为3个, 分别有5个、6个、4个标记。Chr.1、Chr.9连锁群上的只有1个SDR, 且较小, 只有3个标记。
| 表3 SSR引物序列名称 Table 3 SSR primers and their sequences |
| 表4 SSR 偏分离标记卡方检验 Table 4 Chi-square test of SSR with segregation distortion |
SRAP属于显性标记, 具有操作简便、低成本、多位点以及基因组分布均匀等优点, 因而被广泛应用于植物遗传多样性分析以及遗传育种等研究中。现已成功应用在黄瓜、棉花、油菜、棉花、烟草等作物中[ 14, 15, 16, 17]。甜菜分子标记方面, SRAP只应用到遗传多样性分析, 如王茂芊等[ 18]利用SRAP分子标记对我国甜菜三大产区骨干材料的遗传多样性分析。但在甜菜图谱构建方面, 尚未见报道。本研究利用SRAP构建的连锁图只是初步的框架图, 目的是检验SRAP在甜菜作图中的可靠性。本试验表明SRAP在甜菜F2群体中的多态性较好, 适合甜菜遗传图谱的构建。而且SRAP标记在不同物种之间的使用可以相互借鉴, 将成为甜菜研究领域的一种有力工具。
| 表5 SRAP偏分离标记卡方检验 Table 5 Chi-square test of SRAP with segregation distortion |
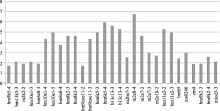 | 图1 偏分离标记分离程度柱形图横坐标: 标记名称; 纵坐标: -lg P。Abscissa: marker name; Ordinate: -lg P.Fig. 1 Separation column chart of loci with segregation distortion |
| 表6 分子标记在分子遗传图谱上的分布 Table 6 Distribution of molecular markers on genetic map |
为提高图谱构建饱和度, 本试验还采用了共显性标记SSR。SSR具有数量丰富、多态性好、呈共显性、对DNA要求不高、操作简便、重复性以及可靠性好等优点[ 19], 成为目前构建遗传连锁图谱、品种鉴定及品种选育的理想工具。现已广泛应用于玉米、大豆、花生等作物的研究中[ 20, 21, 22], 在甜菜方面, 吴则东等[ 23]经优化, 建立了适合甜菜的SSR反应体系; 王华忠等[ 24]利用9对SSR引物组合分析了49份甜菜材料的遗传多样性。2010年, 沙红等[ 25]采用RAPD和SSR分子标记技术, 构建了包含7个连锁群, 16个标记位点的甜菜遗传图谱, 覆盖418.9 cM, 定位了3个与甜菜高糖性状有关的QTL。本试验中20对SSR引物获得19个多态性标记, 有1对SSR引物ms0331在亲本间表现多态性, 在F2群体中却不分离, 这可能与群体类型、群体大小、标记类型等有关。
本研究利用SRAP和SSR两种标记相结合方法构建了一张包含141个标记, 覆盖9个连锁群的甜菜分子遗传图谱。图谱长度长的原因可能是由于虽然两种标记的多态性位点较多, 却不够密集, 如Chr.7长度为194.79 cM, 标记数只有10个, 平均间距 19.47 cM; 有些连锁群标记间距过大, 如Chr.3中标记16到17间距为56.2 cM, 第25和26标记间的间距为94.6 cM, 均表明所构建图谱密度有待于加密。本试验构建的遗传图谱包含了9个连锁群, 虽然尚未完全覆盖甜菜基因组, 但与甜菜实际连锁群个数一致, 且平均间距较小, 为9.92 cM, 是目前国内覆盖率最大最完善的一张甜菜连锁图谱, 对于我国甜菜的分子育种具有一定应用价值。目前我们正继续应用多种标记对F2群体构图, 将尽快构建出一张分布更均匀和位点密度较高的甜菜遗传连锁图谱。
偏分离遗传现象在自然界普遍存在, 甘蓝[ 26]、大豆[ 27]、高粱[ 28]等的分子遗传连锁图谱构建中都有表现。Knox等认为环境因素、非同源重组、基因转换和转座因子等也可能是引起偏分离的重要因素[ 29]。本研究F2作图群体连锁群上共有29个标记偏分离(26个SRAP和3个SSR), 占连锁群标记数的20.56%, 偏分离稍高可能与我们采用的亲本遗传距离相对较远, 或配子体选择性较强有关。偏分离标记中SRAP标记占22.22%, SSR标记占15.78%, 主要是由于显性标记比共显性标记易发生偏分离[ 30]。本试验中有的连锁群上形成了偏分离区域, 可能与配子选择关系有关, 从而导致偏分离的方向趋于一致, 如Chr.3中的SDR的标记都偏向父本, Chr.9和Chr.2上的偏分离标记都偏向母本, 表明偏分离方向相同的标记之间连锁性可能更高。偏分离标记可能会影响连锁的检验, 但偏分离的标记和正常的分离标记有相同的作图效率, 去掉偏分离标记的遗传连锁图可能会使该区域的基因不能定位在连锁图上[ 31]。本试验中就出现类似的现象, 去除偏分离标记后连锁图谱个数与实际个数不符, 反之加上偏分离标记构建出了与实际连锁群个数相等的9个连锁图谱, 表明在多态性标记较少的情况下可以考虑将偏分离标记用于作图。
本研究构建了一张包括9个连锁群, 141个标记位点的分子连锁图谱, 标记间平均间距9.07 cM。有4个标记未进入连锁群, 连锁群的标记位点数在3~26个之间, 长度为15.69~237.21 cM, 其中Chr.1最长, Chr.9最短。连锁群上有20.56%的标记出现偏分离, 主要集中在Chr.3连锁群上, 形成3个偏分离区域, 其余分散在Chr.1、Chr.2、Chr.8和Chr.9中。偏分离标记中, SRAP标记占22.22%, SSR标记占15.78%; 并有23个偏向父本, 占连锁群偏分离标记的79.31%, 6个偏向母本, 占20.69%。图谱覆盖基因组总长度为1399.88 cM, 甜菜遗传图谱的预期长度是1499.15 cM, 框架图谱覆盖率是93.38%。SRAP在甜菜F2群体中的多态性较好, 将成为甜菜图谱构建的有力工具。
| 1 |
|
| 2 |
|
| 3 |
|
| 4 |
|
| 5 |
|
| 6 |
|
| 7 |
|
| 8 |
|
| 9 |
|
| 10 |
|
| 11 |
|
| 12 |
|
| 13 |
|
| 14 |
|
| 15 |
|
| 16 |
|
| 17 |
|
| 18 |
|
| 19 |
|
| 20 |
|
| 21 |
|
| 22 |
|
| 23 |
|
| 24 |
|
| 25 |
|
| 26 |
|
| 27 |
|
| 28 |
|
| 29 |
|
| 30 |
|
| 31 |
|



