甜叶菊是我国一种重要特种经济作物, 其分子标记相关遗传背景研究甚少。本研究基于生物素与链霉亲和素的强亲和性原理, 用链霉亲和素顺磁颗粒捕捉人工合成的标记有生物素的寡核苷酸探针(AG)15, 间接筛选出含有甜叶菊基因组微卫星序列的DNA酶切片段, 将筛选得到的片段连接到pUC-T载体中, 构建甜叶菊微卫星序列的富集文库。挑取354个克隆进行菌落PCR检验, 从中筛选出158个阳性克隆进行测序。结果表明, 134个(84.81%)克隆中含有微卫星序列, 其中完美型85个(63.43%)、非完美型15个(11.19%)、复合型34个(25.38%)。根据微卫星序列共设计出71对微卫星引物, 其中62对能扩增出稳定的条带。利用24个甜叶菊品系对这62对引物的遗传多样性的分析表明, 有16个位点表现出多态性, 等位基因数为2~8个, 平均每个位点扩增得到4.5个等位基因, 多态性信息含量在0.3163~0.7595之间, 观测杂合度(
甜叶菊( Stevia rebaudiana Bertoni)又名甜菊、甜茶、甜草, 是菊科多年生草本野生植物[ 1]。南美巴拉圭等地的土著族将甜叶菊作为甜味剂食用[ 2]。甜叶菊叶片中甜菊糖甜度很高, 是蔗糖的250~300倍[ 3], 但其热值较低, 仅为蔗糖的1/300, 是一种可替代蔗糖的非常理想的甜味剂[ 4]。甜叶菊的提取物用途广泛, 可作甜味剂、药品辅料和矫味剂[ 5], 另外提取物还具有抗高血糖、抗癌和抗菌等功效[ 6]。甜叶菊是一种新型糖料栽培作物, 含有14种微量元素、32种营养成分, 它既是继蔗糖、甜菜糖后的第三种天然糖源, 又是良好的营养来源[ 7]。榨糖后的甜叶菊渣中含有多种营养成分, 如粗蛋白、粗纤维、粗脂肪及微量元素等, 可以从中提取微晶纤维素, 还可作饲料、肥料及食用菌的培养基等。甜叶菊中含有蛋白质、鞣酸、淀粉、叶绿素等, 随着研究的深入, 它的利用价值会进一步开发出来, 是一种综合开发利用经济价值较高的作物。甜叶菊适应性很广, 几乎所有从事种植业生产的农业区域都能生长, 自我国70年代从日本引种成功后, 种植面积逐年扩大, 现已在全国20个省市种植, 种植面积占全世界总种植面积的80%以上, 国内每年大约消耗甜叶菊干叶3万吨, 加之每年有上千吨甜叶菊叶片出口, 因此甜叶菊在种植应用及出口创汇等方面取得了经济效益和社会效益[ 8]。目前国内外已报道的甜叶菊研究主要集中在栽培技术及甜菊糖苷等方面, 而在分子标记与遗传分析方面的报道甚少, 在一定程度上影响了甜叶菊在遗传分析、分子辅助育种等方面的研究和应用。另外, 我国甜叶菊品种多为80年代初筛选的国外实生群体品种, 品质不高、病害严重, 迫切需要培育高产优质的优良品种, 提高原材料质量。因此需要获得更多多态性丰富、重复性较好、特异性较强的甜叶菊分子标记, 运用分子标记辅助育种为培育高产优质的优良甜叶菊品种提供保障。
微卫星(microsatellite), 又称简单重复序列(simple sequence repeat, SSR)[ 9], 是基因组中由1~6个核苷酸组成基本单位的多次重复DNA序列, 广泛且随机存在于基因组中不同位置。微卫星标记在真核生物基因组中含量丰富且随机分布, 能表现出较好的稳定性和多态性, 具符合孟德尔遗传规律、呈共显性、易操作等优点。现已广泛应用于不同物种的遗传连锁图谱构建、品种鉴定、进化分析、基因定位、遗传辅助育种、QTL分析等研究[ 10, 11, 12, 13]。但甜叶菊微卫星开发进展缓慢, 尚未开展甜叶菊基因组测序工作, 仅有少量报道关于甜叶菊EST数据库应用的研究。
磁珠富集法作为一种简单高效分离微卫星标记的方法, 其基本原理是生物素(biotin)标记重复序列的特异性探针与基因组DNA酶切片段杂交后, 由于生物素与链霉亲和素(streptavidin)具有强亲和性, 用包被有链霉亲和素的磁珠进行磁力吸附可以完成全基因组DNA酶切片段中含有重复序列片段的富集[ 14, 15]。本研究利用生物素标记的(AG)15寡核苷酸探针, 成功地构建了甜叶菊微卫星富集文库, 并筛选出具有较高多态性的微卫星标记, 为今后甜叶菊遗传多样性分析、遗传连锁图谱构建、品种鉴定、标记辅助育种、QTL分析等提供了有效的分子遗传标记。
选用具大叶的Lil/守田2号杂交种, 其命名为177-11; 另选24个甜叶菊品系用于多态性位点的筛选(表1)。表中具有相同名称的甜叶菊材料来自同一原始材料, 但在种植过程中发生了较大的性状变异, 本试验中这些具有不同性状变异、但名称相同的甜叶菊材料标以不同品系编号以示区分。将所有材料种植于浙江大学试验基地, 取幼嫩叶片保存于-80℃冷藏箱。
1.2.1 基因组DNA的提取与酶切 采用Doyle等[ 16]的方法提取甜叶菊幼嫩叶片基因组DNA, 利用限制性核酸内切酶 Sau3AI (康为世纪)酶切基因组DNA, 用PCR产物纯化试剂盒(康为世纪)纯化酶切产物。
1.2.2 接头连接与接头产物扩增 将相同浓度的单链寡核苷酸Oligo A (5°-GATCGTCGACGGTACC GAATTCT-3°)和Oligo B (5°-GTCAAGAATTCGGT ACCGTCGAC-3°)等比例混合, 使其终浓度为 10 μmol L-1, 95℃变性3 min后, 自然冷却至室温形成具有黏性末端的接头。用T4 DNA连接酶(上海生工)连接接头与酶切片段, 16℃连接过夜。以接头连接产物为模板, Oligo B为引物进行PCR扩增后, 用PCR产物纯化试剂盒(康为世纪公司)纯化产物。
1.2.3 磁珠富集微卫星序列 将0.6 mL链霉亲和素顺磁颗粒(Promega公司)用1 mL TEN100 (10 mmol L-1 Tris-HCl, 1 mmol L-1 EDTA, 100 mmol L-1 NaCl, pH 7.5)冲洗磁珠3次后, 悬于70 μL TEN100中。采用生物素标记探针(AG)15与PCR扩增产物杂交。将杂交溶液置95℃加热5 min, 迅速冷却至低于
| 表1 24份不同地区甜叶菊品系编号及表型性状 Table 1 Line number of 24 Stevia rebaudiana lines from different areas and their phenotypic traits |
Tm值5~12℃下杂交20 min, 室温放置15 min。将杂交溶液加入到制备的磁珠中, 并加入300 μL TEN100(pH 7.5), 室温放置30 min。对杂交后磁珠依次用 400 μL TEN1000 (10 mmol L-1 Tris-HCl, 1 mmol L-1 EDTA, 1 mol L-1 NaCl, pH 7.5)、400 μL 0.2×SSC+ 0.1% SDS、400 μL 1×SSC于室温下各洗涤3次, 每次5 min。最后用80 μL TE (pH 8.0)重悬磁珠, 于95℃下变性 10 min, 以磁力架固定磁珠, 吸出含有微卫星单链DNA片段的上清液备用。
1.2.4 构建克隆与克隆筛选 通过PCR将富含微卫星的单链DNA片段恢复成双链, 建立50 μL反应体系含富含微卫星单链DNA溶液5 μL、10 μmol L-1Oligo B 3 μL、10×PCR buffer 5 μL、10 mmol L-1dNTPs 1 μL、25 mmol L-1Mg2+ 3 μL、 Taq DNA Polymerase (上海生工) 7.5 U, 灭菌ddH2O补足 50 μL。反应程序为94℃ 30 s, 55℃ 40 s, 72℃ 50 s, 共33个循环; 72℃ 20 min后, 用PCR产物纯化试剂
盒(康为世纪)纯化PCR产物。采用康为世纪T载体试剂盒将PCR产物与pUC-T连接, 并转化到高效感受态细胞DH5α中, 即构建成甜叶菊微卫星富集文库。
经蓝白斑初筛选后, 挑取白色单菌落接种到含有0.1 mg mL-1 Amp的LB液体培养基中, 37℃培养过夜。以菌液为模板, 以Oligo B及微卫星核心序列(AG)8为引物进行菌液PCR筛选。用1.5%的琼脂糖凝胶电泳检测PCR产物, 将阳性克隆送到桑尼公司测序。
测序序列在chromas软件(http://www.bioon. com/Soft/Class1/Class7/200408/205.html)上进行峰图分析, 用DNAstar软件(http://www.bbioo.com/ download/58-241-1.html)查找载体和接头并将其去除, 使用SSR Hunter软件(http://www.bbioo.com/ download/58-255-1.html)查找微卫星位点, 筛选重复次数≥5的测序序列, 并记录下序列的核心序列构成。对含有微卫星序列且侧翼序列足够长的DNA序列使用Premier Primer 5.0软件(http://www.bbioo. com/download/58-166-1.html)设计引物, 由上海生工合成。
将设计好的微卫星特异性引物以甜叶菊品系177-11基因组DNA为模板, 进行PCR最佳退火温度的筛选, 用8%变性聚丙烯酰胺凝胶电泳检测扩增产物, 记录每对引物带型情况以及最佳退火温度。
利用24个甜叶菊品系对设计出的特异性微卫星引物进行多态性筛选。利用分析软件PopGene32 (http://www.seekbio.com/soft/1059.html)处理数据, 计算各位点的等位基因数( Na)、有效等位基因数( Ne)、等位基因频率、观测杂合度( Ho)、期望杂合度( He)、Hardy-Weinberg平衡卡方检验( P检验)等。根据Botstein等[ 17]计算多态性信息含量(polymorphism information content, PIC)的方法, 用PIC-CAL软件计算每个微卫星位点的 PIC值。采用NTSYS-pc2.1软件(http://ishare.iask.sina.com.cn/f/7592245.html)对24个甜叶菊品系进行聚类分析。
从(AG)15构建的富集文库中挑取354个白色单菌落, PCR检测阳性克隆, 其中共有158个(44.63%)克隆产生了2条或2条以上的扩增条带, 表明这些克隆可能含有微卫星序列。对这些阳性克隆测序, 通过SSRHunter软件搜索微卫星位点, 结果显示其中134个克隆含有微卫星序列, PCR筛选微卫星序列的准确率为84.81%, 微卫星富集率为37.85%。
在核心序列组成上, 微卫星核心序列(AG)n在134条含有微卫星的序列中含量最高为87.31%, 另
外还检测到(AC)n、(AT)n、(GATC)n、(ACGT)n、(ATGC)n、(CATG)n、(AGAAG)n等核心序列类型。在碱基构成类型上仅检测到二碱基、四碱基和五碱基的微卫星构成类型, 未发现三碱基微卫星构成类型。所有序列中, 重复次数<10的序列有96个(71.64%), 重复次数为10~20的序列有28个(20.90%), >20次的有10个(7.46%)(表2)。依据Weber[ 18]提出的分类法对开发出的微卫星统计, 结果显示完美型(perfect)微卫星序列85个(63.43%)、非完美型(imperfect)微卫星序列15个(11.19%)、混合型(compound)微卫星序列34个(25.38%)(表3)。
通过对134个含有微卫星的序列设计引物, 除侧翼太短和相似度高的序列外, 使用Premier Primer 5.0软件共设计出引物71对。通过甜叶菊品系177-11对71对引物进行PCR筛选后发现, 有62对引物能够呈现特异性条带。用24个甜叶菊品系对62对引物进行多态性分析, 共筛选到16对具有良好多态性的引物, 可作为甜叶菊分子标记使用(表4)。
通过24个甜叶菊品系对16对具有良好多态性引物的分析, 共获得72个等位基因, 等位基因数在2~8之间, 平均每个位点扩增得到4.5个等位基因, 对多态性信息含量( PIC)的分析表明, 其 PIC值介于0.3613~0.7595之间。根据Botstein等[ 17]的分类方法, 有9个位点(58.8%)的 PIC值高于0.50, 显示出较高多态性; 有7个位点(41.2%)的 PIC值介于0.25~0.50, 显示出中度多态性。分析发现, 观测杂合度( Ho)在0.2174~0.9167之间, 期望杂合度( He)在0.3555~0.8076之间。经Hardy-Weinberg平衡卡方检验, 有5个位点显著偏离Hardy-Weinberg平衡(表5)。总体表明筛选得到的16对甜叶菊微卫星引物具有良好的多态性。
| 表2 甜叶菊微卫星富集文库中序列类型比例 Table 2 Sequence type ratio of Stevia rebaudiana microsatellite enriched library (%) |
| 表3 甜叶菊微卫星重复类型 Table 3 Type of Stevia rebaudiana microsatellite repeats |
| 表4 甜叶菊微卫星分子标记信息 Table 4 Information of Stevia rebaudiana microsatellite molecular markers |
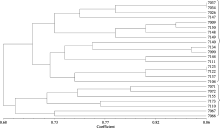
利用NTSYS-pc 2.1软件, 以遗传相似系数0.68为阈值, 将24个甜叶菊品系分为两大类(图1), 基本上是按照叶形区分的。同时, 按照材料中总糖甙含量(total glycoside, TG)以及瑞鲍迪甙A (rebaudioside A, RA)和甜菊甙(stevioside, ST)的不同, 可将全部供试材料分为TG类与RA和ST两大类型。
目前微卫星分子标记被广泛应用于遗传学和生物进化等方面, 由于微卫星在真核生物基因组中广泛且随机分布, 能表现出较好的稳定性和多态性, 另外符合孟德尔遗传规律、呈现共显性、易操作等优点, 使其近年来被广泛应用, 但不同物种中微卫星出现的频率差异较大, 在植物整个基因组中, 最为普遍的二核苷酸重复单位是(AT)n、(AC)n和(AG)n。
由于SSR分子标记为特异性引物, 所以设计引物须在物种DNA序列已知情况下进行, 在应用研究上花费较大, 一定程度上阻碍了其运用[ 19]。至今对甜叶菊基因组尚未开展测序工作, 仅在EST数据库方面有少量报道。Brandle等[ 20]通过构建甜叶菊叶片cDNA文库, 得到5548条甜叶菊EST序列, 并对其分析和注释, 但未进行微卫星标记研究。Brandle等[ 20]构建的甜叶菊EST数据库为甜叶菊微卫星标记的开发奠定了重要基础, 朱琼琼[ 21]通过搜寻甜叶菊EST数据库, 从5573个非冗余序列中筛选出含有SSR的EST序列, 获得重复次数大于5的序列261
| 表5 甜叶菊16个位点多态性分析 Table 5 Analysis of 16 polymorphic loci of Stevia rebaudiana |
个, 共设计EST-SSR引物58对; 利用EST-SSR标记对F1群体筛选分析后, 仅有6个EST-SSR标记可用于遗传图谱的构建。由于至今甜叶菊已知序列还很有限, 同时EST数据库中包含的微卫星标记有很大的局限性, 不能从已有的DNA数据库中分离出大量具有良好多态性的微卫星位点, 所以运用独特的方法来发掘甜叶菊微卫星序列是必要的。
磁珠富集法是目前开发微卫星标记的一种简便高效的方法。本试验采用磁珠富集法, 结合(AG/TC)15探针构建了甜叶菊微卫星富集文库。在应用磁珠富集微卫星的过程中, 很多因素都可能最终影响到微卫星的筛选效率[ 22], 高纯度基因组DNA是试验的前提保证, DNA浓度、接头连接效率、富集过程中杂交温度以及洗脱条件的控制都是重要影响因素[ 23], 另外磁珠平衡质量对成功开发微卫星标记也会产生重要影响[ 24]。
本试验从甜叶菊富集文库中挑取354个单菌落, 经过初次筛选后得到158个阳性克隆, 测序后发现其中134个克隆含有微卫星序列, PCR筛选微卫星序列的准确率达到84.81%, 微卫星富集效率为37.85%, 均高于Hye等[ 22]开发绿鳍马面鲀的效率(45%与21.5%), 本试验所用方法能够很好地开发甜叶菊微卫星标记。在筛选得到的序列中, 66.42%序列的核心为(AG/TC)n, 同时还检测到(AC/TG)n、(AT/TA)n、(GATC)n、(ACGT)n、(ATGC)n、(CATG)n、(AGAAG)n等核心序列类型。朱琼琼[ 21]从甜叶菊EST数据库筛选出30种不同的核心序列类型, 其中以(AG/TC)n为多, 这说明甜叶菊基因组中存在着大量不同类型的微卫星序列可供利用。但在这些序列中, 仅检测到了二、四、五核苷酸构成类型, 未检测到三核苷酸构成类型。朱立静等[ 25]在筛选西施舌微卫星标记时也发现这种状况。其原因可能是抽样测序克隆较少, 不能完全反映甜叶菊微卫星的分布情况, 也可能与检测片段来自基因组不同位置有关。
不同生物微卫星的重复次数比例各有不同, 郭昱嵩等[ 26]从勒氏笛鲷基因组文库中筛选微卫星, 重复次数分布在7~15之间的约占80.80%。贾小东等[ 27]从藏酋猴中分离微卫星, 重复次数在15~24的约占60.20%。朱琼琼[ 21]对甜叶菊EST数据库进行筛选, 微卫星重复次数在10次以下的序列占91.70%, 而介于5~6次的序列为67.30%。表明不同研究者从不同物种微卫星文库中所分离得到的微卫星结果不一样, 这可能与物种基因存在较大差别或对测序克隆的代表性有关。本试验开发出的微卫星, 初次筛选后发现28.36%的序列微卫星重复次数达到10次以上, 其中重复数最多的为AG-R6, 其重复次数达到37次; 而71.64%的序列的微卫星重复次数介于5~10之间, 高于朱琼琼[ 19]开发甜叶菊EST-SSR的效率。高焕等[ 28]研究表明微卫星重复拷贝数与多态性是有一定关系的, 较高拷贝数的位点, 其多态性较高。贾舒雯等[ 29]在研究脊尾白虾时也发现, 核心序列重复次数与微卫星位点多态性相关, 核心序列的重复次数较多的微卫星序列, 表现出高多态性。Weber[ 18]认为二碱基核心序列重复次数大于12时, 才可能表现高多态性, 本研究中有9个位点核心序列重复次数达到12次以上, 都表现出良好多态性, 而核心序列重复次数低于12次的位点也发现具有多态性。
Squirrell等[ 30]对1997—2003年微卫星标记开发的情况统计发现, 平均24.10%的引物具有多态性, 17.70%的引物无多态性, 19.50%的引物无法扩增出条带。Smulders等[ 31]在研究番茄时发现, 较长的微卫星序列变异程度更高, 从而在物种间与物种内都有良好的多态性。所以需要对开发出的引物进行多态性的筛选, 尽量筛选重复次数较多的微卫星标记。本试验筛选开发出71对引物, 得到16个多态性良好的微卫星位点, 占开发得到引物总数的22.54%, 筛选出的引物多态性比例与Squirrell等的结论相近。
等位基因数表示某位点在遗传进化过程中的遗传变异程度, 某个位点的等位基因数越多, 说明该物种在进化过程中在此位点上的遗传变异积累越大, 这有利于物种在自然界中的进化[ 32]。本试验开发的16个位点的等位基因数介于2~8之间, 平均每个位点获得等位基因数4.5个, 显示了良好的多态性, 在AG-R23与AG-J50位点仅检测到2个等位基因, 这与位点微卫星重复次数不高有很大关系。
多态性信息含量(PIC)是衡量基因变异程度的重要指标, 能反映出某个标记所包含或提供的遗传信息容量。多态性信息含量越大, 在一个群体中该座位杂合子比例会越大, 提供的遗传信息就越多[ 33]。 PIC>0.50的座位为高度多态座位, 该位点可以提供丰富的遗传信息; 0.25< PIC<0.50的座位为中度多态性座位, 该位点可提供较为合理的遗传信息; PIC<0.25的座位为低多态性座位, 该标记可提供的遗传信息较差[ 34]。本研究中16个微卫星位点PIC值在0.3613~0.7595之间, 其中9个位点( PIC>0.50)可提供丰富的遗传信息、7个位点(0.25< PIC<0.50)可提供合理的遗传信息。平均PIC值为0.5539, 高于吴勤超等[ 35]开发的黄颡鱼微卫星位点的PIC平均值0.5511。显然本实验所筛选出的16个甜叶菊微卫星位点多态性信息含量丰富, 可为甜叶菊遗传分析提供有效依据。
杂合度是反映群体遗传多样性高低的主要参数, 群体的杂合度高, 表明该群体的遗传变异大, 群体遗传多样性高。筛选出的16个甜叶菊微卫星位点的观测杂合度( Ho)和期望杂合度( He)分别介于0.2174~0.9167和0.3555~0.8076之间, 平均观测杂合度( Ho)为0.6615, 平均期望杂合度( He)为0.6261, 观测杂合度( Ho)与期望杂合度( He)较为符合, 表明甜叶菊群体遗传变异较大, 杂合度较高, 可为甜叶菊品系提供一定种质资源。
Hardy-Weinberg平衡会受人为干扰、自然选择、种群的不完全随机交配、近亲交配及迁移等多种因素的影响[ 36]。经Hardy-Weinberg平衡检验, 11个微卫星位点的 P检验值>0.05, 说明其观测杂合度和期望杂合度之间没有显著差异, 处于Hardy-Weinberg平衡( P>0.05); 其中AG-R23、AG-R27、AG-J50、AG-J63、AG-J69等5个位点偏离Hardy-Weinberg平衡( P<0.05), 这可能由于甜叶菊为多年生植物, 每生长1~2年需要移栽或重新选育, 品系间存在自然杂交或产生品系中杂交假植株, 导致基因频率变化, 偏离Hardy-Weinberg平衡。
遗传多样性是生物多样性的基础也是研究生物多样性的核心内容, 了解种内遗传变异的与环境关系, 对于遗传多样性研究有重要帮助, 进而为遗传改良提供保障。本试验将24个甜叶菊品系聚为两大类, 第一类为小叶、中叶为主的甜叶菊材料, 共17份, 其中7150与7104两份材料为大叶品系; 第二类全为甜叶菊大叶材料, 共有7份。在小叶和中叶甜叶菊的聚类中出现的两份大叶材料, 可能与产地有关, 其中大叶材料7150与小叶材料7148和7149的原始材料都是守田3号, 故其遗传相似度较高。另外材料7037与7034, 7123与7122有很高的相似度, 猜测可能是同一个品系。从来源地分析发现, 部分符合地区一致性, 能聚类到一组, 有个别材料通过来源地聚类发生偏离, 如材料7009与守田3号材料, 这可能由于一些材料受多年的自然选择以及地域环境的影响, 发生了一定程度的变异。这种变异可增加甜叶菊品系的多样性, 为甜叶菊品系的改良与选育提供种质资源。
根据24个甜叶菊品系在RA、ST、TG含量上的不同, 可将材料分成以TG为主的大类与以RA和ST为主的大类。以TG为主大类为总糖甙含量大于13%, 其中5份表现为TG类型, 仅有7155与7110两份材料分别表现为RA与ST类型。以RA和ST为主大类为RA或ST含量均大于7%的材料, 总共有17份材料, 有14份表现出RA类型, 2份表现出ST类型, 仅有7090材料表现为TG类型。通过糖甙含量分类可以发现, RA、ST、TG含量与叶型可能有着一定的相关性, 小叶、中叶材料多为RA和ST含量较高的品系, 大叶材料多是TG含量较高的品系。这一聚类结果一方面可能与选用材料本身局限性相关, 所用材料尚不能完全涵盖所有材料表型性状; 另一方面本实验开发的16个微卫星标记全为AG系列标记, 在甜叶菊基因组中还存在着大量其他类型的微卫星标记, 运用这16个标记进行品系聚类的代表性有限, 造成了聚类基本按照叶型区分品系的现象, 尚需进一步搜索更多的甜叶菊材料并进一步开发更多甜叶菊分子标记进行相关研究。
构建了甜叶菊全基因组微卫星富集文库, 提出了一种适合甜叶菊微卫星开发的新方法。通过富集文库筛选, 选取合适的富集序列设计引物, 共合成71对微卫星引物并筛选其多态性, 获得特异性强、多态性良好的微卫星位点16个, 为今后揭示甜叶菊品系多样性、构建遗传图谱、分子标记辅助育种、QTL定位等研究工作提供了有效的遗传基础。基于16个微卫星位点多态性信息构建了24个品系的聚类图, 初步揭示了各品系间的遗传关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|