突变体
A
叶片衰老是植物叶片发育的最后阶段, 是一种细胞死亡的过程, 也是营养物质从衰老部位被重新调运到种子和成熟组织等贮藏器官的过程, 所以叶片衰老是植物正常生长发育的必然结果[ 1]。然而, 叶片是水稻最重要的光合器官, 据理论推算, 水稻籽粒灌浆的60%~80%营养物质来自叶片的光合作用[ 2]。因此水稻抽穗到灌浆期间功能叶的过早衰老, 将影响水稻灌浆速度, 进而导致产量和品质下降[ 3]。研究表明, 若在水稻籽粒灌浆后期设法延长功能叶片1 d 的寿命, 稻谷产量可以增加1%~2%左右[ 4]。
水稻叶片衰老是一个复杂的过程, 最主要的表现为叶片由绿变黄, 而内部的生理生化变化主要表现为叶绿素含量降低, 蛋白质降解, 游离氨基酸积累, 各种水解酶含量和活性提高, 糖类物质、RNA和DNA含量下降, 以及细胞分裂素含量减少, 而诱导衰老和成熟的脱落酸含量增加。究其衰老的原因, 有持续黑暗、涝害、干旱、盐害、极端温度和紫外线等[ 1]外界环境的影响, 也受各种内源激素和大量衰老相关基因(SAGs)[ 1, 5]精细调控(http://www.eplan-tsenescence.org/)等内部因素影响, 。随着现代生物技术的迅速发展, 迄今已从水稻中克隆并经遗传转化验证或T-DNA插入验证的叶片衰老相关基因有27个, 包括 Osl2[ 6]、 Osh 69[ 7]、 SGR[ 8, 9]、 NOL1[ 10]、 NYC1[ 11]、 OsDOS[ 12] 、 YGL1[ 13]、 A12[ 14]、 SPL28[ 15]、 NYC3[ 16]、 OsRCCR1[ 17]、 OsPAO[ 17]、 RLS1[ 18]、 OsRab7B3[ 19]、 OsABC1-2[ 20]、 OsLMS[ 21]、 SUB1A[ 22]、 OsTZF1[ 23]、 GDCH[ 24]、 NYC4[ 25]、 SGRL[ 26]、 OsSIK2[ 27]、 OsNAP[ 28]、 Cga1[ 29]、 OsFBK12[ 30]、 OsSAMS1[ 30]和 OsSAG12- 1[ 31]等。
本课题组利用60Co辐射诱变中籼品种自选1号, 获得了1个隐性突变的叶片早衰突变体, 其叶片早衰表型在水稻分蘖期(约4~5片叶龄时)就开始显现, 之后随着水稻的生长发育及其叶龄数的增加, 各叶位的新叶在其完全展开后的10~15 d也相继出现早衰症状, 至抽穗开花时, 所有叶片(未枯死的功能叶)均出现不同程度的早衰表型。此外, 经本课题组初步鉴定, 在盐胁迫下, 该突变体的叶片和野生型相比明显卷曲, 同时株高明显变矮, 表现为盐敏感, 但是根系和生物学产量没有明显的变化。因此将该突变体命名为 o sles( O ryza s ativa leaf early-senescence and salt-sensitive)。本文对该突变体叶片早衰的基本表型、生理变化及基因定位进行研究, 以期为进一步揭示其早衰的分子和生理机制提供理论基础。
从60Co辐射诱变持久高抗稻瘟病材料自选1号(来源于恩施农业科学院水稻研究所)后代中获得叶片早衰及盐敏感突变体 osles, 经杭州和海南连续5代回交和自交, 获得突变性状稳定的株系。之后, 以 osles作母本, 分别与自选1号和粳稻材料02428杂交, 获得F1并自交获得F2, 对2个F2群体进行遗传分析, 并利用 osles/02428的F2群体进行基因定位。其中, 02428是一个带有广亲和基因的常规粳稻品系, 叶色正常浓绿。
2011年将 osles突变体及其野生对照自选1号、2012年将上述2个F2群体及其亲本分小区种植于浙江省农业科学院实验农场(浙江杭州)。每材料各2次重复, 每小区4行, 每行8株, 田间随机排列; 2个F2群体的田间种植不设重复, 单本插植。2年试验的播期、栽培管理方式相同, 6月1日播种, 6月27日移栽, 栽插密度为17 cm × 22 cm, 大田常规水作管理, 及时防治病虫和杂草。试验地前茬为大麦, 地力中等偏上。在水稻播种后60 d至分蘖期(主茎处于约7~8片叶龄时), 对 osles突变体与其野生对照材料分不同叶片取样, 冷冻保存, 测定有关生理指标; 与此同时, 对2个F2群体材料分单株观察其早衰表型并取样, 提取DNA, 用于后续遗传和基因定位分析。在水稻成熟时, 取每个材料20株, 调查株高、单株有效穗数、穗长、穗粒数、千粒重、结实率、粒长和粒宽等主要农艺性状。
选取成熟种子发芽后7 d且株高和根长基本一致的幼苗在内含100 mmol L-1 NaCl的Yoshida营养液中培养1周[ 32], 而后在Yoshida营养液中恢复生长5 d。培养条件为白天28℃、光照16 h; 夜晚25℃、黑暗8 h。分别在处理7 d后和恢复生长5 d后调查株高、根长、整株干重并观察照相。
分别选取12株播种60 d后的野生型和突变体主茎倒一、倒二、倒三全展叶测定叶绿素含量、可溶性蛋白含量、MDA含量、SOD含量和POD含量。用80%丙酮提取叶绿素, 测定波长663 nm和646 nm下的吸光值; 用考马斯亮蓝G-250染色法测定可溶性蛋白含量; 参考《植物生理学实验技术》[ 33]测定各生理指标。采用日本岛津公司UV-2450紫外分光光度计, 每个指标测定3个重复。
在大田分单株剪取F2定位群体中具有突变体性状的早衰单株、双亲以及F2群体中10株正常植株的叶片, 采用CTAB法提取基因组总DNA[ 34]。同时, 分别选取10株正常植株和10株具有突变体性状的单株的等量DNA混合, 构建正常基因池和突变基因池, 用于 OsLES基因连锁标记筛选。
利用本实验室保存的均匀分布于水稻12条染色体的500对SSR标记, 其引物序列来自Gramene数据库(http://www.gramene.org/), 以及Shen等[ 35]开发的50对InDel标记对突变体和粳稻品种02428进行多态性筛选, 用所筛选到的多态性标记分别对正常池和突变池进行连锁性分析, 获得连锁性标记并进一步分析所有F2群体中突变极端单株。PCR体系20 μL, 含0.8 U Taq, 1×PCR buffer (Mg2+Plus), 1 mmol L-1 dNTP Mixture, 50 ng DNA, 上下游引物各0.25 μmol L-1。PCR程序为95℃预变性5 min; 94℃ 30 s, 55℃ 30 s, 72℃ 30 s, 共33个循环; 72℃ 10 min。PCR产物经8%非变性聚丙烯酰胺凝胶电泳, 快速银染后观察[ 36]。
从1276株的 osles/02428 F2群体中获得315个具有早衰性状的单株组成基因定位群体, 利用上述筛选到的基因连锁分子标记进行定位群体的遗传分析, 获得每个标记的重组交换单株数。同时在Gramene数据库查找基因连锁分子标记在染色体上的物理位置以确定其排列顺序, 结合每个标记的重组交换单株数确定 OsLES基因与连锁标记的顺序, 构建基因定位图谱。
早衰性状始于 osles突变体植株的分蘖期(图1-A), 主要表现为叶片的叶尖和叶边缘变黄, 叶尖略卷曲, 随后叶缘出现线状黄化条纹和斑点, 并逐步扩展到叶片内侧, 最后发展为枯斑和条状枯纹(图1-B), 直到整个叶片枯死。在整个分蘖期, 仅有一叶一心为正常叶, 其他叶片均表现一定程度衰老症状(图1-A和B)。而到抽穗开花期时, 所有叶片均出现不同程度的早衰症状(图1-C)。由于叶片的过早衰老, 导致 osles突变体的主要农艺性状, 如株高、有效穗、每穗粒数、千粒重、结实率、籽粒的宽度均显著低于对照(表1), 其中株高、每穗粒数和结实率下降最多, 分别为对照的70.8%、56.5%和45.0%。
| 表1 os les突变体及其野生型(WT)的主要农艺性状 Table 1 Main agronomic traits of os les mutant and the wild type (WT) plants |
在100 mmol L-1 NaCl胁迫下处理7 d后, osles突变体叶片卷曲, 叶尖部分枯死(图2-A)。同时株高和生物学产量比对照分别下降10.6%和29.0% (图2-A), 而7 d植株净生长高度、根净生长长度与净生物学产量则分别比对照下降13.7%、9.9%和37.5% (表2)。恢复生长5 d后, 有12.4%的突变体萎焉枯死, 而且处理后的突变体生长速度明显慢于处理后的野生型对照(图2-b), 突变体株高和生物学产量分别比对照少43.5%和53.8%, 而5 d植株净生长高度、根净生长长度与净生物学产量则分别比对照下降81.0%、11.2%和61.6% (表2)。
| 表2 100 mmol L-1 NaCl盐胁迫下 os les突变体及其野生型(WT)的基本特性 Table 2 Basic characteristics of os les mutant and the wild type (WT) plants under 100 mmol L-1 NaCl stress |
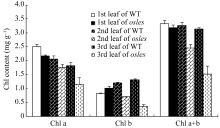
2.3.1 叶绿素含量分析 分蘖期 os les倒一叶的叶绿素a含量比对照低13.4%, 而叶绿素b则高24.4%, 但总的叶绿素含量差异不显著(图3-a)。与野生型相比, 倒二和倒三叶的叶绿素a含量分别下降14.8%和36.8%, 叶绿素b含量分别下降41.1%和72.6%, 叶绿素总量分别下降24.4%和51.7%。
2.3.2 抗氧化酶SOD和POD活性分析 由图4-A和B可见, 相对 osles突变体倒一叶, 其倒二和倒三叶的SOD活性分别上升18.7%和29.1% (图4-A), POD活性分别上升173.6%和240.3% (图4-B)。而对
照倒一、倒二和倒三叶之间的SOD和POD活性差异不显著(图4-A和B)。相对于对照, osles突变体倒一叶SOD含量下降0.6%, 倒二和倒三叶的SOD含量分别上升14.9%和21.4%, 而POD含量则分别上升34.7%、279.5%和322.2% (图4-A和B)。
2.3.3 MDA和可溶性蛋白含量分析 作为衡量植物体内细胞膜、脂蛋白以及其他含脂质结构损害程度的重要指标MDA, osles突变体均比对照高, 且
倒二和倒三叶升高显著, 倒一、倒二和倒三叶分别增加10.6%、19.9%和19.9% (图4-C)。同时, 由于叶片不同程度的衰老, 导致 osles突变体倒一至倒三叶叶片中的可溶性蛋白含量依次降低, 而对照倒一和倒二叶可溶性蛋白含量差异不显著(图4-D)。当然, 相对于对照的可溶性蛋白含量, osles突变体倒一和倒二叶分别增加24.8%和17.7%, 而倒三叶则下降12.9% (图4-D)。
osles/自选1号和 osles/02428两个杂交F1单株均表现正常, 说明该突变性状是由隐性位点控制。在 osles/自选1号的698个F2单株中, 有173株表现早衰性状, 525株表现正常性状, 正常单株∶早衰单株=3∶1 [χ2=0.0076<3.84(χ2(0.05,1))]; 在 osles/02428的1276个F2单株中, 正常单株为961株, 早衰单株为315株, 正常单株∶早衰单株=3∶1 [χ2=0.051<3.84 (χ2(0.05, 1)]。这2个群体的结果均符合孟德尔单基因隐性遗传的分离比例, 说明 osles突变体受1对单隐性核基因控制。
选择均匀分布于水稻12条染色体上的500对SSR标记和50对InDel标记, 逐条对 osles突变体与02428进行多态性筛选, 结果表明178对引物在两亲本之间具有多态性, 多态性比例为32.4%。然后用这178对标记分别对所构建的2个基因池进行BSA分析, 结果发现水稻第6染色体长臂上的3个SSR标记RM20361、RM20491和RM3430, 以及1个InDel标记R6M44在突变池和正常池间具有明显偏分离。随后, 用以上4个标记分别对315个F2突变表型单株进行基因型分析, 结果表明RM20361、R6M44、RM20491和RM3430的交换单株分别为58、36、24和4株(图5)。结合每个分子标记的基因型确定4个标记与 OsLES基因在第6染色体的物理排列位置为RM20361-R6M44-RM20491- osles-RM3430(图5)。为了进一步定位 OsLES基因, 于是在RM20491和RM3430之间设计20对SSR标记或InDel标记, 其中6对标记在亲本间有多态性(表3), 利用这6对标记对F2早衰群体进行基因连锁分析, 最终把 OsLES基因定位在InDel标记IN6-005769-11/12和SSR标记RM20547之间, 物理距离为210 kb, 横跨OSJNBa0051O02和OSJNBb0065C04两个BAC, 并且与SSR标记RM005769-15/16和InDel标记IN6-005769-3/4共分离(图5)。其间包含30个开放阅读框(ORF), 它们分别编码2个反转座子、3个转座子、8个花粉过敏源蛋白、1个ATP合成酶、1个泛素结合酶、1个DNA结合蛋白、1个B-box锌指蛋白、1个含bZIP转录因子结构域的蛋白、1个tRNA假尿苷合成酶、1个蛋白激酶APK1B、1个白喉酰胺生物合成蛋白、1个网格蛋白装配蛋白、1个DNA光解酶结构域的FAD结合域蛋白、1个含rabGAP/ TBC结构域的蛋白、1个STRUBBELIG受体蛋白前体和4个功能未知蛋白(表4)。
| 表3 OsLES基因连锁的SSR标记及其引物序列 Table 3 SSR markers and primer sequences linked to OsLES gene |
| 表4 定位区间内的基因及功能注释 Table 4 Gene names and their annotations in the target interval |
叶片衰老是植物叶片发育的最后阶段, 受多种因素影响。自然条件下, 叶片衰老的主要外部形态和生理表现为由绿变黄直至枯死, 而在细胞结构上最早且最明显的变化是含有叶片70%蛋白质的叶绿体解体, 因此叶绿素含量的降低是叶片衰老的最重要标记之一[ 1]。当然, 伴随叶绿体的解体, 不仅蛋白合成减少, 而且蛋白水解酶活性还会增加, 致使叶片中的Rubisco和叶绿素a/b结合蛋白等蛋白不断被降解, 因此叶片中可溶性蛋白含量的降低也是衡量叶片衰老程度的重要指标之一[ 1]。同时细胞膜系统的膜质组分发生变化, 导致膜的透性增大, 膜的完整性也不同程度受到破坏, 致使细胞内的物质渗出而加速失水[ 1]。
本研究中的 osles突变体从分蘖期开始, 叶尖和叶边缘变黄并伴有红褐色斑点(图1), 叶绿素含量急剧下降, 且相对于其倒一、倒二和倒三叶中的叶绿素a含量分别下降19.2%和46.9%, 叶绿素b分别下降30.8%和64.9% (图3), 说明在该突变体中, 叶绿素b比叶绿素a下降更快, 这可能是由于叶绿素b在叶绿素b还原酶作用下转换成叶绿素a, 而不是在脱镁螯合酶(Mg-dechelatase)和叶绿素b还原酶等酶的作用下降解成脱镁叶绿酸a[ 37]。至于倒一叶中, 突变体叶绿素a含量比对照低, 而叶绿素b更高, 可能是基因突变, 或与诸如 OsDET1存在一定关联所致, 因为 OsDET1的突变将导致水稻中的叶绿素b含量增加1倍[ 38]。
研究已证实, 伴随叶片衰老和叶绿素含量的急速下降, 衰老叶片中的活性氧急剧增加且不能及时被清除, 导致MDA含量快速增加[ 39], 而可溶性蛋白含量则显著下降。本研究中, 相对于 osles突变体倒一叶, 其倒二和倒三叶的MDA含量显著升高(图4-C), 而可溶性蛋白含量则显著下降(图4-D), 该结果预示着倒二和倒三叶已经出现了不同程度的衰老, 这也与图1-b所示的倒二叶轻度衰老而倒三叶则已明显衰老的表型一致。当然, 在图4-D中, 突变体倒一和倒二叶中的可溶性蛋白含量显著高于对照, 究其原因, 其一可能是基因的突变导致突变体自身可溶性蛋白含量显著高于对照, 如同水稻抽穗后期早衰突变体TM313的可溶性蛋白含量就显著高于对照9311[ 40]; 其二, 尽管在 osles突变体和野生型水稻种植过程中没有额外增加盐胁迫, 但田间条件是复杂的, 因此适合于野生型水稻的条件也许足以成为盐敏感突变体的胁迫条件, 而可溶性蛋白是植物体内亲水性较强的有机物, 它能明显增加细胞的持水力, 束缚更多的水分, 因此胁迫条件下植物可溶性蛋白的含量会先升高后降低[ 41, 42]。此外, 作为可溶性蛋白的抗氧化酶SOD, 在细胞内担负着清除活性氧或其他过氧化物自由基, 保护膜系统的重任; 虽然POD在植物体内作用并非专一, 但它的部分同工酶依然与膜脂过氧化息息相关, 是细胞防御活性氧毒害酶系统的重要成员之一, 不仅参与了清除过氧化物, 还能使MDA转变为正常的脂肪酸而降低MDA对植物细胞的损害。因此 osles突变体叶片衰老中SOD和POD活性显著上升(图4-A和B), 此研究结果与杜青等[ 43]的研究结果一致。
研究表明, DNA结合蛋白可以与转录因子的启动子部分区域结合, 影响转录因子的表达, 进而调控细胞的程序化死亡而使组织器官表现早衰, 如在黑暗诱导的衰老叶片中, 水稻的DNA结合蛋白基因OsWRKY80上调表达[ 44]。而锌指蛋白是一类具有手指状结构域的转录因子, 对基因调控起重要的作用, 利用RNAi干涉技术研究编码CCCH-型锌指蛋白基因 OsDos显示, 转基因水稻生长到籽粒充实期时, 大部分叶片均已衰老[ 12]; 而RNAi干扰水稻另一个锌指蛋白基因 OsTZF1后, 转基因水稻不仅表现叶片早衰, 而且对250 mmol L-1盐敏感[ 23]。当然, 蛋白质的降解也是叶片衰老过程中的内在表现形式, 蛋白质的泛素化也会引起本身的特异性降解, 如水稻泛素结合蛋白基因 LTN1的功能缺失会导致叶片黄化[ 45]。此外, 研究显示, 其他转录因子如NAC 、WRKY和MYB, 以及蛋白激酶/磷酸酶, 酶调节因子, 信号因子, 抗逆胁迫因子, 代谢加工降解相关基因, 生物合成相关基因和转运相关基因均参与植物叶片衰老进程[ 5], 其中, 拟南芥NAC转录因子 AtMYBL和MYB转录因子 VIN2均涉及到延缓叶片衰老和植株的耐盐特性[ 46, 47]。在本研究的精细定位区间, 尚没有衰老相关的基因被定位或克隆, 而与报道相关的导致水稻叶片衰老的基因仅有一个DNA结合蛋白基因(LOC_Os06g45110)、一个锌指蛋白(LOC_Os06g 45040)和一个泛素结合蛋白基因(LOC_Os06g 45000), 但对这3个基因测序后发现, 均没有序列差异。因此, OsLES基因很可能是其中的bZIP转录因子、蛋白激酶基因、白喉酰胺生物合成蛋白基因或rabGAP/TBC功能域的蛋白基因。总之, OsLES基因是水稻中的一个全新的不仅与衰老相关, 还与盐胁迫相关的基因, 最后的候选基因需要我们进一步分析方能确定。
osles是一个叶片早衰且对盐敏感的突变体。除倒一叶外, 分蘖期突变体叶片中的叶绿素总量显著下降。与野生型相比, 其倒二和倒三叶的SOD活性、POD活性和MDA含量则显著上升。而突变体和野生型3片叶中的可溶性蛋白含量依次下降, 但突变体的倒一和倒二叶中的可溶性蛋白含量显著高于野生型, 而倒三叶则相反。 osles突变性状受1对隐性核基因控制。将该基因定位于第6染色体长臂的IN6-005769-11/12和RM20547两个标记的210 kb区间, 为进一步克隆该基因并揭示叶片的早衰分子生理机理奠定了基础。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|