利用57对小豆SSR标记和31对绿豆SSR标记, 用5份日本材料作对照, 对249份中国小豆种质进行遗传差异、群体结构和连锁不平衡(LD)分析。结果表明, 共检测到630个等位变异, SSR位点等位变异数在2~17之间, 遗传多样性指数范围为0.024~0.898, 平均为0.574。15个不同地理来源群体间表现出显著的遗传多样性差异, 其中中国云南最高, 河北和天津最低。聚类分析将254份材料划分为3个类群, 在一定程度上和地理生态环境相关。LD分析显示和其他作物相比, 小豆LD衰减距离较短, 最大衰减距离为5.8 cM (
A total of 249 Chinese adzuki bean accessions were analyzed with 57 adzuki bean SSR markers and 31 mungbean SSR markers to ascertain genetic diversity, population structure and linkage disequilibrium (LD) with five Japanese materials as the contrast. The results indicated that 630 alleles were detected with 2-17 alleles per locus and a mean genetic diversity index of 0.574, ranging from 0.024 to 0.898. There were significant differences among 15 populations of adzuki bean resources in genetic diversity from diversed geographic origins, with the highest in Yunnan and the lowest in Hebei and Tianjin. The 254 adzuki bean accessions could be divided into three subgroups based on STRUCTURE and NJ cluster. The population structure derived from them was positively correlated to some extent with the geographic eco-type. LD analysis revealed that there was a shorter LD decay distance in adzuki bean than that in other crops. The maximum LD decay distance, estimated by curvilinear regression, was 5.8 cM (
小豆( Vigna angularis)主要分布在中国、日本、韩国和喜马拉雅山麓[ 1], 是我国主要食用豆类作物。中国主产区为东北、华北、黄河中游、江淮下游, 最佳生产区是华北及江淮流域[ 2]。随着人民生活水平的提高, 国内外市场对小豆的需求量大幅增加, 对小豆遗传育种的研究有待深入。
以往对小豆种质资源的研究主要集中于表型分析与鉴定, 近年来, 紧密联系在目标基因上的DNA分子标记可用于标记辅助选择, 也可用于调查种质库中诸如栽培种和近缘种间的遗传多样性水平[ 3, 4], 因此被广泛应用于小豆遗传研究。其中, SSR标记因其多态性丰富、重复性好、具共显性及在基因组中表现出相当均匀的分布而被最广泛应用[ 5]。
遗传多样性是推动作物育种发展的重要因素。金文林[ 2]报道20世纪50年代初在全国范围内进行小豆地方品种筛选, 从20世纪80年代开始利用杂交改良品种; 而在当代种质育种进程中发现的遗传差异程度可能会间接反应出未来栽培种可以实现的遗传进展水平[ 6]。因此小豆遗传多样性的评估可以有效促进育种进程中遗传资源的充分利用。
连锁不平衡(LD), 也被称为等位基因关联, 对于鉴定关联农艺性状的基因区域越来越重要[ 7, 8, 9]。而对于一个核心种质的群体, 其大小、交配和混合模式对LD水平都有很大影响[ 7]。事实上, 评价种质资源群体中的群体结构可以正确解释并确认功能和分子多样性间的关联[ 10, 11]。评估群体结构有几种方式, 并且都会把其信息整合到关联分析中, 基于Bayes框架的Structure分析方法[ 12, 13], 邻结法聚类分析的方法[ 14, 15]可从分子标记数据或亲缘关系信息得到遗传距离矩阵。
由于小豆中适合于关联分析的单位点分子标记数量较少, 对小豆群体结构和LD情况的研究报道还很少。本研究利用分布于小豆全基因组的88个
SSR标记位点分析取自核心种质的254份小豆资源, 利用贝氏定理和分层聚类法(NJ法)分析群体结构, 并讨论其LD模式和一些小豆基因组特殊的特性。而群体结构和LD衰减水平的信息可以在之后的综合关联作图的研究中展开。
参试的254份小豆资源均来自中国农业科学院作物科学研究所, 是依据地理来源随机选取的小豆核心样本。其中覆盖中国15个省份的249份资源, 包括安徽8份、北京17份、河北20份、河南20份、黑龙江20份、湖北23份、吉林30份、江苏9份、辽宁14份、内蒙古21份、山西22份、陕西16份、天津17份和云南12份, 能较好代表传统栽培区栽培资源的分布以及小豆种内的遗传多样水平; 另选5份日本小豆作对照。部分相关品种和资源名称见表1。
| 表1 部分相关品种和资源名称 Table 1 Some related adzuki bean collections and resource name |
于2012年5月底在昌平区种子站温室播种参试材料, 温室温度25~30℃, 出苗后20 d左右采集5株幼苗展开的嫩叶, 在液氮中研磨成细粉; 用改良CTAB法提取基因组DNA[ 16], 得到254份小豆纯化样品, 经紫外分光光度计测定浓度后稀释标定到 25 ng μL-1, 放-20℃冰箱备用。
所用SSR标记具有多种来源, 前缀含X的197对小豆SSR标记引自日本, 几乎覆盖全基因组[ 17, 18], 前缀含P的是由本课题开发的约3000对绿豆SSR标记[ 19]。
筛选后共得到57对小豆和31份绿豆多态性良好的引物。PCR总体积10 μL, 其中含40 ng基因组DNA, 1× Taq buffer (10 mmol L-1Tris-HCl, pH 8.8; 10 mmol L-1KCl; 10 mmol L-1 (NH4)2SO4; 1.5 mmol L-1 MgCl2; 0.1% Triton X-100), 1 mmol L-1dNTPs, 上下游引物各0.25 μmol L-1和1 U Taq DNA聚合酶。反应程序为95℃预变性5 min; 95℃变性30 s, 51~60℃退火45 s, 72℃延伸45 s, 32~35个循环, 最后72℃延伸5 min。整个反应在东胜EDC-810 PCR扩增仪上进行。用8%的聚丙烯酰胺非变性凝胶电泳分离扩增产物, 银染法染色。
带型记录方式是根据SSR标记长于揭示等位基因的特点, 配合相应统计软件对SSR原始数据格式特殊要求确定的[ 20, 21, 22]。因此, 记载同一对SSR引物扩增条带(等位变异)在各参试品种中有或无, 若无记为0, 若有, 则按其分子量从小到大分别记为1、2、3、……。
1.4.1 遗传多样性估计 在Popgene中得到比较资源群体遗传多样性差异显著性的基因流系数(Nm), 遗传距离(GD), 遗传一致度(GI), Shannon’s信息指数(I)和群体间遗传分化系数(Fst)[ 22]; 在Power Marker Ver. 3.25中得到不同群体间和群体内某一位点的等位变异数及等位变异频率, 有效等位基因数, 每个SSR位点的多态性信息含量(PIC)[ 23]。
1.4.2 群体遗传结构分析 利用Structure软件完成254份小豆资源群体结构剖析及参试材料的群体划分, 若软件输出的后验概率值[ln P( D)]随亚群数的增大而增大, 则根据Evanno等[ 24]提出的方法, 利用连续2个ln P( D)间的变化速率(Δ K)确定合适的亚群数和最好的输出结果。计算每个品种源于特定亚群的概率 Q值, 当某亚群的 Q值最大时, 相应品种被划分到该类群[ 13]。
构建系统发生树时, 若在进化分支上发生趋异的次数不同, 用UPGMA算法可能会给出错误的聚类图, 因此本研究用PowerMarker Ver.3.25软件以邻结法(NJ)计算种质间Nei’s遗传距离, 进行聚类分析并用FigTree作树状聚类图[ 25]。
1.4.3 LD水平估计 利用TASSEL 2.1[ 26]软件衡量SSR标记两两间的连锁不平衡水平。分别用总SSR标记、同一染色体上连锁的SSR标记和不同染色体上的非连锁的SSR标记计算两两之间的LD相关系数 R2, 来分析遗传连锁和LD的关系。仅那些已知染色体信息的SSR标记用于LD估计[ 17]。如果 P<0.001则认为位点间的连锁不平衡显著。用非线性回归模型模拟出LD值随遗传距离衰减的趋势线。
88对SSR引物在254份小豆材料中共检测到630个等位变异(表2)。SSR位点等位变异数从2~ 17不等, 平均每SSR位点检测到7.15个等位变异, 其中检测等位变异数最多的是位于LG5的X101和位于LG10的X174。稀有等位变异(指分布频率小于5%的等位变异)有336个, 占总等位变异数的50.90%, 说明在所选微核心种质中存在很多新的等位基因。
基因多样性指数的变异范围是从0.0238~0.8984, 平均为0.5744, 多态性信息含量PIC值的变异范围0.0236~0.8904, 平均为0.5392, 二者最高的都是位于LG10的X174。
| 表2 小豆种质88个SSR位点的多样性信息 Table 2 Allelic diversity of adzuki bean collections at 88 whole genome SSR loci |
遗传多样性指数和等位基因数目在给定范围内通常呈显著正线性相关。简单相关分析(图1)表明遗传多样性指数和等位基因数显著相关( r=0.711, P<0.001)。以等位基因数为独立变量 x, 基因多样性指数为因变量 y, 构成曲线回归方程, y= 0.352 ln ( x)-0.078 (1< x<18), R2=0.648。
栽培小豆资源群的参试份数、检测等位变异数、基因多样性指数和多态性信息含量(表3), 以中国河南、云南最高, 中国湖北、山西、陕西其次, 河北和天津最低。日本小豆资源等位变异数(4.045)小于中国的平均值(16.850), 基因多样性指数(0.450)低于中国平均值(0.472), 多态信息含量(0.395)也低于中国平均值(0.428)。
利用88个SSR标记和Structure软件对254份材料进行群体遗传结构分析表明, 后验概率[ln P( D)
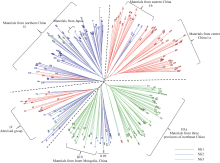
值随亚群数的增加而增加, 即采用基于Δ K的最大似然估计确定适宜亚群数, 参试材料在 K = 3时Δ K出现峰值, 即所分析的小豆种质从遗传结构上可被分为3类血缘(图2)。由此, 用Structure软件计算出每个品种归属于3亚群的后验概率 Q值, 若设置每个品种分到各亚群概率的临界值最大, 则有72份材料被分到亚群(SG)1, 85份材料被分到亚群2, 97份材料被分到亚群3; 将概率临界值设置为大于0.6, 则有66份被分到亚群1, 69份被分到亚群2, 80份被分到亚群3, 39份被分到混合组(AD)。进一步研究Structure软件分析得到的亚群和地理生态型间的关系发现(表4), SG1主要包括中国安徽、湖北和云南所有材料及江苏、陕西的大部分材料SG2主要包括中国黑龙江、吉林、辽宁和内蒙古的大部分材料, SG3主要包括中国北京、河北、山西和天津的大部分材料, 而混合组则包括了一些省份的部分材料, 其中中国河南居多。
| 表3 88对SSR引物在16个小豆群体中检测到的遗传多样性参数 Table 3 Estimates of genetic parameters for 16 populations of adzuki bean based on 88 SSR loci |
基于NJ法聚类分析的结果, 在遗传距离约为0.110时254份材料明显被分为4类(图3)。同一省份内的种质在聚类图上大多成簇状分布, 但较难完全聚在一起。其系统树图显示从遗传角度来讲亚群I和II遗传关系更近构成姐妹群, 与亚群Ⅲ构成外类群, 亚群IV与其遗传距离最远。当遗传距离约为0.133时亚群I和II被聚为一类, 当遗传距离约为0.142时所有材料被聚为一组。与Structure软件分群结果相比, 居群I-a和I-b对应Structure软件中的SGI, 主要为中国江苏、安徽、陕西和湖北材料, 也有云南省材料; 居群II对应SGIII, 主要为中国北京、河北、天津和山西材料和日本对照材料; 居群Ⅲ对应SGII, 主要为中国东北三省及内蒙古材料; 居群IV包括大部分中国河南省材料(65.0%)和其他省的少数材料。
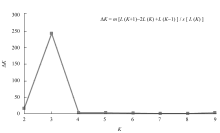 | 图2 通过Δ K值估计从1到10最合理的群体结构Fig. 2 Structure estimation of populations for K ranging from one to ten by delta K-values (Δ K) |
| 表4 以Structure分析小豆群体与种质地理来源的分布 Table 4 Geographical distribution of adzuki bean within populations divided by Structure |
各省份间的聚类分析结果与Structure软件分群和个体间聚类分群结果基本一致(图4)。亚群I由中国江苏、安徽、陕西、湖北和云南的资源组成, 其中华东两省江苏和安徽、华中两省陕西和湖北遗传距离最小, 但四省均和云南省遗传距离较大, 反映了虽然南方有共同的光照气候条件, 但云南省种质间遗传变异显然更丰富, 河南省材料也被分到该居群, 原因有待进一步分析; 亚群II由中国天津、中国河北、中国陕西、中国北京和日本材料组成, 其中中国华北4个主产省份中天津与河北间的遗传距离最小, 与山西、北京的遗传距离也很小, 显示生态气候条件相似的省份栽培小豆资源群间的亲缘关系很近或遗传差异很小, 但日本材料也被分到该类群, 原因有待进一步分析; 亚群Ⅲ由中国内蒙古、辽宁、吉林和黑龙江的资源组成, 其中内蒙古与辽宁、吉林和黑龙江遗传距离最小, 反映了东北三省和内蒙古较为特殊的生境气候条件。
利用57个已知所在染色体信息的小豆SSR标记估计整个基因组的连锁不平衡水平(表5)。整个群体中连锁的和非连锁位点的组合共有1596种, 其中在显著性水平 P<0.001的条件下, 有331对(20.73%)位点组合间存在LD; 在显著水平 P<0.001且 R2>0.1的条件下, 有9对位点组合间存在LD。在所有组合中, 有147对共线组合。共线组合在显著性水平 P<0.001的条件下, 有50对(34.01%)位点间存在LD; 在上述所有有意义的位点间, 有5对(147对的3.40%)的 R2>0.1。在共线组合(3.40%)中观测到的LD水平比非共线组合(0.28%)的高。
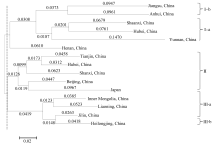 | 图4 基于SSR标记数据的种质资源省际间遗传距离聚类图Fig. 4 Dendrogram of provincial group of the adzuki bean accessions based on SSR |
| 表5 小豆种质材料中SSR位点的连锁不平衡模式 Table 5 Linkage disequilibrium (LD) patterns of SSR locus pairs in the adzuki bean collection |
为了得到参试小豆种质中的LD衰退图, 利用所有连锁SSR标记间的 R2值对两两位点间的遗传距离(cM)作散点图, 构建非线性回归曲线表现其趋势(图5)。对于本研究群体共线的SSR标记对, 通过对 R2值与连锁的标记间遗传距离的回归分析得到方程 y = -0.007 ln ( x)+0.0359, 其中 x为标记间遗传距离; y为两标记间的LD值。
由图可以看出, 随着位点间遗传距离的增加, R2值减小。回归方程结合LD值可得, 在 R2≥0.1的条件下的平均遗传距离小于1 cM, 观测到的最大衰减距离为5.8 cM; 在 R2≥0.03时能观察到的LD最大衰减距离为2.32 cM。
目前已有不少小豆遗传多样性研究的报道, 包括表型数据[ 27, 28, 29]和DNA分子标记研究[ 30, 31]。叶剑等[ 32]仅用7对SSR 引物分析不同国家104份小豆种质, 检测到平均等位变异为6.29个, 最多只检测到8个等位变异; 王丽侠等[ 33]用87对SSR引物分析国内外112份栽培小豆、野生小豆和近缘属植物, 检测到每SSR位点的等位变异数2~22不等, 平均为7.58个。
本研究所使用的引物, 一部分是从国外引进的开发的小豆SSR引物197对, 筛选到57对多态性引物, 多态性引物比率为28.93%; 一部分是本课题自主开发的约3000对绿豆SSR引物, 筛选到31对多态性引物, 多态性引物比率为1.03%, 与钟敏等[ 19]报道的绿豆引物在豇豆属中的通用性结果基本一致。88对多态性引物共检测出630个等位基因, 平均每位点检测到7.15个等位变异, 其中有效等位变异数3.18个, SSR位点等位变异数的变化2~17不等。不同指标均说明小豆SSR引物有较高的多态性水平, 在揭示遗传多样性方面存在巨大差异。
本文得到的结论较之以前的研究成果能检测到更丰富的变异类型, 很大程度上是因为参试材料来源广泛, 能较大程度代表小豆的遗传多样性, 而且所用SSR引物相对较多, 能覆盖较广的全基因组信息。但相关结果低于王丽侠等[ 33]的研究, 推测因为其所选野生及近缘材料居多, 而本研究所选全国范围内小豆资源栽培种居多。
当所用研究群体结构较复杂时, 由于群体结构分层及亚群内等位基因的不均衡分布将导致基因型与表型间的假阳性关联[ 34]。本研究中小豆种质资源的Structure遗传结构分析和NJ法树状聚类图分析的结果相互间基本吻合, 说明所选国内材料亲缘关系很近, 群体内遗传背景较窄, 遗传结构较单一。这和PowerMarker软件的遗传多样性估计结果相同。
Structure和NJ聚类分析的结果都显示, SG1(I)主要包括中国华东区(江苏、安徽), 华中区(陕西、湖北)和云南省材料, 而华东区和华中区又很明显地分为2个不同亚组, 云南省材料和华中区遗传关系较近。SG2(III)主要为东北三省及内蒙古材料, 东北区和内蒙古区明显分为2个亚组。SG3(II)主要为华北地区(北京、河北、天津、山西)材料, 这些省份均为小豆主产区, 详细分析发现其中的地理分布并无明显规律, 但日本材料也被分到该组内, 显示与中国北京材料遗传关系较近, 可能因为国外材料仅5份, 不具有代表意义, 而且所用试验材料包括一些杂交选育的品种, 不同种质间存在一定基因漂流。两种群体结构分析结果均显示中国小豆种质资源的遗传背景与其地理来源有较大的一致性。
群体的LD水平是关联分析定位QTL的理论基础, LD衰减距离直接决定了关联分析所需的标记密度, 并影响关联作图的精度[ 7]。Nordborg等[ 35]第一次对拟南芥 FRI基因位点附近的连锁不平衡度进行评估, 发现其衰减距离为250 kb (约为1 cM), 之后随着整个基因组更深入的研究发现LD距离接近 50 kb[ 36]。Hyten等[ 37]对大豆种质中的4个不同群体LD水平分别估计, 发现在26份野生原种( Glycine soja)中LD衰减距离没有超过100 kb, 而在其他3个群体材料中为90到574 kb不等。本研究群体的平均 R2值为0.022, 最大衰减距离( R2>0.1)为5.8 cM, 254份小豆的平均LD衰减距离小于1 cM ( R2>0.1)。小豆基因组大小约为539 Mbp[ 38], 遗传连锁图总长约为832.1 cM[ 17], 因此粗略估计本研究中LD衰减距离小于648 kb。相比而言, 小豆的LD衰减速率与大豆栽培种[ 37]差不多, 但慢于拟南芥[ 36]。
因目前对小豆LD和QTL定位的研究极少, 本研究只是基于少量分子标记(57个位点), 旨在初步了解小豆基因组连锁不平衡状态, 估计位点LD的衰减。
对小豆种质资源遗传背景的分析有助于新品种选育、种质创新等工作的进一步开展。研究均表明, 小豆种质资源呈现一定的地理区划。不同省份资源间的比较发现, 中国河南、云南、陕西、山西、湖北的多样性最丰富。这与徐宁等[ 39]和王丽侠等[ 40]的分析结果基本一致。进一步说明中国中西部省份的小豆资源中含有更丰富的遗传变异、外引种质有助于拓宽当地小豆品种的遗传背景, 这些为以后的资源收集、种质创新等工作提供可靠信息。
参试的日本材料与中国的华北地区小豆表现出较近的遗传距离, 可能原因是与中国这部分种质的最初来源上相近。日本小豆最初来源于中国, 但在日本长期的生态环境下, 经人工选择和改良, 在遗传结构上发生了很大的变异[ 41]。如果能引进日本的大粒、高产性状优异品种, 无疑将有利于我国小豆育种, 提高小豆产量。这一工作也为今后小豆品种改良中亲本的选择、发掘优异基因提供重要参考。
本研究得到的我国小豆资源的遗传多样性水平较其他研究中的小豆种质要高。利用SSR标记进行Structure分析得到的群体结构要比使用地理生态型信息得到的更精确。小豆图谱上的共线性的或是非共性的SSR位点组合都有一定程度的LD。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|