* 通讯作者(Corresponding author): 祁建民, E-mail:qijm863@163.com
第一作者联系方式: E-mail:gaoyangzhang2009@163.com
以圆果黄麻(
Using homologous cloning and modified RACE techniques, we cloned the full-length cDNA of jute cellulose synthetase gene excluding the 500 bp of 5' terminal from the stem bark of jute cultivar 179 (
黄麻( Corchorus capsularisL.), 又称绿麻, 为椴树科黄麻属一年生草本韧皮纤维作物, 是世界上最重要的长纤维作物之一, 产量和种植面积仅次于棉花[ 1]。由于黄麻纤维产量高、有光泽、吸湿性能好、散水快、易降解、质地柔软, 在商业上有金色纤维之称。近年来随着育种家的不断努力, 黄麻纤维产量有了大幅度的提高, 但由于黄麻韧皮纤维细胞壁木质化、单纤维细胞较短、粗硬等因素, 影响了黄麻纤维在面料纺织品上的有效应用和经济效益。
纤维素是细胞壁的主要成分, 也是黄麻育种的重点目标, 其基本结构单位是吡喃式D-葡萄糖, 是众多的葡萄糖残基以B-1,4糖苷键相连, 纤维素合成酶催化合成B-1,4糖苷链, 从葡聚糖的聚合开始, 逐步进入纤维素的结晶过程[ 2]。自从1996年从植物中克隆出第一个纤维素合成酶基因以来[ 3], 迄今, 在棉花、杨树、烟草、水稻、拟南芥、苎麻等40多种不同植物中[ 4, 5, 6, 7], 已有164 000多个与纤维素合成酶基因的相关序列信息报道。已有研究表明, 用含 Ces基因特异片段的质粒转化烟草, 可使植株表现出节间缩短、叶小和植株矮小症状, 叶表面细胞膨胀, 叶片的多聚糖含量和 CesA基因的表达水平降低, 说明cDNA片段可抑制一个或多个纤维素合成酶基因的表达[ 8]。拟南芥 AtCESA2基因被抑制后, 表现为微观定位变化, 细胞变得膨胀, 表明 AtCESA2基因在拟南芥细胞壁形成过程中起关键作用[ 9], 但这些研究在黄麻上未见报道。由于黄麻作物在基因工程研究上相对滞后, 本研究是在基因工程技术和一些模式植物的纤维素合成酶基因所取得的新进展基础上, 对黄麻纤维素合成酶基因, 进行序列和表达分析, 进一步构建载体, 阐述其功能, 为纤维素合成多基因互助机理和改善黄麻纤维品质提供新的视角。
以国家一级黄麻种质资源黄麻179为研究材料, Taq plus DNA聚合酶、dNTPs均购自生工生物工程(上海)股份有限公司, 感受态细胞DH5α为本实验室自备, 载体pMDl8-T、逆转录试剂盒(TaKaRa AMV Ver 3.0)购自大连宝生物公司, 植物表达载体pCAMBIA1301和拟南芥种子由福建省农业科学院作物研究所黄庭旭研究员提供, 其他试剂均为国产分析纯。
取田间生长旺盛的黄麻179茎皮组织100 mg迅速放入液氮速冻, 按OMEGA公司RNA提取试剂盒操作方法并改进, 提取总RNA, 用2 μL总RNA为模板, 按TaKaRa公司反转录试剂盒反应体系和方法合成cDNA第1条链并作为后续反应的模板。以拟南芥茎皮高表达的纤维素合成酶基因 AtCesA7的蛋白氨基酸序列(GenBank登录号为AF091713)为探针, 在NCBI网站运用BLAST程序在线检索, 选取SCOLE和E值较高的前20个物种的氨基酸序列, 运用CLUSTALW1.82程序进行多重比对, 根据在N末端和C末端的保守区域设计简并引物F1: 5°-CARC AYCAYAARAARGCNGG-3°, R1: 5°-RAANCCNGT NARDATRTC-3°。
采用Eppendorf公司梯度PCR仪, 以合成的cDNA第一条链为模板进行PCR, RT-PCR体系为 25 μL, 含ddH2O2(17.5 μL)、10×PCR buffer (2.5 μL)、10 mmol L-1 dNTPs混合物(2.5 mmol L-1各2 μL)、10 μmol L-1 F1 (2.5 μL)、10 μmol L-1 RI (2.5 μL)、 5 U μL-1 Taq(0.25 μL)、DNA聚合酶(1 μL), 反应程序为94℃预变性3 min; 94℃变性30 s, 53℃退火50 s, 72℃延伸3 min, 共35循环; 最后72℃延伸10 min。PCR产物使用百泰克公司琼脂糖胶回收试剂盒回收纯化, 连入PGEM-T EASY载体, 热激转化DH5α感受态细胞。对挑取的单克隆进行菌落PCR和质粒酶切验证, 将正确克隆送华大基因测序。cDNA 3°端扩增: 以前期得到的纤维素合成酶关键片段为模板设计3°端扩增引物, 3R1: 5°-TGGCATTGACTTGCACG ATCGATAT-3°和3R2 5°-TATGGAAAGAAAACGTG CTATGTGC-3°, 5°端扩增引物5R1: 5°-CTTGCAGAA AGGCACCCACT-3°和5R2: 5°-CAAACTCTGCGTT GATACCACT-3°, 以TaKaRa公司RACE试剂盒操作方法和程序进行3°端扩增。5°端克隆采用所描述的方法并作改进[ 10], 按关键片段测序的方法检测3°和5°端扩增片段, 最后运用DNAMAN软件拼接纤维素合成酶cDNA关键片段, 5°和3°端。
运用DNAMAN和BioEdit软件序列比对、ORF查找、ORF翻译和确定蛋白质基本性质等, 运用NCBI BLAST网站对核苷酸和相应的氨基酸序列进行分析。为进一步了解 CcCesA1在黄麻不同部位组织中的表达情况, 以40 d黄麻植株苗为材料, 以黄麻18S RNA基因片段为内参, 正向引物为F3: 5′-GTGGAGCGATTTGTCTGGTT-3′, 反向引物为R3: 5′-TGTACAAAGGGCAGGGACGT-3′。以 CcCesA1基因3′端非保守区域为检测目的基因的引物, 正向引物为F4: 5′-GAGGAAGGTGTTGAAGGAT-3′, 反向引物为R4: 5′-GAATAGAGGTAAGGGGGTAA-3′, 运用RT-PCR分析黄麻纤维素合成酶基因在不同组织中的表达情况。
设计引物扩增黄麻纤维素合成酶基因3′UTR区, 扩增引物F5为5′-GGGTAACCCAGAAGATGCCTG AAGAAG-3′, R5为5′-GAAGATCTATAACCCATAA GGCGAAG-3′, 下画线部分分别为 BstE II和 BglII酶切位点。利用 BstE II和 BglII酶对扩增片段和植物表达载体双酶切, 回收酶切目的片段和载体大片段, T4连接酶16℃进行正义片段定向连接16 h, 电激转化并筛选, PCR和酶切检测。将酶切检测正确的质粒载体送华大基因测序。利用冻溶法将构建的反义表达载体pCAMBIA1301-CcCesA1转化农杆菌感受态细胞EHA105。利用农杆菌介导的花絮侵染法[ 11], 用构建好的载体侵染即将开花的花絮。
拟南芥种子先用75%的乙醇消毒5 min, 然后在5%的次氯酸钠中浸泡15 min, 无菌水冲洗3次, 每次停留2 min, 放入MS培养基(添加50 mg L-1卡那霉素和25 mg L-1潮霉素), 两周后, 将仍保持绿色的小幼苗移栽到营养土上生长。40 d后, 提取潮霉素筛选阳性植株的DNA, 根据转化载体质粒图谱pCAMBIA1301, 选择3个单酶切位点, 即 BstE II、 BglII和 NcoI, 将5~10 μg基因组DNA分别酶切过夜、电泳、转膜、印迹、预杂交、杂交。采用碱性磷酸酶探针标记试剂盒(ROCHE公司提供)标记HYG探针。HYG探针引物为(R: 5′-CGATGTAGGA GGGCGTGGAT-3′和F: 5′-CGCTTCTGCGGGCGATT TGT-3′)。以pCAMBIA1301质粒为阳性对照, 以非转基因拟南芥为阴性对照。采用凝胶成像仪FluorChem SP 50 mm f11.4镜头采集。通过NCBI在线网站分析 CcCesA1基因的核苷酸序列发现与拟南芥 CesA10基因相似性高达80%, 因此运用 CesA10基因特异片段设计引物(F: 5′-ACTGGCAATGTCTT TGTCGC-3′; R: 5′-GGAGGCTCTTTCATCGGGTC-3′), 提取40 d时期转基因和非转基因拟南芥RNA, 对 CesA10基因进行表达分析。
为进一步了解 CcCesA1基因对拟南芥产生的影响, 我们对转基因拟南芥和阴性对照的生长速率和植株大小进行观察记录, 对60 d时期的转基因拟南芥和阴性对照植株纤维素含量进行测定, 参照Updegraff描述的方法进行[ 12]。
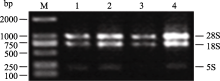
经过比较分析不同公司的总RNA提取试剂, 获得了高质量的RNA (图1), 图1显示, 3条主带清晰, 条带锋利, 无明显降解, 28S条带约为18S的2倍, 紫外检测RNA浓度为876 ng μL-1, A260/ A280=1.91, 其浓度和纯度均达到进一步试验的要求。
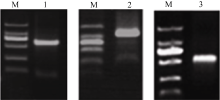
黄麻纤维素合成酶基因cDNA关键片段、3°端和5°端序列的克隆结果见图2。以cDNA为模板, 以F1与R1引物扩增得到约600 bp的关键片段; 3°RACE扩增反应中, 以3R2与通用引物扩增得到约1400 bp的条带; 5°RACE扩增反应中, 以5RI、通用引物及5R2、通用引物进行巢式PCR扩增得到约500 bp的条带。
利用DNAMAN软件对测序结果进行拼接后得到2529 bp的核苷酸序列(5°端缺少包括起始密码子在内的约500 bp), 编码一段627个氨基酸的推导性蛋白质(图3)。利用在线分析软件(http://www.pred-ict- protein.org/)分析了黄麻纤维素合成酶蛋白质结构特征, 结果显示在黄麻纤维素合成酶蛋白质氨基酸序列中包含了酪蛋白激酶II磷酸化位点、酪氨酸激酶磷酸化位点、N糖基化位点、蛋白激酶C磷酸化位点、氨基化位点和N一肉豆蔻酰化位点等, 这与其他植物纤维素合成酶蛋白研究的结果一致[ 13]。用BLAST在线分析软件(http//ncbi.nlrn.nig.gov/blast)将 CcCesAl基因cDNA部分序列及推导的氨基酸序列与
GenBank中的序列进行同源性比较后发现核苷酸相似性与棉花(JQ345694.1)为92%, 与杨树(AY162181.1)为87%, 与苎麻(KC112993.2)为86%, 与桉树(HQ 864587.1)为84%, 与相思木(AY643519.1)为83%, 与蒺藜状苜蓿(XM_003603371.1)为82%, 与水稻(NM_ 001061323.1)为80%, 与拟南芥(XM_002880638.1)为80%, 与玉蜀黍(NM_001061323.1)为79%。氨基酸相似性与棉花(AFB18635.1)为97%, 与苎麻(AAY78952.3)为95%, 与杨树(AAO25536.1)为95%, 与葡萄(CAN 60659.1) 为95%, 与拟南芥(XP_002867245.1)为90%。核酸水平和蛋白质水平上均与棉花CesA6同源性最高, 分别为92%和97%。这表明我们已经成功地克隆了黄麻 CesA基因(5°端缺少包括起始密码子在内的约 500 bp) cDNA序列。以不同黄麻组织cDNA为模
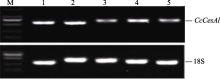
板, 18S为内参, PCR电泳结果表明(图4), 黄麻纤维素合成酶基因在不同组织中均有表达, 利用凝胶定量分析软件BandScan对电泳条带密度扫描后数据分析发现, 其表达量是茎部韧皮>根>叶>顶芽>麻骨。
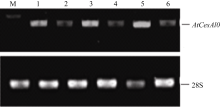
提取经潮霉素筛选的阳性植株基因组DNA, 通过Southern杂交显示, 阳性对照Pcambia1301质粒和转基因植株都有一个拷贝位点, 而阴性对照没有该拷贝位点(图5)。这说明外源基因已经整合进入拟南芥基因组, 且只有一个插入位点。纤维素合成酶基因在植物所有组织和不同类型的细胞中都有表达, 我们分析了拟南芥 AtCesA10基因在40 d的表达情况, 在所用转基因拟南芥中 CesA10基因都不同程度地被抑制(图6), 而在非转基因植株中 CesA10基因表达正常, 这说明转基因拟南芥纤维素含量不同程度的降低与 CesA10基因被不同程度抑制密切相关。
测定转基因拟南芥和阴性对照在50 d后的生长速率发现, 携带 CcCesA1基因反义片段的拟南芥与对照相比植株生长速度变慢, 矮小并伴随着茎杆弯曲(图7)。为了进一步确定 CcCesA1基因的功能, 我们检测了转基因拟南芥中的纤维素含量, 试验共重复3次, 结果显示, 转基因拟南芥中的纤维素含量都有不同程度的减少(表1), CcCesA1反义基因片段能抑制拟南芥中纤维素的合成, 这进一步表明 CcCesA1基因除了主要参与纤维素合成, 也参与植物的其他生长发育过程。
| 表1 转基因拟南芥与性对照纤维素含量 Table 1 Cellulose content intransgenic and non-transgenic lines |
数值为6个株系重复的平均值±标准差, 同一列数值后字母不同表示差异显著性0.05水平差异显著。
Each value represents mean of six replicates ± SD. Means were compared using ANOVA. Means followed by different letter within a column are significant difference at 0.05 probability level.
植物纤维素的合成是一个极其复杂的过程, 参与纤维素合成的基因有上千种, 纤维素合成酶基因是一个非常庞大的基因家族[ 14], 除了植物, 一些细菌、真菌和部分动物也能合成纤维素, 世界上第一个纤维素合成酶基因在细菌中被克隆, 随着大量纤维素合成酶基因在不同植物中相继克隆。揭示其蛋白质氨基酸在N端可折叠形成类似于锌指状(zinc finger)结构或LIM转录因子构象[ 15]。目前, 对这些结构域的功能还不清楚, 植物纤维素合成酶包含2个高变区和8个跨膜区[ 16, 17], 本文在黄麻中所克隆的纤维素合成酶基因也具有类似的结构域, 这表明, 纤维素合成酶基因在进化过程中具有一定的保守性。
植物纤维素合成酶基因表达规律研究也有大量报道, 准确地说是纤维素合成酶基因的不同亚基, 例如 AtCesA 1基因几乎在植物的各个组织均有都表达, 而 AtCesA仅在叶片和茎的连接处和胚的发育过程中特异表达[ 15]; 其次, 在植物不同部位不同的 CesA基因表达模式也不一样, AtCesA7(Irx3)表达量与 AtCesA 1相当, 但 AtCesA7 (Irx3)基因仅在木质部特异表达[ 19, 20] CcCesA1在根、茎和叶中均有表达, 但表达量有所差别, 这符合植物 CesA的表达规律。
有关拟南芥纤维素合成酶基因研究主要集中在 At CesA1、 AtCesA2、 AtCesA3、 AtCesA4、 AtCesA6、 AtCesA8和 AtCesA9等基因上, 这些基因参与初生细胞壁的合成, 参与纤维素合成酶复合体的组装, 或参与种子外粘液的形成等[ 18]。目前普遍认为 AtCesA1、 AtCesA3和 AtCesA6与初生壁的合成相关, AtCesA4、 AtCesA7和 AtCesA8与次生壁合成有关, AtCesA9和 AtCesA10在生长点表达, 但是 AtCesA2、 AtCesA5、 AtCesA9和 AtCesA10的具体功能尚不清楚[ 19]。彭良才等[ 20]通过筛选和鉴定4个拟南芥的突变体(rsw 1, 2, 3, 5), 首次发现和鉴定了植物纤维素合成酶基因, 并提供了充足的生化和遗传证据, 这些拟南芥突变体的纤维素合成严重受阻并同时生产大量的非晶体状纤维素(non-crystalline cellulose)和淀粉(starch)。由于此非晶体状纤维素具有能够有效被纤维素酶(endo-cellulase)分解或被弱酸全部降解成单糖(glucose)的特性, 为利用现代生物技术提高植物纤维素降解并高效转化成生物能源提供了可行性的理论依据。为纤维素生物合成机理的研究迈出了关键的一步[ 20]。目前有关纤维素合成酶的研究主要集中在纤维素合成酶多基因家族复合体的组装、结构功能的调控以及细胞壁的结晶度和复合体的合成速率等方面[ 21, 22]。对拟南芥 AtCesA10基因研究目前只见于序列的报道和分析[ 23], 对其功能分析方面还未见有人研究。在本研究中, 转基因拟南芥的生长表型、纤维素含量等变化, 与 At CesA10基因的表达受到了不同程度的抑制密切相关。转基因个别株系之间差别较大, 这是不同的转化事件, 转基因插入拷贝数不同等原因引起的, 类似的现象在转基因水稻, 玉米等也有报道。
携带 CcCesA1基因干扰片段的拟南芥在生长速率和生物量上与非转基因对照均有明显差异, 同时, 还伴随着植株的矮化和易倒伏, 说明 CcCesA1基因不仅参与纤维素和次生木质部的生物合成, 在植物的生长发育过程中也其重要作用, 这与在烟草中的研究结果相一致, 因此, 本研究无论对黄麻纤维素合成酶基因( CcCesA1), 还是拟南芥 At CesA10基因的功能的进一步分析都具有非常重要的意义。
成功克隆了黄麻纤维素合成酶基因 CcCesA 1 5°端500 bp序列以外的全部cDNA序列, 序列长2529 bp, 编码一段627个氨基酸的蛋白质。其蛋白质序列包含植物纤维素合成酶全部功能性基序(motif)和保守结构域。 CcCesAl基因在黄麻根、茎皮、麻骨、叶和顶芽中均有表达, 其中在茎皮中表达量最高。与茎皮中纤维素的大量合成有关。外源基因以单拷贝方式整合进入基因组。携带黄麻纤维素合成酶基因干扰片段的拟南芥与非转基因对照相比, 表现出生长速率变慢, 植株矮小且易于弯曲倒伏, 并伴随着纤维素含量降低。黄麻纤维素合成酶基因不仅参与纤维素和次生木质部的合成, 也参与植物的其他生长发育过程。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|