芜菁花叶病毒(turnip mosaic viruses, TuMV)是侵染重要经济作物的主要病毒之一, 寄主范围十分广泛, 尤其是对十字花科作物的危害最为严重。为了获得对TuMV持久稳定的高度抗性, 本研究以TuMV
TuMV (turnip mosaic virus) with broadest known hosts, is one of the most major viruses in important economic crops, especially for cruciferous plants. In order to get durable and stable high resistance to TuMV, in this study, a 453 bp segment of TuMV
芜菁花叶病毒(turnip mosaic viruses, TuMV)属马铃薯Y病毒科( Potyviridae)马铃薯Y病毒属( Potyvirus)。TuMV寄主范围十分广泛, 能够侵染43科156属超过318种植物[ 1, 2]。TuMV是危害十字花科作物最大的病毒病。感染病毒后的作物还容易感染霜霉病和软腐病, 造成复合侵染, 直接影响产量和商品价值[ 3]。近年来, 在我国油菜主产区病毒病发生有日益流行加重趋势。据调查发病田块发病率一般为20%~30%, 重的发病率在50%以上, 造成油菜籽产量的重大损失[ 4]。感病油菜不仅产量降低, 而且产油率和种子萌芽率也明显降低, 品质下降[ 5, 6]。我国白菜因该病毒危害平均每年造成5%的产量损失, 有些年份减产10% 以上, 病害严重的地块几乎绝收[ 6]。TuMV主要是靠蚜虫以非持久性的方式传播, 据报道目前至少有89种蚜虫参与, 而采用杀虫剂不可能很快杀死全部蚜虫以阻止病毒的传播, 不但效果不明显, 还易造成环境污染[ 7]。因此寻求新的抗病方法成为一项迫切的任务。
基因工程技术的发展为植物病毒病的防治提供了一条崭新的途径。1986年Abel等[ 8]首次将烟草花叶病毒(TMV)的外壳蛋白(CP)基因导入烟草, 培育出抗TMV的烟草植株。在植物抗病毒基因工程中, 利用病毒的 CP基因最早获得成功, 也是至今应用最为广泛的一种策略。也有许多利用病毒的其他序列进行抗病毒的研究报道, 但相比之下要少些。HC-Pro蛋白(helper component protease)是TuMV的10个蛋白产物中的一员, 因最初被发现是蚜虫传毒的辅助成分而得名; 它具有蛋白酶活性, 参与多聚蛋白翻译后的水解加工过程[ 2]。病毒在植物体内的长距离移动和维持复制也与其有关[ 9]。HC-Pro还是RNA沉默的抑制因子[ 10]。尽管马铃薯Y病毒中李痘病毒(plum pox virus)利用 HC-Pro基因片段抗病毒已有成功的报道[ 11], 但利用 HC-Pro基因片段抗TuMV的报道较少。因为病毒的沉默抑制因子在与植物的防御系统对抗中扮演重要的角色, 我们选择TuMV的 HC-Pro为靶标进行抗病毒研究。试验以TuMV的 HC-Pro的保守区段453 bp为目的片段构建植物表达RNAi载体, 并转化TuMV的天然宿主拟南芥, 以验证利用 HC-Pro基因片段构建的载体对TuMV的抗性。
野生型拟南芥Col-0购自Arabidopsis Biological Resource Center。北京地区TuMV主要的流行强致病株系BJ-C4 (HQ446217)[ 12]由中国农业科学院蔬菜花卉研究所提供。BJ-C4的cDNA克隆为本实验室前期构建并保存。大肠杆菌( Escherichia coli) DH5α菌株、农杆菌( Agrobacterium tumefacieus) GV3101:pMP90由本实验室保存。RNAi载体pHannibal[ 13]由CSIRO的Peter Waterhouse博士惠赠。植物表达双元载体pBBBasta由地中海大学的Christophe Robaglia博士惠赠。
高保真酶Fast pfu DNA Polymerase、Easy Taq酶和分子量标记购自全式金公司。质粒小提试剂盒、胶回收试剂盒、PCR纯化试剂盒购自康为世纪生物科技有限公司。M-MLV反转录酶、定量PCR试剂、限制性内切酶和Klenow Fragment购自TaKaRa公司。T4 DNA连接酶购自Promega公司。TRIzol提取液购自Invitrogen公司。引物由上海捷瑞生物工程有限公司合成, 由上海生工生物工程技术服务有限公司测序。
比对GenBank登录的122个TuMV株系序列, 根据TuMV不同株系间的保守性, 选取 HC-Pro基因的第841~1293位核苷酸序列共453 bp的保守片段为靶点, 构建RNAi载体。模板为BJ-C4的cDNA克隆。根据pHannibal载体上的酶切位点设计引物HC-Pro F和HC-Pro R (表1, 下画线部分为添加的酶切位点 EcoR I、 XbaI和 KpnI、 ClaI), 用高保真酶Fast pfu DNA Polymerase进行PCR扩增。扩增程序为94℃ 5 min; 94℃ 45 s, 52℃ 30 s, 72℃ 1 min, 35个循环; 72℃延伸7 min。PCR产物经琼脂糖电泳纯化后回收。
| 表1 研究中所用引物序列 Table 1 Primers used in this study |
用 ClaI和 XbaI分别酶切pHannibal载体和 HC-Pro目的基因片段, 经纯化回收后4℃连接过夜。连接产物经热击转化大肠杆菌。挑取克隆, 提取质粒并用 XbaI和 EcoR I酶切验证, 获得含反向片段的中间载体pHannibal+HC-Pro(-)。
用 KpnI和 EcoR I分别酶切 HC-Pro目的基因片段和连入反向片段的pHannibal+HC-Pro(-)中间载体。酶切产物经纯化回收后4℃连接过夜。连接产物转化大肠杆菌, 挑取克隆, 提取质粒并用 XbaI和 EcoR I双酶切验证, 获得同时含反向和正向片段的中间载体pHannibal+HC-Pro- RNAi。
pHannibal的发卡结构两端各含有1个 NotI酶切位点。把含发卡结构的中间载体pHannibal-HC-Pro-RNAi用 NotI酶切, 再用Klenow和dGTP在酶切片段的黏性末端补2个碱基G, 产物以1%琼脂糖凝胶电泳纯化, 回收含发卡结构的 NotI目的片段备用。
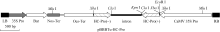
用 XmaI酶切植物表达双元载体pBBBast, 再用Klenow在黏性末端补2个碱基C, 产物经1%琼脂糖凝胶电泳, 回收载体片段, 再与含发卡结构的RNAi片段连接。所得质粒用 MluI酶切验证, 获得植物表达RNAi质粒pBBBTu-HC-Pro (图1)。
利用电击法将pBBBTu-HC-Pro载体转入农杆菌GV3101:pMP90, 采用农杆菌介导的花序浸渍法侵染拟南芥[ 14]。将收获的拟南芥T0代种子播于盛有营养土的培养托盘中, 置22℃(昼)/18℃(夜)及光照周期为16 h (光)/8 h (暗)的人工气候室。待幼苗长出两片子叶后喷洒浓度为0.1%~0.2%的商业Basta除草剂筛选阳性苗。
经喷洒2~3次Basta除草剂筛选后, 从能够继续生长且长势良好的植株用小量快速法[ 15]提取植物基因组DNA。用引物HC-Pro F和HC-Pro R进行PCR扩增检测。反应条件为94℃ 5 min; 94℃ 45 s, 52℃ 30 s, 72℃ 30 s, 35个循环; 72℃ 7 min。PCR产物经1%琼脂糖凝胶电泳检测, 扩增出目的基因片段的为转基因阳性苗。
转基因T1代苗自交留种, 对T2幼苗喷洒0.1%~0.2% Basta除草剂后统计存活率。存活/死亡比约为3∶1, 初步确定为单拷贝植株, 并单株自交留种。继续筛选后代至不分离株系即为纯合株系。
将T3代单拷贝纯合株系播种, 待幼苗长至8~10片莲座叶时期摩擦接种TuMV的BJ-C4株系。接种液按病叶鲜重/磷酸缓冲液体积(pH 7.0)约1 g 10 mL-1比例配制。每个株系接种12~16株, 每株接种2片较大叶片。以非转基因野生型拟南芥Col-0作为对照接种。接种2 min后立即用清水冲洗叶片。在人工气候室中网罩隔离观察, 约20 d后进行抗病鉴定及病情指数统计。病情等级划分为0级(完全无症状); 1级(1~2片未接种叶花叶或发黄, 能抽薹); 3级(3~4片未接种叶花叶或发黄, 能抽薹); 5级(5~6片未接种叶花叶或发黄, 影响抽薹); 7级(多数叶片花叶或发黄, 不能抽薹); 9级(大部分叶片枯死, 植株濒临死亡)。
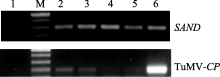
接种后约20 d, 选取各株系未接种叶片分别提取总RNA, 用M-MLV反转成cDNA。分别以TuMV- CP的196 bp片段为检测对象, 以拟南芥 SAND基因(AT2G28390)的298 bp片段为内参进行半定量和荧光定量分析。 SAND上游引物跨过内含子(表1, 一条下画线是外显子a, 2条下画线是外显子b), 以避免因基因组DNA污染所造成的影响。反应程序为94℃ 5 min; 94℃ 30 s, 57℃ 30 s, 72℃ 30 s, 30个循环; 72℃ 7 min。PCR产物经1.2%琼脂糖凝胶电泳检测。荧光定量PCR的反应以稀释40倍的cDNA为模板, SYBR-green I作荧光指示剂, 反应程序采用两步法扩增。扩增条件为95℃ 30 s; 95℃ 5 s, 57℃ 30 s, 72℃ 30 s, 40个循环。每个株系样品3次重复, 取平均值。
利用引物HC-Pro F和HC-Pro R以TuMV BJ-C4株系的cDNA克隆为模板, PCR扩增481 bp的目的带。纯化的PCR产物经双酶切后按先反向后正向的顺序插入pHannibal载体。
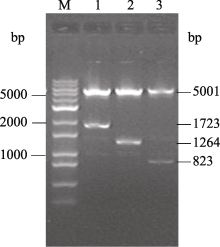
用 XbaI和 EcoR I分别双酶切pHannibal+HC-Pro- RNAi、连入反向片段的pHannibal+HC-Pro(-)和pHannibal空载体验证。将pHannibal+HC-Pro-RNAi载体酶切成5001、1723和6 bp, 连入反向片段的pHannibal+HC-Pro(-)载体切成5001 bp和1264 bp, pHannibal空载体切成5001 bp和823 bp。酶切结果显示质粒构建正确(图2)。
用 NotI酶切pHannibal+HC-Pro-RNAi中间载体, 获得含正反向片段的RNAi发卡结构片段。将该片段插入植物表达双元载体pBBBast, 获得植物表达RNAi载体pBBBTu-HC-Pro。
植物表达载体pBBBTu-HC-Pro进行 MluI酶切检测。pBBBast载体本身有一个 MluI酶切位点, 连接的目的RNAi片段的CaMV启动子上另有一个 MluI酶切位点, 酶切后显示2条带的为阳性克隆。因是单酶切位点插入, 根据酶切条带大小可判断插入是与标记基因同向连接或反向连接(图1)。同向连接酶切条带为542 bp和9889 bp, 反向连接条带为3935 bp和6494 bp。结果显示RNAi结构已经连入植物表达载体, 并且为反向连接(图3)。
播种转化的拟南芥T0代种子。幼苗经喷洒2~3次Basta除草剂后非转基因苗不能继续生长, 逐渐枯黄死亡; 转基因苗叶片保持绿色, 长势茁壮。共获得60株除草剂抗性植株。提取T1代转基因苗的叶片基因组DNA进行PCR验证, 共有26株为PCR阳性苗。部分幼苗的PCR鉴定结果见图4。阳性苗自交留种, 后代经除草剂鉴定有约13个株系符合3∶1分离比, 初步认定为单拷贝插入株系。T2代继续分株自交留种, 后代经除草剂鉴定, 获得纯合株系。
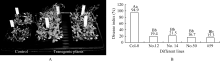
将T3代13个单拷贝的纯合株系与野生型Col-0同时进行抗病接种鉴定。接种后20 d左右, 对照野生型Col-0
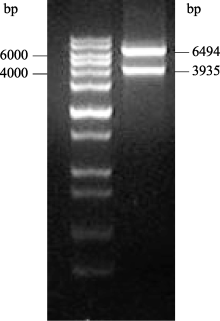 | 图3 pBBBTu-HC-Pro Mlu I酶切结果M: 1 kb plus分子量标记; 1: pBBBTu-HC-Pro。Fig. 3 Digestion results of pBBBTu-HC-Pro by MluI restriction enzymeM: 1 kb plus marker; 1: pBBBTu-HC-Pro. |
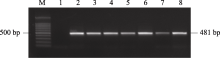 | 图4 转基因植株的PCR鉴定M: 100 bp分子量标记; 1: 阴性对照; 2~8: 不同的转基因阳性植株。Fig. 4 Identification of the transgenic plants by PCRM: 100 bp marker; 1: negative control; 2-8: individual transgenic plants. |
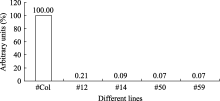
已基本枯萎死亡, 而转基因株系表现出不同的抗性, 其中4个株系(12号、14号、50号和59号)表现出较好的长势。高抗株系除部分叶片发黄外, 整株生长健壮, 且能正常抽 
本研究利用TuMV的 HC-Pro基因片段构建了抗病毒的RNAi植物表达载体pBBBTu-HC-Pro。该载体转化拟南
 | 图7 转基因株系接种TuMV BJ-C4荧光定量PCR检测结果Fig. 7 Real-time PCR result of transgenic lines inoculated with TuMV BJ-C4 strain |
芥后得到了高抗TuMV的株系。鉴定的13个独立的单拷贝转基因株系中有4个表现出高抗TuMV。在接种后的高抗转基因植株体内几乎检测不到病毒的累积; 其他株系则表现出对TuMV的不同程度的抗性。这应该是因不同的转化插入位点效应所致[ 16]。外源基因在受体基因组中的插入方式、转基因的甲基化状况、转基因的拷贝数等都对外源基因的表达效率有影响。转基因植株的抗病性与转基因的拷贝数的关系目前还存在着争议。Goodwin等[ 17]认为转基因植株的抗病性与外源基因的拷贝数有关。但Nomura等[ 18]研究表明植株的抗病性与转基因的拷贝数关系不大, 含单个拷贝和多个拷贝的转基因植株都有表现高度抗病的株系。本研究只对推测的单拷贝转基因株系进行了抗病鉴定。在获得的T1代植株中从分离比看存在多个多拷贝株系(数据未显示); 其抗病性情况有待以后进一步确定。
现在新的抗病毒方法在不断涌现, 利用amiRNA策略抗TuMV也已经获得成功[ 19]。但由于amiRNA技术只是利用相对较短的21-nt病毒保守序列, 为了防止因病毒的变异引起的抗性丢失, 研究者建议可针对同一种病毒同时共表达多个amiRNAs[ 19]。当前利用病原体介导的抗性(PDR)仍是抗RNA病毒最有效的策略, 且商业化的转基因抗病毒作物也已经面向市场[ 20]。许多研究实验证明病毒自身基因介导的转基因抗性取决于靶区域的同源性高低而非整个序列[ 21]。Nomura等[ 18]把TuMV -CP基因转入拟南芥, 发现转基因株对17种TuMV分离物表现出广谱抗性。转入序列与这17种TuMV分离物序列的同源性至少为86.6%, 且 CP基因3°端380个核苷酸的同源性超过了95.5%。我们选取的 HC-Pro基因453 bp的保守区域与GenBank已登录的全部254个TuMV的该段序列同源性为79%~100%, 其中同源性≥87%的占48.8%。我们构建的抗病毒植物表达载体pBBBTu-HC-Pro获得了高抗TuMV的结果, 因序列具有较高的同源性, 推测对多数的TuMV株系应具有广谱性抗性, 实际效果需进一步验证。
致谢: 感谢Peter Waterhouse 博士和Christophe Robaglia博士惠赠RNAi载体pHannibal和植物表达双元载体pBBBasta。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| 22 |
|
| 23 |
|
| 24 |
|
| 25 |
|
| 26 |
|
| 27 |
|