以我国10个大豆育成品种重要家族的179个品种为材料, 选用161个均匀分布于大豆基因组的SSR分子标记, 采用PowerMarker Ver. 3.25软件分析参试材料的遗传多样性、相似性与特异性。结果表明, 161个位点上共检测到1697个等位变异, 单位点变幅为5~24个, 平均10.5个; 多态信息含量在0.549~0.937间, 平均0.819; 群体具有丰富的遗传变异。聚类分析表明, 179个品种可归为6大类11小类, 同一家族的品种有聚为一类的趋势。品种间亲本系数和遗传相似系数显著相关(
Analysis of the affinity relationship and genetic similarity among cultivars is important for crop genetic improvement. In this study, a total of 161 SSRs covering the entire soybean genome were analyzed for the genetic diversity, similarity and specificity of 10 important families composed of 179 cultivars using PowerMarker Ver. 3.25. The results showed that there were totally 1697 alleles, averaging 10.5 per locus, ranging from 5 to 24 with average polymorphism information content of 0.832, ranging from 0.545 to 0.943 in the population. According to the SSR cluster analysis, the 179 cultivars were clustered into six groups, eleven subgroups, with a tendency that the cultivars in a family tended to be grouped into a same cluster. There existed significant correlation (
亲本系数(CP)是指品种间携带血缘等同基因的概率, 它所阐明的是品种间的亲缘程度, 可以反映出品种整体亲缘关系的相近程度。品种间的亲本系数是一个非常有用且有说服力的遗传参数, 具有可比性, 可以反映总体的基因流, 是对基因组的宏观分析。近年来, 国内外都重视大豆育成品种及其亲本的研究, 主要是分析品种的系谱, 系统获得品种亲缘关系信息, 为大豆育种理论与应用研究提供参考[ 1, 2, 3]。Sneller等[ 4]分析了美国大豆育成品种间的亲缘关系(亲本系数或共祖先度), 发现群体亲本系数均值为0.173, 遗传多样性高。Bharadwaj等[ 5]对印度1968—2000年的育成品种系谱分析表明66个品种亲本系数平均值为0.050。Bonato等[ 6]对巴西1984— 1998年育成的100个品种CP分析的结果, 平均值为0.21, 比美国的育成品种略低。
近年来分子标记技术在作物亲缘关系及其遗传相似性研究中发挥重要作用[ 7], 尤其是共显性的SSR标记, 具有丰富的等位变异, 是研究品种间相似性较理想的分子标记。分子标记方法是通过抽取整个基因组上有代表性的部分标记来研究品种间的遗传相似程度。迄今已有利用分子标记分析大豆的遗传多样性、品种间遗传关系[ 8, 9, 10, 11, 12, 13, 14, 15]和群体分化[ 16, 17]的大量报道, 也有一些利用分子标记和系谱资料综合分析作物品种间亲缘关系及其遗传相似性研究的报道[ 17, 18, 19, 20]。
作物育成品种对环境的适应能力是作物育种的关键, 拓宽栽培品种的遗传基础是作物品种能否大量推广的关键所在。大豆育成品种不仅是重要的受体亲本也是重要的供体亲本, 是直接应用于育种实践的核心种质资源。若要拓宽作物育种材料的遗传背景, 一方面要通过表型鉴定不断挖掘丰富的优异种质, 另一方面要分析比较作物种质间的相似性和相异性, 为作物育种的亲本选择提供依据。
熊冬金等[ 21, 22]对我国2005年以前育成的1300个育成品种进行系谱分析, 追溯到来自670个祖先亲本, 并归纳出54个最为重要的祖先亲本, 其中51-83 (A002)、白眉(A019)、滨海大白花(A034)、奉贤穗稻黄(A084)、滑县大绿豆(A122)、即墨油豆(A133)、上海六月白(A201)、铜山天鹅蛋(A231)、武汉菜用大豆混合群体(A291)和山东寿张县无名地方品种(A295)等10个祖先亲本衍生的品种数共计1228份(次)。本研究以这10个家族群体为对象, 通过计算亲本系数和SSR标记分析, 研究其品种间的遗传关系, 揭示其衍生群体的遗传基础, 以期为我国大豆育种亲本选配, 扩大亲本来源, 拓宽我国大豆育成品种遗传基础提供参考。
选用我国51-83 (A002)、白眉(A019)、滨海大白花(A034)、奉贤穗稻黄(A084)、滑县大绿豆(A122)、即墨油豆(A133)、上海六月白(A201)、铜山天鹅蛋(A231)、武汉菜用大豆混合群体(A291)和山东寿张县无名地方品种(A295)等10个主要亲本家族的179份系谱信息完整的大豆品种(表1), 为研究材料。这些大豆分布于I、II、III、IV和V等5个不同生态区, 包括从1943年育成的丰地黄到2005年育成的GS郑交9525等, 具有广泛的代表性。
利用苗期的幼嫩叶片, 参考Aljanabi等[ 23]的CTAB法, 提取和纯化大豆基因组DNA。所用的161个SSR标记均匀覆盖大豆20条染色体[ 24]。PCR体系为10 μL, 含模板基因组DNA溶液(50 ng) 3.0 μL、10×buffer 1.46 μL、10 mmol L-1 dNTPs 0.24 μL、0.5 U Taq酶0.1 μL、25 mmol L-1 Mg2+0.8 μL、引物3.0 μL、ddH2O 1.4 μL。PCR程序为94℃预变性3 min; 95℃变性30 s, 55℃退火40 s, 72℃延伸40 s, 30个循环; 72℃延伸8 min; 4℃保温。PCR扩增产物中加入2 μL溴酚蓝缓冲液, 在8%聚丙烯酰胺胶上20 W恒功率电泳1 h左右, 然后用银染法显色, 进行凝胶成像扫描保存图像, 接着用Bio-Rad的Quantity One软件、根据晶美生物工程公司生产的pBR322DNA/ BsuRI ( HaeIII) Marker测定扩增条带的分子量大小。
参考Cox等[ 25]和Cui等[ 26]方法, 计算亲本系数计算原则如下: 品种分别从其双亲得到一半的基因; 所有祖先种、亲本及其后代品种都是纯合的; 最早的祖先品种(系)间的亲缘系数为0; 混合授粉材料, 各花粉供体享有相同的等同于雌配子的亲本系数概率; 品种与其系选所得品种间的 CP=0.75; 自然突变和诱导突变材料与其祖先的亲本系数为0.75; 品种与其自身的 CP=1.0; 含有部分相同亲本的旁系品种间的亲本系数, 计算公式为 CP= RSD=Σ[(1/2) n], R代表品种S和品种D之间的亲本系数, n代表品种S和品种D的共同亲本到品种S和品种D的世代数之和。
| 表1 供试材料来源 Table 1 Source of tested materials |
利用PowerMarker Ver. 3.25软件[ 27]对群体进行遗传多样性分析、聚类分析和遗传距离计算。遗传多样性的评价指标主要是等位变异数和多态性信息量(polymorphism information content, PIC)。根据Nei’s等[ 28]的方法计算遗传距离: 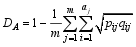
比较2个群体的等位变异时, A群体有而B群体没有的等位变异可以作为用A拓宽B遗传基础的潜力, 此处称为A对B的补充等位变异; A对B与B对A的补充等位变异数之和可以衡量A与B实际相差的等位变异数、也可以评价A和B遗传关系远近, 此处称为A与B的互补等位变异数。群体间互补的等位变异中有一部分变异是群体特殊的。特有等位变异是某群体具有而其他各群体都没有的等位变异; 特缺等位变异是某群体没有而其他各群体都有的等位变异; 特有、特缺等位变异表示了群体的特异性[ 17]。
161个SSR位点在染色体上的分布见表2, 在这些位点上检测到1697个等位变异, 平均每个位点等位变异数为10.5个, 变化介于5~24个; 多态性信息量平均值为0.819, 范围在0.549~0.937间。以上表明10个重要家族群体所构成的样本具有丰富的遗传变异, 品种间多样性高。
10个重要家族群体中, 平均等位变异数较多的是滨海大白花(A034)家族、铜山天鹅蛋(A231)家族、即墨油豆(A133)家族和山东寿张县无名地方品种(A295)家族; 较少的是武汉菜用大豆混合群体(A291)家族, 为5.4个。10个重要家族群体PIC均值变化范围在0.670~0.810之间, 遗传多样性结果与根据等位变异数分析结果基本一致。综上所述, A034家族遗传多样性最高, A231家族其次, A291家族最低, 且各家族群体间遗传差异明显, 其中A034与A291家族间遗传差异最为明显(表3)。
| 表2 用于分析179个大豆品种的161个SSR标记所在连锁群及其多样性 Table 2 Position of 161 SSR markers on chromosomes and genetic diversity of 179 cultivars |
| 表3 10个重要家族群体的遗传多样性 Table 3 Genetic diversity of 10 important families |
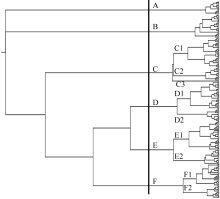
根据Nei’s (1983)遗传距离, 用Neighbor-Joining tree方法进行品种遗传关系聚类, 179个大豆品种可分为A、B、C、D、E和F等6个亚群, A、B、C1、C2、C3、D、E1、E2、F1、F2和F3等11小类。
179个品种的详细归类结果见附表。A亚群包括12个品种, 其中以铜山天鹅蛋(A231)家族的品种为主, 占7个, 主要分布在II生态区。B亚群包括21个品种, 其中以即墨油豆(A133)家族为主, 有17个, 分布的地区主要为II区的河南和山东等地。
C亚群包含品种较多, 共40个品种, 又可以C1和C2两小类及1个独立品种(豫豆29)。C1类包括沧豆4号等27个品种, 其中滨海大白花家族(A034)的品种有17个; 主要分布在北京、安徽和河南等黄淮地区(II); C2类包括12个品种, 以山东寿张县无名地方品种(A295)家族为主有10个品种, 主要分布在II生态区的河南省; C3仅包含一个独立的大豆品种豫豆29。
D亚群包括36个品种, 又可分为D1和D2两个小类。D1小类包括24个品种, 其中有23个品种分布在江苏省, 多属于III生态区, 该亚群以51-83 (A002)家族的品种为主, 占12个; D2小类的品种以即墨油豆(A133)家族为主, 12个品种中有7个属于该家族, 主要分布在II和IV生态区。
E亚群包括39个品种, 可分为E1和E2两小类。E1小类包括26个品种, 以滨海大白花家族(A034)、即墨油豆(A133)家族的品种为主, 分别有14和11品种聚在此类; 东北白眉(A019)家族的品种, 如绥农14和合丰33等也多聚在此类。E2类包括13个大豆品种, 其中有11个品种属于滨海大白花家族(A034), 分布地区集中在II和III生态区。
F亚群包括31个品种, 可分为F1和F2两小类。F1小类包括20个品种, 以上海六月白(A201)和武汉菜用大豆混合群体(A291)家族的品种为主。F2类包括11个大豆品种, 以上海六月白(A201)为主, 有7个, 该类品种主要分布在IV生态区。
2.3.1 亲本系数 179个育成品种成对组成的 15 931个组合中, 亲本系数平均值为0.02, 分布范围在0~0.75间(表4); 其中有7371个组合的亲本系数为0, 且总体有95%以上的亲本系数分布在0~0.13之间。以上说明, 尽管这些品种来自10个祖先亲本家族, 但其遗传基础仍相对较宽广。
| 表4 主要家族品种间亲本系数和遗传相似系数的比较 Table 4 Comparison of parentage coefficient and genetic similarity coefficient among cultivars in the families |
由表5和图2-A可知, 家族群体内, 寿张县无名地方品种家族(A295)、墨油豆(A133)家族、滑县大绿豆(A122)家族、铜山天鹅蛋(A231)家族、滨海大白花家族(A034)和白眉(A019)家族的亲本系数均值相对较小, 且分布较为集中。如A295家族的亲本系数平均值为0.04, 分布范围在0~0.50间, 99%以上分布在0~0.13间。
由表5可知, 主要家族群体间, 白眉家族(A019)与其他家族间的亲本系数均较小, 说明其遗传相似性低, 彼此拓宽遗传基础的可能性较大, 而奉贤穗稻黄家族(A084)与51-83家族(A002)间和菜用大豆混合群体家族(A291)与上海六月白家族(A201)间的亲本系数较大, 说明二者间遗传基础较窄。
从上述亲本系数数据分析数据可知, A291家族的亲本系数平均值最大, 其遗传基础最为狭窄, 其次为A002、A084和A201; 相对来说, A295和A133家族的遗传基础最为宽广。东北的白眉家族(A019)与其他家族亲本系数较小, 以后育种中要加强彼此间种质交流。
4个生态区中, II生态区品种家族较大、涉及祖先亲本数多, 其亲本系数平均值最低, 为0.04, 并较为紧凑地分布在平均值附近, 该区品种的遗传基础最为宽广; III生态区的品种家族自然选择育种较多, 涉及祖先亲本数少, 其亲本系数平均值最高, 为0.08, 且较分散, 该区品种的遗传基础最为狭窄(图2-B)。
| 表5 主要家族间亲本系数和遗传相似系数的比较 Table 5 Comparison of parentage coefficient and genetic similarity coefficient among the families |
51-83家族(A002)、奉贤穗稻黄家族(A084)和上海六月白家族(A201)的亲本系数居中, 分布相对分散。如A002家族品种间亲本系数平均值为0.12, 分布范围在0~0.56间, 74.5%密集分布在0~0.15范围间。武汉菜用大豆混合群体家族(A291)亲本系数均值最大, 为0.20, 分布最为分散, 0.06~0.28间居多, 63.0%分布0.13~0.28间。
2.3.2 遗传相似系数 179个品种成对品种间GSC分布见表6, 其均值为0.16, 分布范围在0.03~ 0.80间, 95%以上分布在0.07~0.22之间, 其中齐黄29与合豆3号间的遗传相似系数最小, 为0.03, 湘春豆23与湘春豆22间的遗传相似系数最大, 为0.80。品种间遗传相似性较小, 群体遗传基础便较广泛。
10个家族亚群内, 滨海大白花家族(A034)、即墨油豆(A133)家族和铜山天鹅蛋(A231)家族品种间遗传相似系数平均值最小, 均为0.16, 且分布较为集中; 如A034家族, 85.0%分布在0~0.20间, 以(0.10~ 0.20)最为密集。
51-83家族(A002)、奉贤穗稻黄家族(A084)、寿张县无名地方品种家族(A295)、白眉(A019)家族和滑县大绿豆(A122)家族的遗传相似系数均值居中, 分别为0.17、0.17、0.17、0.18和0.18, 且分布相对分散。如A002家族品种间遗传相似系数分布范围为0.04~0.61, 多数分布在(0.10~0.20)区间内, 79.0%集中于(0~0.20)区间。武汉菜用大豆混合群体家族(A291)和上海六月白家族(A201)品种间遗传相似系数最大, 分别为0.23和0.25, 且分布范围相对分散。如A201家族遗传相似系数分布在0.05~0.80间, 仅55.0%以上分布在0~0.20间。
10个重要家族群体间, 白眉家族(A019)与其他家族间的遗传相似系数均较小, 说明其遗传相似性低, 而菜用大豆混合群体家族(A291)与上海六月白家族(A201)间的亲本系数较大, 与基于亲本系数的分析结果相同(表5)。
| 表6 成对品种间遗传相似系数次数分布表 Table 6 Frequency distribution of GSC in pairwised soybean cultivars |
从上述遗传相似系数分析可知, A291和A201家族的遗传基础应最狭窄, A034、A133和A231的遗传基础最宽广; 东北的白眉家族与其他家族遗传相似系数均较低。上述结果与亲本系数分析结果一致。
4个生态区中, II生态区所包含的品种及家族最多, 平均遗传相似系数最高, 为0.16, 并较为紧凑分布在平均值附近, 所以该区品种的遗传基础最宽广; 其次为I生态区, 尽管该区仅包含A019家族, 但品种间遗传相似系数仍高达0.16, 这与其育种进程相对较快有关。IV生态区的品种家族遗传相似性系数平均值最高(GSC=0.21), 且分布较松散, 该区品种的遗传基础最狭窄, 这与该区的品种多经过选择育种相关。
2.3.3 亲本系数与遗传相似性系数间关系 表4列举了各主要家族的亲本系数和SSR遗传相似性系数情况, 家族亲本系数均值大小顺序为A133= A295
2.4.1 家族间种质互补 从成对家族来看, 补充等位变异数最多的是滨海大白花家族(A034)对武汉菜用大豆混合群体家族(A291), 为770个, 说明A291家族拓宽A034家族遗传基础的潜力最大; 最少的是滑县大绿豆(A122)家族对寿张县无名地方品种家族(A295), 为7个, 其他家族间的补充等位变异数见表7。亚群间互补等位变异数最多的是铜山天鹅蛋家族(A231)与武汉菜用大豆混合群体家族(A291)间, 为787个。
从整体来看, II生态区的滨海大白花家族(A034)、寿张县无名地方品种家族(A295)、铜山天鹅蛋家族(A231)分别对其他9个家族具有较多的补充等位变异总数, 所以这些家族具有拓宽其他家族遗传基础的较大潜力。I区的A019、III区的A201和A291分别与其他家族具有最多的互补等位变异总数, 所以其他7个家族具有拓宽这3个家族遗传基础的较大潜力。
2.4.2 各家族种质的特异性 特有等位变异是某群体具有而其他各群体都没有的等位变异; 特缺等位变异是某群体没有而其他各群体都有的等位变异; 特有、特缺等位变异表示了群体的特异性[ 17]。10个家族群体中, 东北的A019家族与其他9个家族地理距离最远, 特有、特缺等位点数最多, 其特有标记(特有等位变异数)是10(10)、103(145)个。南方的A002与黄淮海的A231和A122家族的育成品种主要分布于江苏、河南和山东等地家族间种质交流较多, 所以无特有等位变异。南方的A084、黄淮海的A034和A231来自江苏, 其向江苏南北交流, 因此这3个家族和南方的A201家族间无特缺等位变异(表8)。
| 表7 不同家族间互补等位变异数 Table 7 Number of complementary alleles among different families |
| 表8 不同家族群体间特有等位变异和特缺等位变异 Table 8 Specifically existent and specifically deficient alleles between different ancestor families |
现代大豆育种中, 由于大量使用相同的骨干亲本, 育成的大豆品种群体内的遗传基础非常狭窄。从10个重要家族群体的生态区分布来看, 相同家族的品种有主要分布在1个或少数几个区域的趋势, 如A019家族主要分布在I生态区, A133家族主要分布在II区, 但是不同生态区间也有少数的种质交流, 尤其是II与III生态区间, 如南方的A084家族、黄淮海的A034家族和A231家族。上述结果与盖钧镒等[ 29]通过对我国1923—1995育成品种的系谱资料分析后的研究结果相一致, 认为我国大豆育成品种的祖先亲本主要在本生态区使用, 异生态区间的种质交流利用较少, I区(东北)极少利用其他生态区的种质, 相对来说, II区(黄淮海)、III区(南方)稍好些。
同时, 本研究SSR标记聚类分析在分子水平上显示10个家族育成品种的遗传组成与其所在的地理分布有一定关系, 如D1小类的24个品种中, 有23个分布在江苏省。因此, 加强不同生态区间大豆种质的交流, 对拓宽大豆育种品种的遗传基础是十分必要的。今后我国大豆育种在使用现有祖先亲本时, 还应该注重其他生态区优异种质亲本筛选、发掘及应用。美国大豆育种的成功是很好地应用了国外和异地的种质, 这个经验值得中国借鉴。加强国内外优异种质在各生态区间的交流与应用应是行之有效的方法。问题是如何交流使用这些优异种质材料, 一方面可以驯化这些优异种质适应新的环境以便和当地品种杂交, 另一方面是建立一个由多个生态区优异祖先亲本组成的群体在各区进行轮回选择。无庸置疑, 今后大豆育种中, 亲本材料的改良比祖先亲本的研究更为重要。
本研究结果表明, 利用亲本系数和遗传相似系数评价群体遗传基础的结果相对一致, 而又不完全一致。这与亲本系数和遗传相似系数间的相关系数仅为 r= 0.67**相一致, 因为2个品种间的亲本系数是指该两品种携带相同的等位基因来自同一个亲本的概率, 遗传相似系数则仅指品种间携带相同的等位基因的概率但并不计较是否来自同一个亲本。前者称为传递相同(identical by descent, 或ibd)后者, 称为状态相同(identical by state, 或ibs)。前者用于研究2个品种间的亲缘关系, 后者用于研究2个品种间种质的相似程度, 有亲缘关系必然具有相似性, 但有相似性不一定有近缘关系。本研究结果, 亲本系数和遗传相似系数有相关, 而又不完全相关, 就是因为上述差异。一般在研究群体中品种间的亲缘关系时常用亲本系数做度量, 而在育种过程中作选择时则常用遗传相似系数做度量。亲本系数的计算必须知道品种间的系谱关系, 遗传相似系数则可以通过分子标记作估计。有时系谱关系难找, 无法估计亲本系数, 有人用遗传相似系数当作亲本系数, 显然这只是近似估计, 以本研究为例, 准确性只有 r=0.67。实际上亲本系数反映了整体基因流向, 是对基因组的宏观分析, 不考虑人工选择和遗传漂变等外界环境和育种条件的影响, 而遗传相似系数是对基因组受各种影响后的实测相似性程度, 而且受标记抽样误差的影响。所以亲本系数和分子标记方法在研究品种间遗传关系中各有特点[ 30]。本研究中群体亲本系数均值为0.02, 比Zhou等[ 2]分析86个大豆品种的结果低( CP=0.04), 与Cui等[ 1]分析651份中国大豆育种品种的亲本系数相当( CP=0.02), 表明大豆重要家族群体遗传基础广泛。根据亲本系数和遗传相似系数具有相关性的特点, 两者都可以对育种过程中的亲本组配方案提供指导。育种家可以用当地的优良品种为受体, 选择与其亲本系数或遗传相似系数较小的供体优先组配; 也可以根据SSR聚类结果, 选择与受体遗传距离较远的品种作为供体。
大豆主要家族群体遗传互补性与特异性分析表明, 不同群体间存在大量的互补等位变异, 如A231家族对A291家族具有787个互补等位变异, 占总等位变异数1697个的46.4%。同时不同群体间还存在着特有等位变异和特缺等位变异。这显示了不同家族间遗传交流的潜力。
作物育成品种对环境的适应能力是作物育种成败的关键。东北的A019家族与其他家族间遗传交流较少, 相比具有最多的互补等位变异、特有及特缺等位变异数。根据本文的结果, 为拓宽作物育种材料的遗传背景, 一方面要通过表型鉴定不断挖掘丰富的优异种质, 另一方面要利用分子标记技术, 分析比较作物种质间的相似和相异性, 从而对亲本进行基因型差异基础上的组配, 获得超亲的后代。
以我国10个大豆育成品种重要家族的179个品种为材料, 采用161个均匀分布于大豆基因组的SSR分子标记进行遗传多样性分析, 群体具有丰富的遗传变异, 161个位点上共检测到1697个等位变异, 每位点5~24个, 平均10.5个; 多态信息量(PIC)变化为0.549~0.937, 平均0.819。179个品种可归为6大类11小类, 同一家族的品种有聚为一类的趋势。亲本系数和SSR标记计算的相似系数间 r=0.67; 山东寿张县无名地方品种(A295)、即墨油豆(A133)、滑县大绿豆(A122)和铜山天鹅蛋(A231) 4个家族两者均较小, 其遗传基础最宽广; 矮脚早(A291)、上海六月白(A201)、奉贤穗稻黄(A084)和51-83(A002) 4个家族两者均较高, 遗传基础较狭窄。东北白眉(A019)家族和其他9个家族地理距离较远, 存在较多特有、特缺等位点, 而III区和II区地理位置较近, 种质交流较多, 两区祖先亲本家族间特有、特缺等位点数较少, 其中A002、A231和A122家族未发现特有等位变异, A084、A201、A034和A231家族未发现特缺等位变异。
| 附表 供试的179份大豆品种聚类结果 Supplemental Table Clusters of 179 soybean cultivars |
| (续附表) 供试的179份大豆品种聚类结果 Supplemental Table Clusters of 179 soybean cultivars |
| (续附表) 供试的179份大豆品种聚类结果 Supplemental Table Clusters of 179 soybean cultivars |
| (续附表) 供试的179份大豆品种聚类结果 Supplemental Table Clusters of 179 soybean cultivars |
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|




 Minsoy
Minsoy ×
×