第一作者联系方式: E-mail:zhoujinchao0230@163.com
以农家品种四粒红和冀农黑3号构建的包含有251个家系的重组自交系(RIL)群体为材料, 在河北保定市和邯郸大名县两地进行表型鉴定, 利用Pearson’s相关和逐步多元回归分析了花生农艺性状之间及其与标记的相关性。结果表明, 多数农艺性状间存在显著或极显著相关, 其中相关性最高的为单株生产力和单株仁重(
A recombinant inbred line (RIL) population including 251 lines, derived from the cross between Silihong and Jinonghei 3 was used to study the correlations between SSR markers and agronomic traits. The phenotypese were evaluated in two locations of Baoding and Handan, Hebei Province. Pearson’s correlation and stepwise multiple linear regression analysis were conducted based on SSR and phenotyping data. The results showed that there were significant correlations among 18 agronomic traits, with the higher correlations between pod weight per plant and seed weight per plant (
作物的许多农艺性状如产量、品质和抗逆性等大多属于数量性状, 遗传基础复杂, 易受环境影响。通过表型选择的传统育种方法改良农艺性状周期长, 选择效率低[ 1]。随着分子生物学的飞速发展, 分子标记技术得到广泛应用, 通过筛选与目标性状紧密连锁的分子标记, 即分子标记辅助选择(marker-assisted selection, MAS)对目标性状进行改良, 可以加快育种进程, 提高育种效率[ 2]。众多分子标记技术中, 简单序列重复标记技术(simple sequence repeat, SSR)以其检测容易、重复性高和稳定性好等优点已被广泛用于作物目标性状的遗传改良研究[ 3]。由于花生基因组学研究如遗传图谱构建和基因定位等进展缓慢, 加之其栽培种间基因组高度保守, 导致花生品种间遗传多样性偏低。因此, 仍有学者认为分子育种, 如MAS很难在短期内用于花生各种性状的改良[ 4]。基于上述花生分子遗传改良存在的问题, 使得鉴定标记与农艺性状间的连锁关系成为花生分子育种的必要手段。
目前, 利用覆盖全基因组的分子遗传连锁图和合适的分离群体进行连锁分析以及数量性状位点(quantitative trait locus, QTL)定位是解析植物数量性状遗传基础的主要方法[ 1], 并且在花生中已被广泛应用[ 5, 6, 7, 8, 9, 10, 11, 12]。然而, 通过分离群体筛选目标性状的QTL存在一定的局限性, 由于不同实验室采用的群体类型、标记种类及数量和环境等诸多因素的干扰, 导致QTL定位结果重复性较低[ 13]; 其次, QTL定位需要预先构建高分辨率连锁图谱, 而后才能进行标记与性状的连锁分析。前人的研究结果大都是通过复合区间作图法将目标性状相关QTL定位在包含2个或2个以上位点的标记区间。采用多元线性逐步回归的统计方法, 依据测验值的显著水平确定标记与性状间是否相互关联, 从而估算出单个标记对性状的贡献(即决定系数), 该方法已在水稻、棉花及白桦等植物中应用。例如, 徐云碧等[ 14]在研究水稻农艺性状时, 计算了性状与标记间的相关系数, 在显著( P=0.05)和极显著( P=0.01)水平上获得了4个标记-性状的关联, 表明相关系数的方法同样可用于数量性状位点的分析和鉴定。Wu等[ 15]在研究棉花的农艺性状时发现, 通过标记与性状的多元线性逐步回归分析, 获得50个AFLP标记与7个农艺性状呈显著相关, 其中1/4标记的贡献率在22%~93%之间。魏志刚等[ 16]通过RFLP标记与性状值间的多元回归分析, 筛选出与白桦纤维长度显著相关的分子标记, 并成功应用于白桦纤维长度分子辅助育种。
多元线性逐步回归法可以快速建立标记与性状之间的相关, 在花生分子育种研究上应用较少。因此, 本研究采用关联遗传学的研究方法, 筛选与农艺性状显著相关的SSR标记, 以期应用于花生农艺性状的分子标记辅助育种。
以农家品种四粒红为母本, 黑花生品种冀农黑3号为父本杂交, 经过连续多代自交, 构建了包含251个家系的重组自交系群体。
1464对SSR引物序列来自Peanut Marker Database (http://marker.kazusa.or.jp/Peanut/marker/list/), 由生工生物工程(上海)有限公司合成。
取鲜嫩叶片约1 g, 采用CTAB法提取基因组DNA[ 17]。在提取液中加入10% 3 mol L-1的NaAc, 以降低样品糖分含量。用0.8%琼脂糖凝胶电泳检测DNA浓度和纯度, 并将母液浓度稀释至25 ng μL-1, -20℃保存备用。
PCR扩增根据崔顺立等[ 18]的方法并对其优化。PCR体系为15 μL, 含25 ng μL-1模板2.5 μL, Premix Taq Version 2.0 (TaKaRa) 7.5 μL, 10 μmol μL-1正、反引物各0.7 μL, ddH2O 3.6 μL。反应程序如下: 94℃预变性5 min; 94℃变性45 s, 55℃复性45 s, 72℃延伸 1 min, 35个循环; 最后72℃延伸10 min; 4℃保存。PCR产物经8%非变性聚丙烯酰胺凝胶电泳, 检测标记的多态性。
2012年分别在河北农业大学育种中心(保定)和邯郸两地对构建的RIL群体进行种植和表型鉴定。行长1 m, 行距40 cm, 株距10 cm。重复1次, 管理同大田。依据姜慧芳等[ 19]田间调查方法和标准。从每个株行随机取3株, 测定主茎高(height of main stem, HMS)、第一侧枝长(length of first branches, LFB)和总分枝数(total branching number, TNB)。从每个单株选择3个花生测定荚果长(pod length, PL)、荚果宽(pod width, PW)、荚果厚(pod thickness, PT)、种子长(seed length, SL)、种子宽(seed width, SW)、种子厚(seed thickness, ST)、荚果网纹(pod reticulation, PR)、果壳厚度(thickness of pod shell, TPS)。统计收获的3个单株的单株结果数(pod number per plant, PNP)、单株多粒果数(multi-seed number per plant, MSNP)、单株生产力(pod weight per plant, PWP)和单株仁重(seed weight per plant, SWP)等性状。其中, 选取5个典型饱满荚果, 测量果脊两侧最宽处为荚果宽, 取平均值; 测量果脊到腹缝的长度为荚果厚, 取平均值。测量两子叶最宽处为种子宽, 取平均值。测量两条缝之间最厚处为种子厚, 取平均值。测量果壳厚度与去纹果壳厚度之差为荚果网纹, 取平均值。统计收获的3个单株的多粒果数取平均值为单株多粒果数。
采用Pearson’s相关分析( P≤0.05, P≤0.01), 计算各农艺性状之间的相关系数和性状与标记的关联程度。利用与农艺性状显著相关的SSR分子标记进行多元线性回归分析, 各个性状作为因变量, 各标记产生的基因型作为自变量。依据Kraakman等[ 20]的逐步回归程序, 选择线性分析中的标记。本研究所有数据均由SPSS 17.0软件统计分析。
四粒红和冀农黑3号间差异幅度较大、变化范围广且呈连续分布, 其中四粒红变异系数范围为3.67%~76.61%, 冀农黑3号变异系数范围为0~53.00%。各家系差异明显且存在超亲现象, 其变异范围为8.38%~87.73% (表1)。
| 表1 亲本及RIL群体农艺性状的遗传变异 Table 1 Variation of agronomic traits in parents and RIL population |
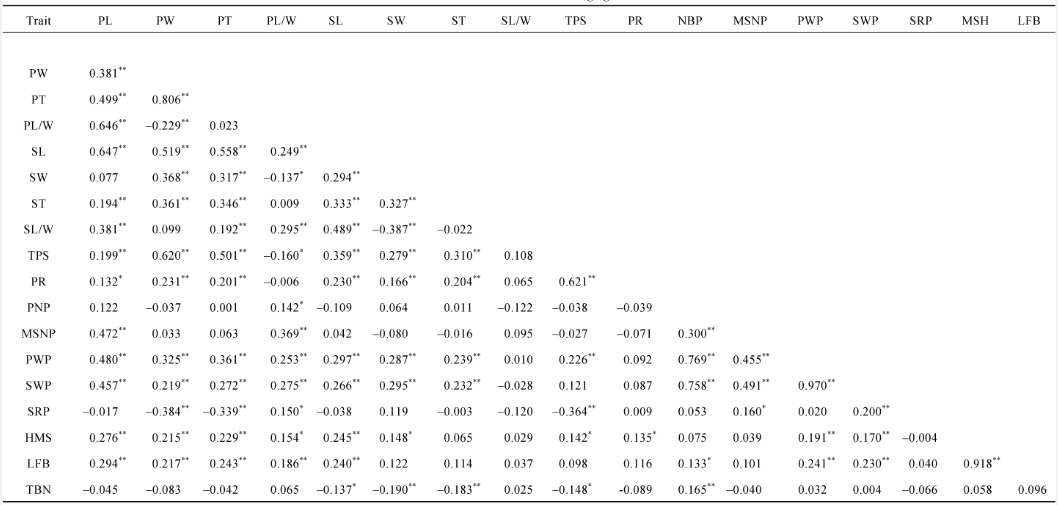
由表2可以看出, 荚果长与荚果宽、荚果厚度、荚果长宽比、种子长、种子厚度、种子长宽比、果壳厚度、单株多粒果数、单株生产力、单株仁重、主茎高和第一侧枝长呈正相关且达到极显著水平( P≤0.01), 而与荚果网纹显著相关( P≤0.05)。其中相关性最高为单株生产力和单株仁重( r=0.970), 其次是主茎高和第一侧枝长( r=0.918), 荚果宽和荚果厚度( r=0.806)。10个农艺荚果性状均与单株生产力呈不同程度极显著正相关。荚果宽与荚果厚( r=0.806)、果壳厚度( r=0.620) 和种子长( r=0.519)呈显著正相关。单株结果数、单株多粒果数和单株生产力分别与单株仁重( r=0.758, r=0.491, r=0.970)呈极显著正相关。单株生产力与单株仁重分别与主茎高、第一侧枝长呈极显著正相关。单株结果数与总分枝数呈极显著相关( r=0.165)。荚果长/宽、果壳厚度、单株多粒果数均与产量性状(果重、仁重、出仁率)呈不同程度的极显著正相关。由此看出, 多个农艺性状都对单株生产力有影响, 对这些性状的直接选择, 将对单株生产力有所改变[ 21]。
| 表2 农艺性状间相关性系数 Table 2 Correlation coefficients among agronomic traits |
经过1464对SSR引物的双亲多态性筛选, 获得多态性引物139对, 多态率为10.5%。SSR标记和农艺性状的相关分析表明(表3)多个标记与单一性状相关, 其中, 2个与荚果长相关, 14个与荚果宽相关, 12个与荚果厚相关, 2个与荚果长宽比相关, 5个与种子长相关, 12个与种子宽相关, 4个与种子厚相关, 5个与种子长宽比相关, 14个与果壳厚度相关, 2个与荚果网纹相关, 6个与单株结果数相关, 9个与单株多粒果数相关, 7个与单株生产力相关, 4个与单株仁重相关, 16个与单株出仁率相关, 13个与主茎高相关, 12个与第一侧枝长相关, 6个与总分枝数相关。另外, 部分SSR标记与多个农艺性状相关联。标记AhTE0561与荚果宽( r=0.164)、荚果厚( r=0.163)、果壳厚度( r=0.185)、第一侧枝长( r=0.170)、单株生产力( r=0.129)和主茎高 ( r=0.155)均呈显著正相关, 推测携带标记AhTE0561的花生品种, 其荚果宽、荚果厚、果壳厚度、单株生产力、第一侧枝长和主茎高等性状将有一定程度的提高。标记GM2638与荚果网纹( r= -0.158)、单株结果数( r= -0.140)、单株生产力( r= -0.163)和单株仁重( r= -0.168)均呈显著负相关, 推测携带标记GM2638的花生其网纹比较浅, 单株结果数、单株生产力和单株仁重将会相对较低。
| 表3 部分SSR标记和农艺性状之间的相关系数 Table 3 Correlation coefficients between partial SSR markers and agronomic traits |
获得了14个标记与13个农艺性状关联, 贡献率 R2的范围在5.2%~11.5%之间(表4)。其中标记ARS754、Ah3TC38C01对花生荚果长所解释的总变异范围为5.2%~8.5%, 标记GM2638对荚果宽和单株出仁率所解释的变异分别为6.2%和7.7%, 标记GM1996、S54和seq2E06对荚果厚所解释的变异的范围是5.4%~6.1%, 标记GM2137对荚果长/宽、种子长和种子宽所解释的变异分别为11.5%、9.6%和7.7%。标记Ah3TC38C01对花生荚果网纹所解释的变异为5.4%, 标记GM1878、RM13A12对果壳厚度所解释的总变异为5.5%~10.0%。与产量密切相关的标记seq9A7_2、gi_0832和GM2638分别对单株生产力、单株仁重和单株出仁率所解释的总变异为6.5%、6.9%和7.7%, 与花生植株性状相关的标记AhTE0561对第一侧枝长所解释的表型变异为5.7%。标记GM1878、pPGSseq18C5、RM13A12和gi_0832对总分枝数所解释的表型变异范围为6.1%~ 8.9%。表2中与性状显著相关的SSR标记与逐步回归分析后的部分标记部分一致。例如, 标记AhTE0561与第一侧枝长, 标记pPGSseq18C5和总分枝数, 标记TC11A04和种子宽, 标记RM13A12和果壳厚度均呈极显著相关, 同样还有标记seq9A7_2与单株生产力, 标记GM1878与总分枝数呈显著相关。未获得与种子厚、种子长宽比、单株结果数、单株多粒果数和主茎高相关的SSR标记。
| 表4 SSR标记和农艺性状之间的多元逐步回归分析 Table 4 Stepwise multiple regression analysis between SSR markers and agronomic traits |
许多复杂的数量性状可通过数理统计分析法将其剖分为若干离散的孟德尔因子进行研究, 进而将数量性状位点(QTL)锚定于染色体的特定位置, 并确定其与周围基因或标记间相互关系。利用单一标记为基础的方法检测QTL时, 常用的分析统计指标为均值或方差, 通过比较基因型位点表达性状的均值或方差, 对均值进行 t测验或对方差进行 F测验, 依据测验值的显著水平确定标记与性状间相互关联的可能性。Wu等[ 15]通过标记与性状的多元线性逐步回归分析, 获得50个AFLP标记与7个棉花农艺性状显著相关, 其中1/4标记的贡献率在22%~93%之间, 此结果与其QTL研究吻合。杨鑫雷等[ 22]通过标记与性状的逐步多元回归分析, 建立了两者的关联关系, 并且所解释的总变异率最高达30%。例如, 标记EM_245与多个性状存在显著的相互关联, 并且与杨鑫雷等[ 22]的QTL定位结果一致。由此说明, 采用多元逐步回归分析法可快速、准确地分析标记与性状的关联关系。本研究利用相同的统计方法, 表明农艺性状之间存在显著或极显著相关, 相似性程度最高的为单株生产力和单株仁重, 相关系数为0.970。77个SSR标记与18个农艺性状显著或极显著相关, 标记数量最多的性状为单株出仁率, 最少的为荚果长、荚果长宽比和荚果网纹, 变化范围为2~16个。Pearson’s相关分析表明, 1~2个标记能够影响多个性状的表现, 并与性状建立显著的相关, 进而通过标记与性状的多元逐步回归分析, 建立两者的关联关系, 并且所解释的总变异率的变化范围为5.2%~11.5%。
与目前较为广泛应用的基于最大似然统计的复合区间作图法相比, 逐步多元线性回归方法是利用标记与性状的回归分析, 通过决定系数( R2)的大小, 来确定标记与性状的紧密程度, 可快速确定标记与性状是否存在显著关联关系, 消除复合区间作图时QTL区段两侧标记的影响, 提高QTL定位的准确性。此方法可以对任何单独的分子标记进行相关性分析, 无需构建高密度的遗传连锁图谱, 就可以找到与目标性状相关联的标记, 可用于MAS辅助选择育种, 提高优良性状的选择效率, 缩短育种进程, 为花生的遗传改良提供理论依据。随着遗传连锁图谱标记的不断增加, 这类方法的效率也将有很大的提高, 将成为育种家普遍接受的研究方法。
18个农艺性状间存在不同程度的相关性, 其中10个农艺性状与产量相关性状极显著相关。77个SSR标记与18个农艺性状相关, 每个性状相关的标记数在2~16之间。标记与农艺性状的多元逐步回归分析获得14个标记与13个性状关联, 贡献率范围为5.2%~11.5%, 为花生农艺性状的分子标记辅助选择奠定了基础。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|