以陆地棉
Using upland cotton
病毒介导的基因沉默(virus-induced gene silencing, VIGS)是植物体内普遍存在的一种遗传免疫机制, 属于转录后基因沉默(post-transcriptional gene silencing, PTGS)[ 1]。当携带含有内源基因片段的病毒载体侵染寄主植物后, 能够激活植物自身的免疫系统; 在识别并降解病毒RNA的同时也会产生含内源目的基因的microRNA, 这些microRNA与靶基因的mRNA结合, 之后被Dicer酶降解, 从而使目的基因表达水平下降或功能丧失[ 2]。其中由烟草脆裂病毒(tobacco rattle virus, TRV)诱导的基因沉默(TRV- VIGS), 以其沉默效率高、持续时间长, 寄主植物病毒症状轻, 不会掩盖沉默表型, 且在各种组织均可产生基因沉默等优点, 成为目前应用最为广泛的一类基因沉默体系。TRV-VIGS已在茄属的番茄、烟草、辣椒[ 3, 4, 5]、拟南芥[ 6]、麻风树[ 7]、矮牵牛[ 8]、罂粟[ 9, 10, 11]等植物上成功应用。
植物 CLA 1基因( cloroplastos alterados 1 gene)编码1-deoxyxylulose 5-phosphate synthase, 参与叶绿体发育过程, 进化中高度保守, CLA1基因突变体 cla1-1有明显的白化表型[ 12], 是易于识别的标记性状。棉花是世界性重要的经济作物。近年来, 已有TRV-VIGS体系构建与应用的相关报道。以雷蒙德氏棉 CLA1基因序列设计目标沉默片段, Gao等[ 13]建立了基于农杆菌介导的棉花TRV-VIGS体系, 并在不同遗传背景的6个棉花品种材料中成功应用, 为棉花重要基因功能的鉴定奠定了基础。进一步通过TRV病毒抑制棉花抗黄萎病相关基因 GhNDR1和 GhMKK2表达, 使棉花受体更易感病, 快速鉴定了基因功能[ 14]。Qu等[ 15]发现TRV-VIGS不仅可用于沉默营养器官中基因, 还可用于沉默纤维等组织和生殖器官如花蕾中的基因。然而, TRV-VIGS能否广泛应用于棉花各品种, 即该体系是否受寄主基因型的影响还不清楚, 未见相关研究报道。近年来, 棉花基因组研究取得快速进展, 大量的棉花表达序列信息已在公共数据库释放(http://www.ncbi.nlm.nih.gov/dbEST/), 与四倍体棉种D基因组供体亲缘关系最近的雷蒙德氏棉, 已完成全基因组测序及序列分析[ 15, 16], 大量棉花重要基因的功能需进一步鉴定。建立基于目标性状的不同棉花受体基因瞬时干扰表达体系, 将加速棉花功能基因组学研究进程, 快速获得目标性状的候选功能基因。
本研究以拟南芥 AtCLA1基因为探针, 搜索雷蒙德氏棉( G. raimondiiUlbrich)全基因组序列[ 16], 获得 CLA1基因序列, 进一步设计引物, 通过同源克隆法从陆地棉遗传标准系TM-1中克隆出 CLA 1基因, 命名为 GhCLA1。构建了VIGS-GhCLA1载体, 以海7124为受体材料, 建立了棉花的TRV-VIGS体系。选择不同来源及不同生态区的34份棉花材料, 进行沉默体系重复试验。通过TRV-VIGS技术抑制受黄萎病诱导表达的 GhMAPKKK基因的表达, 发现海7124对黄萎病的感病性显著增强。与通过棉花遗传转化来研究基因功能相比, TRV-VIGS具有无基因型选择性、快速(当代可观察表型)和高通量等特点, 将在棉花基因功能研究中扮演重要角色。
34份棉花品种(系), 包括33份陆地棉材料和1份海岛棉抗黄萎病品种海7124 (表1)。33份陆地棉材料包括: 黄河流域棉区11个, 长江流域棉区8个, 西北内陆棉区9个, 北部特早熟棉区2个; 感黄萎病品系军棉1号, 抗黄萎病品系60182, 以及陆地棉遗传标准系TM-1。TRV-VIGS体系的构建以及 GhMAPKKK的功能研究均以海7124为研究材料。同时诱导34份材料 GhCLA1基因沉默, 观察叶片的白化情况, 以研究TRV病毒在不同遗传背景材料中的适用性。
pTRV1和pTRV2 VIGS载体由清华大学刘玉乐教授惠赠。农杆菌GV3101由本实验室保存。MES (Sigma原装)及卡那霉素、利福平、乙酰丁香酮等主要国产生化试剂, 均购自南京奥斌生物科技有限公司。
| 表1 供试的34份棉花品种(品系)基本信息 Table 1 Information on 34 tested cotton varieties (lines) |
参考Velásquez等的制备方法[ 17]。以pTRV1和含有目的基因片段的pTRV2分别转化农杆菌GV3101。挑取新鲜培养的含pTRV1和目的基因片段的pTRV2的农杆菌GV3101单菌落, 分别接种到3 mL LB液体培养基(Kan, 50 μg mL-1; Rif, 50 μg mL-1)中, 28℃ 180转 min-1培养24 h; 以1%含量接入50 mL LB液体培养基 (Kan, 50 μg mL-1; Rif, 50 μg mL-1)中, 28℃ 180转 min-1培养12 h, 至菌液OD600为1.0左右; 1700× g离心5 min收集菌体细胞, 以适当体积的重悬液(10 mmol L-1 MgCl2, 10 mmol L-1 MES以及200 μmol L-1乙酰丁香酮)重悬至终浓度为0.8 (OD600); 将重悬液于室温下静置3 h以上, 含有pTRV1和目的基因片段的pTRV2的重悬液按体积比1∶1混匀, 用于注射棉花子叶。
以人工气候室培养到出苗后8 d, 子叶完全展平的棉花幼苗为试验接菌材料。先用注射器针头轻轻刺破子叶背面造成微伤口, 用去针头的注射器从伤口处注入1∶1混匀的含有pTRV1和含目的基因片段的pTRV2重悬液。于昼/夜温度23/22℃, 光/暗周期16 h/8 h的人工气候室内培养侵染后的棉花植株。在相同的环境下培养未经侵染处理和注射空载体(1∶1混和pTRV1和pTRV2)的对照植株, 2~5周观察不同处理棉花的表型, 并检测目的基因的表达情况。对每种材料处理10个单株。
2.1.1 棉花 CLA1基因的克隆及pTRV2-GhCLA1载体构建 以NCBI中释放的拟南芥 CLA1基因序列(GenBank登录号为AT4g15560)为探针, 搜索雷蒙德氏棉( G. raimondiiUlbrich)全基因组序列[ 15], 获得其序列信息(Gorai.011G184200.2), 进一步设计引物, 从陆地棉遗传标准系TM-1中克隆 GhCLA1基因 (GenBank登录号为KJ123647)。其ORF全长2163 bp, 编码720个氨基酸。序列相似性分析表明, 该基因与拟南芥 CLA1基因的氨基酸相似性为83.0%。而来自雷蒙德氏棉和陆地棉TM-1中的 CLA1同源基因其核苷酸和氨基酸序列相似性均达99%以上, 非常保守。进一步设计PCR引物, 并在引物的5'端分别加入限制性内切酶 EcoR I和 Kpn I的酶切位点和保护碱基(表2), 扩增位于棉花 GhCLA1基因ORF中部994~ 1414 bp基因片段, 目标扩增片段长度为421 bp。经测序验证后, 将该基因片段插入沉默载体pTRV2, 获得重组载体pTRV2-GhCLA1。
| 表2 本研究所用引物 Table 2 Primers used in this study |
2.1.2 棉花TRV-VIGS体系的建立 以未注射农杆菌及注射含pTRV1/pTRV2空载体的农杆菌混合液的植株为对照, 用含有pTRV1/pTRV2- GhCLA1的农杆菌混合液侵染海岛棉海7124出苗8 d的子叶, 两周后观察真叶表型。结果表明注射pTRV1/pTRV2空载体的幼苗叶片未呈现白化, 表型与未经侵染的对照无异。而注射pTRV1/pTRV2-GhCLA1植株, 其真叶表现为叶脉及边缘部白化, 逐渐扩散到整个叶片; 从第2片真叶起, 白化现象明显, 新生叶几乎完全白化(图1)。
为检测TRV在棉花植株中的扩散情况, 提取注射pTRV1和pTRV2空载体3周后的棉株根、茎、叶组织RNA, 利用TRV-RNA2特异引物将病毒RNA2反转为cDNA, 并设计特异性引物扩增病毒RNA2序列片段, 以检测病毒在棉花不同组织器官的扩散。RT-PCR分析表明子叶接种病毒后, 病毒RNA在棉花根、茎、叶中均有积累(图2), TRV病毒侵染棉花后可扩散到棉花多种组织器官中, 为利用该体系高通量验证根、茎、叶等组织器官优势表达目标基因功能提供了依据。
对pTRV1/pTRV2-GhCLA1处理3周后, 白化表型明显的植株和仅空载体侵染处理的对照植株, 提取叶片总RNA, 通过半定量RT-PCR的方法检测 GhCLA1基因的表达水平。结果表明, 与对照相比, 侵染pTRV1/pTRV2-GhCLA1的植株 GhCLA1基因几乎不表达, 沉默效果明显(图3)。
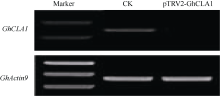 | 图3 半定量RT-PCR分析 CLA1基因沉默的效果Fig. 3 CLA1 expression in silenced and non-silenced cotton plants detected by semi quantitative RT-PCR |
2.1.3 棉花TRV-VIGS体系利用的广谱性 选择不同来源, 不同生态区的34份棉花材料, 进行沉默体系重复。以注射pTRV1/pTRV2空载体的植株为对照, 用含有pTRV1/pTRV2-GhCLA1的农杆菌混合液注射34份供试棉种子叶完全展平的幼苗, 诱导棉花 CLA1基因沉默。侵染3周后统一调查第2片真叶白化表型(图4)。发现在供试的不同棉种材料中, 其 CLA1抑制程度有差异, 如新陆早1号、朝阳棉1号、TM-1以及海7124等整个叶片白化, 中棉所12、晋棉25、豫棉20及华抗棉1号等叶脉明显白化, 叶肉轻度失绿。说明TRV病毒可应用于所有供试材料的 CLA1基因沉默, 但不同的棉花品种对TRV病毒的敏感性存在差异。利用TRV-VIGS技术诱导目的基因沉默受体遗传背景影响较小, 可广泛用于来源不同棉花资源材料中目标基因的功能鉴定。
 | 图4 34份不同来源棉花品种/品系TRV-VIGS诱导 GhCLA1基因沉默表型编号对应的材料见表1。每一材料上方为注射空载体对照, 下方为沉默 GhCLA1基因的叶片。Fig. 4 Photobleaching phenotype of GhCLA1silenced cotton plants in 34 cotton varieties (lines)Code numbers of cotton varieties correspond with these in Table 1. In each column, the upper is the leaves of vector control plants and the lower is those of GhCLA1 silenced plants. |
在MAPK信号通路中包含至少3个顺序的激活蛋白激酶, 依次为MAPKKKs、MAPKKs及MAPKs[ 21]。MAPKKKs参与了病原物侵染植物后引起的过敏性反应[ 22]。 GhMAPKKK(GenBank登录号为FJ966894)是棉花MAPKKKs家族中的一个成员, 本研究表明该基因受黄萎病菌诱导表达。以生长到二叶一心期的海7124幼苗为受体, 接种黄萎病菌株Vd8, 分别检测接种0、24、48、96和144 h后根组织中 GhMAPKKK基因的表达水平。RT-PCR分析表明, 随着接种时间增加, GhMAPKKK基因表达明显增强, 接种后96 h表达量达到高峰, 接种后144 h表达量开始降低(图5)。说明 GhMAPKKK参与棉花抗黄萎病信号传导途径。
 | 图5 GhMAPKKK在黄萎病菌诱导下的表达特征Fig. 5 Expression patterns of GhMAPKKK induced by Verticillium dahliae strain Vd8 |
利用TRV-VIGS体系进一步验证了 GhMAPKKK在黄萎病抗性中的功能。选择 GhMAPKKK基因靠近3'区1519~2077 bp碱基位置, 基因专化性强, 扩增出556 bp的目标片段, 克隆到pTRV2载体中获得重组质粒pTRV2-GhMAPKKK。用含有pTRV1/pTRV2- GhMAPKKK以及pTRV1/pTRV2-GhCLA1和pTRV1/ pTRV2空载体的农杆菌重悬液, 分别侵染海岛棉中抗黄萎病栽培品种海7124出苗8 d、子叶展平的幼苗。3周后, 注射TRV2- GhCLA1的植株出现明显白化。提取pTRV2-GhMAPKKK处理以及注射空载体的对照植株叶片总RNA, 通过半定量RT-PCR的方法检测表明, 与对照植株相比, 侵染pTRV2- GhMAPKKK的植株 GhMAPKKK基因的表达水平显著降低, 沉默效果明显(图6)。
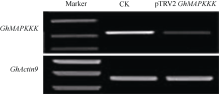 | 图6 半定量RT-PCR分析 GhMAPKKK基因沉默效果Fig. 6 GhMAPKKKexpression in silenced and non-silenced cotton plants detected by semi-quantitative RT-PCR |
对pTRV2-GhMAPKKK处理以及注射空载体的对照海7124植株, 接种大丽轮枝菌菌株Vd8。一个月后观察黄萎病的发病情况。通过3次生物学重复分析, 注射空载体的对照海7124植株, 其平均病情指数为21.9, 而 GhMAPKKK基因沉默后的海7124植株感病性增强, 其平均病情指数达41.25。表明 GhMAPKKK基因参与了黄萎病诱导的过敏反应(图7)。
在病毒诱导的基因沉默中, 最早使用的沉默载体是以烟草花叶病毒(tobacco mosaic virus, TMV)和马铃薯病毒(potato virus X, PVX)为基础的, PVX比较稳定但是宿主范围小, TMV宿主范围广但不稳定。TMV和PVX系统的缺陷还在于二者均能表现出病毒本身产生的症状, 干扰那些能产生轻微表型的基因, 使结果不易区别[ 23]。同时这2种病毒在宿主植物的生长点和分生组织中不易扩散, 影响目标基因沉默的整体效果。而以烟草脆裂病毒(tobacco rattle virus, TRV)为基础的病毒载体具有明显的优势, TRV具有更广泛的宿主范围, 可以在包括植株生长点和分生组织的所有组织器官内扩散, 病毒侵染植株后的症状与其他病毒相比更加轻微。本研究以完全展平子叶的棉花为受体, 成功建立棉花病毒诱导基因沉默体系, 该体系具有稳定性和广谱性, 为快速高通量研究棉花基因功能奠定了基础。
构建pTRV-VIGS载体时需要考虑以下3个方面: (1)插入载体的目的基因片段的大小。要求适中, 以300~500 bp为宜。TRV病毒载体插入基因序列长度上限为1.5 kb, 若超过1.5 kb病毒不易在植物体内扩散或者丢失目的片段, 影响沉默效果[ 24]。(2)选择合适的目的基因区域插入载体。如果是单拷贝基因, 其ORF中的任何区域都可以用来作为插入片段, 若是多拷贝或家族基因, 则要根据研究目的选择基因区域。如果专化沉默某一家族中的一个基因, 应选择非保守区域或基因非翻译区(UTR)来区别高度保守的基因, 如果两基因的同源性达到85%以上就可能被同时沉默。如果同时沉默家族基因中功能相似的基因, 克服功能冗余, 则可选择高度保守区[ 25, 26]。(3)环境因素影响VIGS效果。温度是对沉默效果影响最大的因素之一, 适宜的温度有利于提高基因沉默的效率, TRV介导基因沉默的最佳温度为22℃左右[ 27]。本研究中, 克隆并选择棉花标记基因 GhCLA1插入载体的片段大小为421 bp, 目标验证基因 GhMAPKKK插入载体的片段大小为556 bp。选择播种后两周左右, 子叶完全展开而真叶尚未伸出时是接种病毒的最佳时期。同时农杆菌侵染棉花后, 选择23℃左右的生长温度, 沉默效果好。
陆地棉是异源四倍体, 基因组预测分析显示, 大约含7万个基因[ 28], 而2012年释放的四倍体棉种亲缘关系最近的D基因组供体种―雷蒙德氏棉, 其基因组中约含3.7万基因[ 16], 需要建立高通量、准确、易操作体系快速揭示基因功能。基于下胚轴的棉花组织培养转基因再生体系周期长, 受基因型影响大。建立的VIGS技术具有简单、快速、高通量、不受遗传背景影响等优点, 利用TRV-VIGS体系高通量研究棉花基因功能, 特别是快速查明表型明显, 作用大的重要质量性状基因, 与棉花生长发育、抗病抗逆等密切相关基因, 以及关键代谢通路基因, 将为目标性状基因的快速发掘与育种应用提供重要基因资源与依据。Gao等[ 14]通过TRV病毒抑制棉花抗黄萎病相关基因 GhNDR1和 GhMKK2表达, 阐明了其在棉花抗病研究中利用潜力。Gao等[ 29]通过棉花黄萎病菌侵染后蛋白组学差异表达分析, 结合差异表达基因的VIGS功能验证, 揭示棉酚、油菜素类酯、茉莉酸等对棉花抗黄萎病性具有重要作用。本研究利用建立的棉花TRV-VIGS体系, 证实沉默 GhMAPKKK基因后, 抗黄萎病的海岛棉海7124植株变得更易感病, 说明 GhMAPKKK基因参与了黄萎病诱导的过敏反应, 在棉花黄萎病抗病机制中发挥作用。
利用烟草脆裂病毒载体建立了基于病毒诱导的棉花基因沉默体系。利用该体系, 病毒可高效扩散到受体的根、茎、叶等器官, 受棉花不同遗传背景影响小。应用该体系, 成功抑制了受黄萎病菌显著上调的棉花 GhMAPKKK基因表达。与对照株相比, GhMAPKKK抑制表达的棉花植株接种黄萎病菌后, 更易感病。建立的棉花TRV-VIGS体系具有广谱性、灵敏性和高通量等特点, 将加速棉花重要基因鉴定和功能分析研究进程。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|





