bHLH类转录因子在植物叶片形态建成和发育过程中发挥重要的生理功能。本文通过结球甘蓝转录组数据分析, 筛选出莲座期和结球期的茎尖间与叶片间均显著差异表达的2个bHLH类转录因子, 命名为
Basic helix-loop-helix (bHLH) transcription factors play an important role in the leaf morphogenesis and leaf development. In this study, through transcriptome analyses, two bHLH transcription factor genes, differentially expressed in leaves and shoot tips between cabbage rosette stage and heading stage, were screened, named
结球甘蓝( Brassica oleracea var. capitata L.)属于十字花科芸薹属植物, 营养生长阶段经历幼苗期、莲座期和结球期。每个时期均具有明显的形态特征, 其中莲座期主要进行莲座叶的平展扩张生长, 结球期主要进行球叶的分化和叶片向上向内弯曲生长, 球叶呈现抱合状态, 形成叶球[ 1]。球叶数目和卷曲程度直接决定结球甘蓝重量和结球紧实度2个重要的农艺性状, 同时球叶也为研究叶片卷曲提供了一个较为理想的材料。球叶的分化、向内弯曲生长以及叶球的形成过程受光照、温度、碳素营养以及叶片内源激素不均衡分布等多种因素的影响, 是环境因子影响下的复杂的形态学反应[ 2]。目前人们对结球甘蓝叶片卷曲和叶球发育的分子机制还知之甚少。
研究表明TEOSINTE BRANCHED1 CYCLOIDIA PCF (TCP)蛋白是一种植物体特异存在的蛋白, 参与多种生长发育过程[ 3]。金鱼草 cin突变体中发现一类TCP蛋白, 能使平展叶片边缘皱缩卷曲[ 4]。拟南芥microRNA介导 TCP4基因信使RNA的分裂来阻止TCP4正常表达, 进而控制叶片的发育, 产生叶片的不正常卷曲[ 5], 随后和TCP4同属于家族II、具有相似功能的其他 CINCINNATA ( CIN)基因 TCP2、 3、 10、 24又相继被报道[ 6, 7]。此外, 一些非家族II的 TCP基因在叶片发育过程中对叶片边缘的扩张、卷曲也有重要影响[ 8, 9, 10]。序列分析表明TCP家族转录因子具有典型的basic Helix- Loop-Helix (bHLH)功能域[ 11]。同时含有bHLH结构域的SPATULA(SPT)在拟南芥叶片中超表达会减小叶片尺寸[ 12], 含有非典型性bHLH结构域转录因子PAC-L-OBTRAZOL RESISTANCE1 (PRE1)和INCREASED LEAF INCLINATION1 (ILI1)同一些bHLH类转录因子结合, 负调控叶片细胞的生长, 引起植株矮小、叶片边缘卷曲等性状[ 13, 14]。上述结果表明含有bHLH结构域的转录因子在叶片发育和卷曲过程中起着重要的调控作用。
本试验通过结球甘蓝519莲座期、结球期茎尖和叶片的转录组对比, 筛选出差异表达且含有bHLH结构域的 BoL H01和 BoLH02基因, 经PCR扩增和测序验证后对其序列进行了生物信息学分析, 利用荧光定量PCR技术分析 BoLH01和 BoLH02以及上游含有bHLH识别序列的 BoHB7、 BoHB12、 BoILL6基因的表达情况, 以期为进一步鉴定 BoLH01和 BoLH02的生物学功能提供依据。
将结球性好、扁圆形的中晚熟结球甘蓝纯合系519, 播种于西南大学十字花科研究所歇马试验基地。2012年9月至12月分别于叶片长度(从叶尖到叶片基部)大于1 cm叶数为21片叶的莲座期, 从每株取叶片长度(从叶尖到叶片基部)为1 cm的2个叶片和长度为3 mm的茎尖(带3~5片未展开的叶片), 共30株; 于叶片长度(从叶尖到叶片基部)大于1 cm叶数为50片的结球期, 从每株取叶片长度(从叶尖到叶片基部)为1 cm的2个叶片和长度为3 mm的茎尖(带3~5片未展开的叶片), 共30株。用于总RNA提取和转录组测序。2012年12月至2013年4月分别于叶片长度(从叶尖到叶片基部)大于1 cm叶数为24片的莲座期, 从每株取叶片长度(从叶尖到叶片基部)为1 cm的2个叶片和长度为3 mm的茎尖(带3~5片未展开的叶片), 共30株; 于叶片长度(从叶尖到叶片基部)大于1 cm叶数为40片叶的中间期(莲座到结球的过渡期), 从每株取叶片长度(从叶尖到叶片基部)为1 cm的2个叶片和长度为3 mm的茎尖(带3~5片未展开的叶片), 共30株; 于叶片长度(从叶尖到叶片基部)大于1 cm叶数为60片的结球期, 从每株取叶片长度(从叶尖到叶片基部)为1 cm的2个叶片和长度为3 mm的茎尖(带3~5片未展开的叶片), 共30株, 用于总RNA的提取和基因表达分析。
植物总RNA提取试剂盒RNAprep Pure Plant Kit DP432购自天根生化科技(北京)有限公司。荧光定量PCR染料SYBR Premix Ex Taq II (Tli RNaseH Plus)购自TaKaRa。大肠杆菌感受态T1、克隆载体pEASY- Blunt等购自TransGen公司。高保真DNA聚合酶Prime STAR、 Taq DNA聚合酶、反转录试剂盒Primescript RT Reagent Kit RR047A购自TaKaRa宝生物工程有限公司(中国大连)。胶回收试剂盒、质粒提取试剂盒购自Bio-Flux公司。引物合成和测序由华大基因完成。
将莲座期和结球期的1 cm叶片随机分成3组, 每组3个叶片, 按照以下方法测量长度, 并求平均值, 计算卷曲指数。叶片下卷时, 侧卷指数 TC = 1 - lm/ pw, lm为叶片自然状态下的叶宽, pw为叶片压平后的叶宽; 纵卷指数 LC = 1 - tb/ pl, tb为叶片自然状态下的叶长, pl为叶片压平后的叶长; 叶片上卷时, 侧卷指数TC = lm/ pw-1, lm为叶片自然状态下的叶宽, pw为叶片压平后的叶宽; 纵卷指数 LC= tb/ pl - 1, tb为叶片自然状态下的叶长, pl为叶片压平后的叶长。
参照RNAPrep Pure植物总RNA试剂盒DP432 (TIANGEN, 北京)提取总RNA, 将RNA溶液稀释至相同浓度后, 参照Primescript RT reagent Kit RR047A (TaKaRa,大连)操作说明反转录合成cDNA, 经NanoVue plus超微量分光光度计(GE, 美国)测定后将反转录产物稀释至同一浓度, -20℃保存备用。
分别将21片叶莲座期和50片叶结球期茎尖和叶片混合提取总RNA后, 送北京百迈克公司用HiSeq 2000进行转录组测序, 利用Trinity软件对各样品数据进行Unigenes组装, 通过RPKM值来反映Uigenes表达丰度, 使用IDEG6软件进行差异表达基因的检测, 以log2(不同时期RPKM比值)绝对值大于等于1为标准统计差异表达基因, 通过Blast软件将差异表达Unigenes序列与NR、NT、SwissProt、TrEMBL、GO、COG和KEGG数据库比对, 获得Unigenes注释信息, 筛选差异表达的含有bHLH结构域的基因。
根据转录组分析获得的cDNA序列结合芸薹属数据库检索(http://brassicadb.org/brad/index.php), 运用Primer primer 6.0软件设计扩增基因引物, 序列见表1, 以50片叶结球期的叶片cDNA为模板, 基因克隆PCR体系为25 μL, 按照高保真DNA聚合酶PrimeSTAR说明书操作。扩增参数为98℃预变性2 min; 98℃ 10 s; Tm: 15 s (表1); 72℃ 30 s; 35个循环; 72℃ 延伸10 min。PCR扩增产物经1.2% 琼脂糖凝胶电泳检测, 回收纯化目的条带, 连接克隆载体pEASY- Blunt, 转化Trans1-T1大肠杆菌感受态细胞, 通过蓝白斑筛选挑取白色阳性菌落, 经PCR验证阳性克隆子后送华大基因公司测序。
| 表1 本研究所用引物 Table 1 Primers used in this study |
用ClustalX l.8.0软件比对分析获得的 BoLH01和 BoLH02基因CDS序列, 用DNAStar软件推导基因编码的氨基酸序列, 通过ExPASy (http://expasy.org/tools/)在线分析氨基酸的理化性质, 用Swiss-model (http://swissmodel.expasy.org/)、PROSITE (http://www.expasy.org/prosite)、InterProScan (http://www.ebi.ac.uk/ interpro/scan.html)和Predict Protein (http://www.predictprotein.org/)预测蛋白质的高级结构和功能位点, 利用NCBI中BlastN (http://www.ncbi.nlm.nih.gov/)在线分析甘蓝 BoLH01和 BoLH02基因和其他物种的同源性, 将搜索的同源序列bHLH结构域及已知卷叶有关的AtTCP转录因子的bHLH结构域用ClustalX l.8.0软件进行多序列联配比对分析, 通过MEGA 5.1软件构建分子进化树分析其进化关系。
依据获取 BoLH01和 BoLH02基因的cDNA序列以及芸薹属数据库(http://brassicadb.org/brad/)比对结果设计用于基因荧光定量PCR检测的引物(表1)。分别以莲座期、中间期和结球期的茎尖、叶片的cDNA为模板, 参照TaKaRa SYBR Premix Ex Taq II试剂盒说明书检测 BoLH01、 BoLH02基因在各组织中表达情况, 以 TIPS和 Actin2作为内参基因[ 15], 反应在C1000/CFX96仪器(Bio-Rad, 美国)上完成。荧光定量PCR体系25 μL, 含2× SYBR Premix Ex Taq 12.5 μL, 引物各1 μL, cDNA 模板2 μL, 超纯水8.5 μL, 混合加样。反应程序为95℃预变性30 s; 95℃ 5 s, Tm值30 s (表1), 40个循环。利用Bio-Rad CFX96荧光定量PCR仪CFX Manager Software软件(2-ΔΔCT法)和Microsoft Excel软件分析数据。
依据文献检索、序列比对和氨基酸功能域分析从转录组差异表达基因中筛选拟南芥叶片卷曲相关基因的同源基因。利用转录组获取的ATG上游序列结合芸薹属数据库(http://brassicadb.org/brad/index.php)比对, 鉴定出拟南芥 AtHB7、AtHB12和 At ILL6的甘蓝同源基因 Bo HB7、 Bo HB12、 Bo ILL6, 3个甘蓝基因ATG上游存在能被bLHLH结构域识别的E-box序列。 Bo HB7、 Bo HB12和 Bo ILL6基因在莲座期、中间期和结球期茎尖、叶片中表达的荧光定量PCR检测见1.2.6, 荧光定量产物经测序鉴定。
为分离甘蓝叶片卷曲相关的调控基因, 我们以结球甘蓝519莲座期和结球期茎尖与叶片为材料, 进行转录组分析, 经Trinity软件对各样品转录组测序数据进行组装, 共得到54 209条Unigenes, 利用RPKM值来反映Uigenes表达丰度, 以log2(不同时期RPKM比值)绝对值大于等于1为标准统计差异表达基因, 其中莲座期茎尖与叶片间差异表达的基因达2923条; 结球期茎尖与叶片间差异表达基因达2307条; 莲座期与结球期茎尖间差异表达的基因达652条; 莲座期与结球期的叶片间差异表达的基因为1786条。
莲座期和结球期茎尖间差异表达基因与叶片间差异表达基因之间的重叠基因为488条, 通过Blast软件将上述Unigenes序列与NR、NT、SwissProt、TrEMBL、GO、COG和KEGG数据库比对, 获得Unigenes注释信息, 发现其中差异表达的bHLH类转录因子分别为 BoLH01、 BoLH02和 BoAIB, 其中 BoLH01基因log2(结球期茎尖RPKM/莲座期茎尖RPKM)为2.58, log2(结球期叶片RPKM/莲座期叶片RPKM)为15.60; BoLH02基因log2(结球期茎尖RPKM/莲座期茎尖RPKM)为4.39, log2(结球期叶片RPKM/莲座期叶片RPKM)为13.87; BoAIB基因log2(结球期茎尖RPKM /莲座期茎尖RPKM)为2.16, log2(结球期叶片RPKM/莲座期叶片RPKM)为3.76。依据在2个时期的茎尖间和叶片间均差异表达, 且差异显著性由高到低的原则, 我们从中选取了 BoLH01和 BoLH02进行后续分析。叶片卷曲指数分析表明莲座期叶片纵卷指数为-0.06, 侧卷指数为-0.19; 结球期叶片的纵卷指数为-0.28, 侧卷指数为-0.29。 BoLH01和 BoLH02基因在卷曲程度更高的结球期叶片中表达量比莲座期叶片中高, PCR扩增测序结果表明获取的基因编码序列与转录组测序结果完全一致。
经序列分析表明 BoLH01基因CDS全长为966 bp、分子量为35964 Da、编码321个氨基酸残基, GC含量40.58%, 通过Predict在线分析表明BoLH01是一种跨膜蛋白, 包括跨膜域(281~298)、膜内域(1~280)和膜外域(299~321); 该蛋白包含1个N糖基化位点(283~286)、7个蛋白激酶C磷酸化位点(9~11、92~94、116~118、131~133、159~161、194~196、250~252)、7个酪蛋白II磷酸化位点(14~17、41~44、54~57、194~ 197、229~232、250~253、303~306)、3个N-豆蔻酰化位点(130~135、233~238、278~283)和1个异戊二烯基结合位点(318~321), 核定位信号为ERKRRE、RKRREK (151、152); 经PROSITE在线分析该蛋白具有典型的bHLH功能域(142~191)(图1)。
序列分析表明BoLH02基因CDS全长870 bp, 分子量为32 963 Da, 编码289个氨基酸残基, GC含量39.66%; 通过Predict在线分析表明BoLH02是一种跨膜蛋白, 包括跨膜域(249~266)、膜内域(1~248)和膜外域(267~289); 该蛋白包含1个N糖基化位点(75~ 78)、1个cAMP和cGMP依赖性磷酸化位点(分别为92~95、96~99), 4个蛋白激酶C磷酸化位点(36~38、95~97、102~104、125~127)、7个酪蛋白II磷酸化位点(11~14、28~31、145~148、197~200、216~219、258~ 261、275~278)和2个N-豆蔻酰化位点(137~142、190~ 195), 核定位信号为RRGSKRK, ERKRRE, RKRREK (92、117、118); 经PROSITE在线分析该蛋白也具有典型的bHLH功能域(108~157)(图1)。
通过NCBI数据库在线Blast比对表明BoLH01与AtbHLH18的核苷酸序列和推导氨基酸的一致性都最高, 分别为86%和81%, 与拟南芥AtbHLH25次之; BoLH02与AtbHLH19的核苷酸序列和推导氨基酸的一致性都最高, 分别为83%和74%。说明BoLH01和BoLH02可能是甘蓝2个bHLH类转录因子基因, 通过NCBI数据库在线收集13条与叶片发育和卷曲相关的bHLH类转录因子, 以及2条与叶片发育和卷曲相关的非特异性bHLH类转录因子, 利用MEGA5.1软件构建氨基酸序列NJ系统进化树(图2), 从图2可以看出BoLH01和BoLH02与拟南芥AtbHLH18、AtbHLH19、AtbHLH20、AtbHLH25、AtPRE1和水稻OsILI1转录因子处于同一个大的分支, 其中BoLH01与AtbHLH18、BoLH02与AtbHLH19亲缘关系最近, 与拟南芥AtPRE1和水稻OsILI1亲缘关系次之; 拟南芥AtTCP2、AtTCP3、AtTCP4、AtTCP10和AtTCP24处于同一个分支, 该分支内TCP与BoLH01和BoLH02所在分支的成员亲缘关系较近, AtTCP9、AtTCP11、AtTCP14和AtTCP15与BoLH01和BoLH02所在分支的成员亲缘关系最远。
利用ClustalX 1.8.0软件对BoLH01和BoLH02在内的12条蛋白bHLH结构域进行多序列联配比对, 结果(图3)显示甘蓝BoLH01和BoLH02均具有典型bHLH功能域, 包含DNA结合域(basic)和负责形成异源或同源二聚体的螺旋-环-螺旋结构域(helix- loop-helix)[ 16, 17]。Atchly等[ 17]和Toledo-Ortiz等[ 18]指出DNA结合域内保守的13E-16R是bHLH功能域特异识别结合下游基因启动子序列中E-box(5°- CANNTG-3°, 其中N代表任意核苷酸)的2个关键位点, 9H-13E-17R是bHLH功能域特异结合下游基因启动子序列中G-box (5°-CACGTG-3°)的3个关键位点, 甘蓝BoLH01中151E-154R和BoLH02中117E-120R位点与13E-16R对应, 甘蓝BoLH01中147H-151E-155R和BoLH02中113H-117E-121R位点与9H-13E-17R对应。Heim等[ 19]指出HLH结构域中保守的Leu23是bHLH形成二聚体的关键位点, BoLH01中Leu165和BoLH02中的Leu131与Heim提及的Leu23相对应。由图3可以看出不同的bHLH功能域存在氨基酸的缺失和插入现象, 上述的关键氨基酸位点在BolH01、BoLH02、AtSPT、AtbHLH18、AtbHLH20、AtbHLH25以及AtTCP15中均高度保守。在具有非典型性bHLH结构域AtPRE1和OsILI1中仅具有保守的与Heim[ 19]提及的Leu23相对应位点, 在DNA结合域均没有13E-16R和9H-13E-17R对应的保守位点。利用Swiss-model在线服务器构建BoLH01的bHLH功能域空间结构, 结果显示生成的三维结构包含150~193氨基酸, 由两个螺旋和一个折叠组成, 其与DNA序列识别关键氨基酸Glu151和Arg154以及bHLH二聚体形成的关键氨基酸位点Leu165处于第一个螺旋。
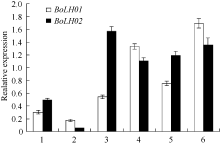
利用实时荧光定量PCR技术, 以 TIPS和 Actin2基因为内参, 分别检测甘蓝3个时期的茎尖和叶片中 BoLH01、BoLH02基因表达情况。从图4可以看出, BoLH01基因在各时期茎尖和叶片中均有表达, 表达量在莲座期、中间期和结球期3个时期茎尖和叶片中均呈逐渐上升的趋势, 其中结球期叶片表达量是中间期叶片的1.27倍, 是莲座期叶片9.81倍; 结球期茎尖表达量是中间期茎尖的1.38倍, 是莲座期茎尖的2.47倍, 莲座期的茎尖 BoLH01基因的表达量高于莲座期叶片表达量, 而中间期和结球期均以叶片的表达量高于茎尖表达量。从图4可以看出, BoLH02基因在各时期茎尖和叶片中也都有表达, 3个时期的叶片中 BoLH02的表达量呈逐渐上升的趋势, 结球期叶片的表达量是中间期的1.22倍, 是莲座期的24.54倍, 3个时期茎尖内的表达量呈先升后降的趋势, 中间期茎尖的表达量是莲座期的3.17倍, 结球期茎尖表达量是莲座期的2.40倍, 在莲座期和中间期叶片内 BoL H02基因的表达量均低于茎尖的表达量, 而结球期叶片的表达量高于茎尖的表达量。上述结果表明 BoLH01和 BoLH02基因在结球期茎尖和叶片的表达量均显著高于莲座期茎尖和叶片的表达量, 这与前述转录组分析的2个基因表达量的变化趋势完全一致。
序列分析表明甘蓝BoLH01和BoLH02蛋白具有典型的bHLH功能域, 能特异性识别目的基因ATG上游序列中E-box序列。通过文献检索、序列比对和氨基酸功能域分析, 首先从转录组差异表达基因中筛选拟南芥卷叶相关基因同源基因, 再结合转录组获取的甘蓝基因ATG上游序列在芸薹属数据库比对, 筛选出拟南芥卷叶相关基因 AtHB7、AtHB12和 ILL6的甘蓝同源基因 BoHB7、BoHB12和 BoILL6, BoHB7基因ATG上游序列274 bp处、 BoHB12基因ATG上游序列431 bp处和 BoILL6基因ATG上游序列137 bp处分别具有能被bHLH功能域识别的E-box序列(CAGATG)。利用荧光定量PCR分析上述3个基因在甘蓝3个时期6种材料的表达情况, 结果见图5, 表明3个基因均在结球期叶片中高量表达, 其中结球期叶片 BoHB7表达量是中间期的1.12倍, 是莲座期的6.25倍; 结球期叶片 BoHB12表达量是中间期的3.85倍, 是莲座期的38.14倍; 结球期叶片 BoILL6表达量是中间期的2.53倍, 是莲座期的78.14倍, 上述3个基因在茎尖的表达量变化不显著, 且远低于结球期叶片中的表达量。
叶片卷曲对于结球甘蓝来说是一种非常有用的农艺形状, 结球期叶片向上向内卷曲抱合形成的叶球是可食用器官, 同时为研究叶片卷曲提供了理想的材料。因此了解结球甘蓝叶片极性或叶片卷曲发生的遗传规律, 揭示该过程中基因调控的分子机制, 将为研究和利用基因工程调控叶片的形态, 发展叶球发育的理论以及培育高产优质的结球甘蓝新品种提供依据。本研究通过结球甘蓝莲座期和结球期茎尖、叶片的转录组对比筛选出 BoLH01和 BoLH02两个差异表达基因, 经PCR扩增和测序鉴定后对BoLH01和BoLH2的核苷酸和氨基酸序列Blast比对表明它们分别与拟南芥AtbHLH18和AtbHLH19同源相似性最高, BoLH01和BoLH02均含有典型的bHLH结构域, 推测两者可能是甘蓝bHLH转录因子家族成员。典型的bHLH结构域是由N端约15个氨基酸组成能特异性结合不同形式的E-box(CANNTG)序列的DNA结合区[ 16]和C端由螺旋-环-螺旋组成的负责bHLH同源或异源二聚体的形成的结构域两部分组成[ 17]。对BoLH01和BoLH02的bHLH结构域分析表明两者均具保守的13E-16R和9H-13E-17R以及Leu23对应的氨基酸位点, 两者具有典型的bHLH功能域活性, 既可以识别结合E-box序列也可以特异性识别结合G-box序列, 同时也可以与其他的bHLH转录因子形成异源或者同源二聚体发挥下游基因表达的调控作用[ 20, 21]。
研究表明TCP4[ 5]、TCP11[ 10]、TCP15[ 8]、TCP20[ 4]、SPT[ 12]等bHLH类转录因子和非特异性bHLH结构域转录因子PRE1/ILI1[ 13, 14]在植物叶片形态建成和发育过程中有着重要功能。通过bHLH结构域多序列联配比对表明BoLH01、BoLH02、AtSPT以及AtTCP15一致, 均具保守的13E-16R和9H-13E-17R以及Leu23对应的氨基酸位点, 结合转录组和荧光定量PCR分析结果均显示 BoLH01和 BoLH02两个基因在结球期叶片中高量表达, 表达量的变化与甘蓝叶片卷曲趋势一致, 由此推测 BoLH01和 BoLH02可能具有类似于 CIN-TCPs[ 22, 23]转录因子功能, 在甘蓝叶片卷曲过程中发挥重要的调控作用。已有研究表明TCP14和TCP15在调节拟南芥节间延伸的功能上具有冗余性[ 8], Danisman等研究证明TCP9和TCP20双突变体在调节叶片发育过程中具有功能冗余性[ 24]。BoLH01和BoLH02序列分析表明两者均具典型的bHLH功能域, 且转录组和荧光定量PCR分析表明 BoLH01和 BoLH02基因在莲座期、中间期、结球期叶片中均呈高度相似的上升变化趋势, 由此我们推测BoLH01和BoLH02在功能上可能存在冗余性。
BoLH01和BoLH02具有典型的bHLH结构域和保守的关键氨基酸位点, 能特异识别下游含E-box和G-box序列的信号元件。Park等发现拟南芥 ATHB7、 ATHB12在受病毒感染植株的不正常卷曲发育叶片中高水平表达[ 25], Widemann等研究表明拟南芥 At ILL6能促进IAA水平的升高[ 26], 进而诱导叶片偏上性生长[ 27]。拟南芥 AtHB7、AtHB12、AtILL6的甘蓝同源基因 BoHB7、BoHB12和 BoILL6的上游均含能被bHLH功能域识别的E-box序列。对 BoHB7、BoHB12、BoILL6在莲座期、中间期和结球期荧光定量分析显示3个基因均在甘蓝卷曲的球叶中高量表达。三者表达量的变化与 BoLH01和 BoLH02表达量的变化趋势完全一致。基于上述结果, 我们推测BoLH01和BoLH02可能通过正向调控 BoHB7、BoHB12、BoILL6基因的表达来参与甘蓝叶片卷曲调控。上述分析结果为进一步利用转基因等技术鉴定BoLH01和BoLH02在甘蓝叶片卷曲过程中的生物学功能提供了参考。
从甘蓝转录组差异表达基因中筛选出 BoLH01和BoLH02基因。BoLH01和BoLH02具有典型bHLH结构域和完全保守的13E-16R和9H-13E-17R以及Leu23对应的关键氨基酸位点, 甘蓝 BoLH01和 BoLH02基因表达量的变化与甘蓝叶片卷曲趋势一致, 在莲座期平展的叶中均低水平表达, 结球期卷曲的球叶中表达水平显著升高, 说明这2个基因可能在甘蓝卷曲调控过程中发挥作用。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|