MYB类转录因子是植物转录因子最大的家族之一, 参与控制植物腺毛细胞的模式和形态建成。本研究利用雷蒙德氏棉(
MYB transcription factor, one of the most important protein families in plants, is involved in the regulation of secondary metabolism, morphogenesis of plant, responding to environment stress and plant hormone. In this study, we used D5 genomic bank of
棉花纤维是最大的可再生纺织原料, 同时也是人工纤维的潜在替代品。棉花纤维细胞是一种不分支的单细胞, 由胚珠表皮细胞分化、发育而形成, 是高等植物中伸长最快、发育最长的单细胞, 也是现在研究单细胞的最好的生物模式[ 1, 2]。所有的表皮细胞(除气孔保卫细胞和珠孔细胞外)都有可能发育成纤维原始细胞, 但并非所有的都能分化成纤维。研究证明约30%~40%的胚珠表皮细胞分化形成纤维原始细胞, 但这些原始细胞只有l/3能形成有效纤维[ 3]。
MYB类转录因子是植物转录因子最大的家族之一, 也是功能最多样化的家族, 现已证实其参与多种植物代谢途径。植物中第一个被鉴定分离出的MYB基因是1987年Paz-Ares等[ 4]发现的与玉米色素合成有关的 Clorless1基因 c-myb。此后在许多生物中也相继鉴定出与c-MYB蛋白同源的蛋白。研究结果显示拟南芥公布的1922个转录因子中, MYB类转录因子约168个, 占整个拟南芥转录因子总数的8.74%。玉米的MYB转录因子数目也已经超过80个, 说明MYB基因是由多个成员组成的大家族(植物转录因子数据库http://planttfdb.cbi.pku.edu.cn/ index.php)。以c-MYB为标准, 将MYB保守结构域分为3类(R1、R2、R3)[ 5], 据MYB保守结构域重复种类和数目的不同, 将整个MYB转录因子家族分为4类, 其中R2R3-MYB是植物MYB转录因子中最大的一类[ 6]。目前R2R3-MYB类转录因子研究的热点主要集中在腺毛发育调节方面, 例如参与控制腺毛细胞模式和形态建成, 其中研究最为广泛的2种模型即拟南芥表皮毛的激活侧抑制模型和棉花纤维起始的机制[ 7]。
棉花纤维与拟南芥表皮毛两者都是单细胞, 都起源于表皮, 两者很可能有类似的发育机制[ 8, 9, 10, 11, 12]。因此目前在棉花中克隆的纤维特异表达MYB基因大都与拟南芥表皮毛发育相关基因 AtMYB0 ( GL1, NM_113708)同源, 这些MYB转录因子的功能主要集中在调控棉花纤维的起始和伸长2个阶段。AtMYB0是一类R2R3-MYB转录因子, 其过表达会抑制拟南芥表皮毛细胞的分化与发育, 功能缺失突变体为无毛表型[ 13]。而最早在棉花中分离到的R2R3- MYB基因是 GhMYB1-GhMYB6, Loguercio等[ 14]根据其表达特征指出MYB基因对棉纤维分化起主要作用。Guan等[ 15]研究证明 GhMYB2能使拟南芥产生种皮毛和果夹异位表皮毛, 且与一些负调控因子有协同作用。在陆地棉中分离出另外1个MYB转录因子GhMYB25, 与金鱼草中的R2R3-MYB型转录因子AmMIXTA 相似, 该基因在长纤维细胞起始时期高调表达[ 16]。研究发现在烟草中过量表达 GhMYB25能增加叶表皮毛分支, 进一步表明棉纤维和表皮毛发育有关联, 通过激光捕获显微解剖试验表明相对于无纤维胚珠表皮细胞, GhMYB25在有纤维起始细胞中富集表达[ 17, 18]; 另外研究发现棉花 GhMYB109基因与 AtMYB0同源性最高[ 13], 其在纤维起始细胞和延伸细胞中特异表达, GL1::GhMYB 109能部分互补拟南芥 gl-1突变体表型[ 19]。
综上所述, R2R3-MYB转录因子在决定棉花胚珠表皮细胞命运、促使纤维原始细胞分化以及在纤维伸长过程中都发挥着作用。因此本研究利用雷蒙德氏棉( Gossypium raimondii) D5基因组数据库以拟南芥R2R3-MYB转录因子AtMYB0 (GL1, NM_ 113708)蛋白为参比序列, 同源克隆了一个新的棉花R2R3-MYB基因 GhMYB0。在表达模式分析的基础上对该R2R3-MYB转录因子遗传转化拟南芥, 对其功能进行了验证, 为更进一步解析R2R3-MYB转录因子在决定胚珠表皮细胞命运的调控机制以及在棉花纤维伸长中的作用提供理论依据, 为棉花分子改良定向育种奠定基础。
陆地棉徐州142及其无绒无絮突变体, 种植于中国农业科学院棉花研究所南试验场, 田间常规管理, 棉花生长期间, 分别采集野生型的根、茎、叶、花及野生型10个发育时期(-3、-1、0、1、3、5、10、15、20和25 DPA)的胚珠和纤维。将所有田间材料立即放入液氮速冻, 于-80℃冰箱保存备用。野生型拟南芥(Columbia)种植于本课题拟南芥培养室。所用数据库为雷蒙德氏棉D5基因组数据库(http://cgp.genomics.org.cn/page/species/index.jsp)。
采用CTAB法提取棉花叶片的基因组DNA[ 20]。利用TIANGEN公司RNA提取试剂盒参照说明书提取不同组织和各发育阶段棉花胚珠及纤维的总RNA。按TaKaRa (宝生物工程有限公司)反转录试剂盒说明书合成cDNA第1链, -20℃保存备用。
电子克隆以拟南芥( Arabidopsis thaliana, NM_ 113708)的AtMYB0蛋白序列为参比序列在雷蒙德氏棉( Gossypium raimondii) D5基因组中比对调取与其相似的序列, 在徐州142野生型中进行 GhMYB0全长cDNA的克隆。采用Oligo6软件设计引物GhMYB0F和GhMYB0R (表1)。
以 GhMYB0为查询参比序列, 在NCBI的GenBank数据库中搜索与该基因同源的其他物种的氨基酸序列, 通过ClustalX2软件进行氨基酸序列比对, 进行保守结构域分析, 采用MEGA5.0软件的
Neighbor-Joining算法构建系统进化树, 用BootStrap检验, 并重复1000次系统进化分子; 用ProtParam (http://web.expasy.org/protparam/)进行氨基酸基本理化性质分析; 利用Predictprotein (http://www.pre-dictprotein.org/)和SignalP (http://www.cbs.dtu.dk/services/SignalP/)分别预测跨膜区、功能位点和信号肽; 由ProtScale tool (http://expasy.org/tools/pscale/ Hphob.Woods.html)预测蛋白质的疏水性。
构建35s::GhMYB:GFP融合表达载体, 引物见表1。取一块长、宽大约4 cm的洋葱幼嫩表皮, 在固体MS培养基上22℃培养3~4 h。采用PDS-1000/ He基因枪(Bio-Rad)轰击洋葱表皮细胞, 可裂膜压力为1100 psi, 轰击距离约为9 cm。于22℃培养箱中培养被轰击的洋葱表皮细胞14~16 h, 于激光共聚焦显微镜下(Leica, TCS SP2)观察GFP的表达定位。
| 表1 GhMYB0基因克隆与表达所用引物 Table 1 Sequences of primers for GhMYB0isolation and expression |
利用在线工具In-Fusion Primer Design Tool (http://bioinfo.clontech.com/infusion/convertPcrPrimers-Init.do)设计In-GhMYB0上下游引物(表1), 构建超表达载体。将扩增得到的片段利用凝胶电泳回收后, 采用In-Fusion HD Cloning System试剂盒(购自Clontech公司)与经 BamH I单酶切后线性化的pBI121植物表达载体(由中国农业科学院棉花所早熟课题组馈赠)相连接, 获得超表达载体。将超表达载体进行大肠杆菌(DH5α)转化, 由上海生工生物工程技术服务有限公司测序。然后进行农杆菌转化(由中国农业科学院棉花所转基因课题组馈赠), 将得到的阳性菌液采用蘸花法侵染拟南芥野生型植株和 gl-1突变体[ 21], 收获成熟T0代种子。T0代种子经消毒后, 点播在MS选择培养板上(1‰卡那霉素)。4℃下春化3 d后, 移入光照培养箱(22℃恒温, 24 h光照, 光强30~40 µmol m-2 s-1)。10 d后挑选深绿色健康真叶和根伸长至培养基中的转化体。将转化体幼苗移入土中(营养土∶蛭石=1∶1, v/v), 至收获T1代种子。经卡方检测, 筛选出符合3∶1分离比例的T1代种子, 种植后获得T2代纯系种子。同时, 提取3个转基因株系和野生型整株RNA后进行反转录, 用qRT-PCR方法检测目的基因在转基因株系中是否正常表达(PCR SYBR Green Real-time PCR Master Mix购自康为世纪), 检测上下游引物GhMYB0-q (表1)。利用SteREO Discovery.V8体视显微镜观察转基因拟南芥叶片及花器官。
分别提取徐州142的根、茎、叶、胚珠(0 DPA和5 DPA)、以及野生型和无绒无絮突变体不同发育时期(-3、-1、0、1、3、5、10、15、20和25 DPA)纤维的总RNA, 反转录成cDNA, 稀释8倍取2 μL用于qRT-PCR分析。以基因 Ghhiston3为内参, 比较不同组织器官不同发育期相对表达水平。反应在iCycler iQ5荧光定量PCR仪(Bio-Rad, USA)上进行, 采用SYBR Premix Ex Taq II试剂盒。反应体系为20 μL, 包括SYBR Premix Ex Taq II (2×) 10 μL、 10 μmol L-1GhMYB0-q上下游引物各0.4 μL、模板cDNA 2 μL, 加ddH2O至20 μL。PCR程序为95℃预变性10 min; 95℃变性15 s, 58℃退火30 s, 72℃延伸20 s, 40个循环; 最后72℃延伸5 min。设3个技术重复。荧光定量引物见表1。
采用电子克隆方法获得含有完整ORF的雷蒙德氏棉 GrMYB0基因, 利用特异引物GhMYB0F和GhMYB0R (表1)在陆地棉徐州142 cDNA中扩增, 产物ORF长度为843 bp, 编码280个氨基酸(图1)将该基因命名为 GhMYB0。通过氨基酸序列分析发现, 与雷蒙德氏棉D5基因组调取的 GrMYB0相比, 保守结构域之外, 在陆地棉中克隆的 GhMYB0多9个氨基酸, 并存在7个氨基酸的差异(图2-A)。保守结构域分析发现, GhMYB0基因编码的蛋白具有典型的MYB转录因子R2和R3结构域(图2-B)。系统进化树分析发现, GhMYB0与棉花GhMYB4以及大豆GmMYB185归为一个小的类群(图3-A)。基因结构分析结果显示, GhMYB0编码区内仅含有一个内含子, 长度为86 bp (图3-B)。
 | 图1 棉花 GhMYB0全长核苷酸序列及推测编码的氨基酸序列方框代表起始密码子; *代表终止密码子。Rectangular frame: initiation codon; * termination codon.Fig. 1 Full-length nucleotide sequence and putative amino acid sequence of GhMYB0 |
 | 图3 GhMYB0 系统进化树和基因组内含子位置图中“●”代表本研究中的 GhMYB0。“●” stands for GhMYB0.Fig. 3 Phytogenic tree of GhMYB0 and intron site of GhMYB0 |
利用ProtParam分析显示, GhMYB0的理论分子量为32.269 kD, 理论等电点为9.22, 平均亲水系数(grand average of hydropathicity, GRAVY)为-0.974。通过蛋白质亚细胞定位PSORT工具预测, GhMYB0蛋白被定位于细胞核, 用TargetP、SingleP、PredictNLS等进行定位信号分析, 综合结果表明 GhMYB0蛋白是核蛋白, 不具有信号肽或者定位信号。PROSITE分析结果显示GhMYB0含有MYB型的螺旋-转角-螺旋DNA结合域(Myb-type HTH DNA-binding domain profile)。
对GhMYB0基因亚细胞定位融合载体用基因枪轰击法转入洋葱表皮细胞, 用pBI121表达载体作为对照。经过培养后, 在激光共聚焦显微镜下观察, 显示对照GFP蛋白可以在细胞膜和细胞核内见到比较均一的明亮的绿色荧光(图4-A, B, C), 而pBI121-GhMYB0融合蛋白则位于细胞核中(图4-D, E, F), 更进一步说明GhMYB0蛋白是核定位的转录因子。
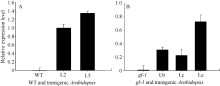
qRT-PCR分析表明, GhMYB0在整个发育过程中表达趋势基本一致, 并且表达量在不同时期均以徐州142野生型(WT)明显高于其突变体( fl)。 GhMYB0在徐州142开花当天(0 DPA)开始高调表达, 开花后20 DPA达到最大, 初步推测该基因可能在棉花纤维的起始和伸长阶段皆发挥一定的作用(图5-A), 该基因在花中表达最高, 在胚珠中其次(图5-B)。
 | 图5 GhMYB0表达的qRT-PCR分析DPA: 开花后天数; WT: 野生型徐州142; fl: 徐州142无绒无絮突变体。-3、-1、0、1、3、5分别代表-3、-1、0、1、3、5 DPA的胚珠和纤维组织; 10、15、20、25分别代表10、15、20、25 DPA纤维组织。Fig. 5 qRT-PCR analysis of GhMYB0 expressionDPA: days post anthesis; WT: wild type of Xuzhou 142; fl: the mutant of Xuzhou 142. -3, -1, 0, 1, 3, 5 indicate ovule and fibers at -3, -1, 0, 1, 3, 5 DPA respectively. 10, 15, 20, 25 indicate fibers at 10, 15, 20, 25 DPA respectively. |
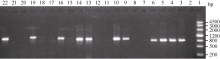
利用抗性筛选和PCR筛选, 共筛选出10个抗性转基因拟南芥株系, PCR结果显示10个转基因株系全部扩增出了与目的基因片段大小(843 bp)一致的特异性条带, 阴性对照(野生型拟南芥叶片)中没有扩增出此特异条带(图6); 分别提取T3代野生型背景下2个转基因拟南芥株系(L2和L5)以及 gl-1突变体背景下3个转基因株系(Lb、Lc和Le)的总RNA, 反转录后进行qRT-PCR分析, 选拟南芥内参基因 AtTUB2作为平行反应, 引物GhMYB0-qF、GhMYB0-qR和AtTUB-qF、AtTUB-qR见表1。结果显示 GhMYB0在野生型拟南芥和 gl-1突变体中几乎不表达, 而在转基因株系中均有表达(图7)。综上说明, GhMYB0在这些转基因株系中正常表达, 并没有发生基因沉默。
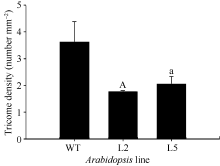
为分析 GhMYB0转录因子基因的功能, 构建了35S::GhMYB0的拟南芥超表达载体, 并获得10个超表达后代纯系。与野生型相比, GhMYB0超表达拟南芥植株的表皮毛减少(图8)。利用SteREO Discovery.V8体视显微镜分别观察2个转基因株系(L2和L5)第3片真叶表皮毛, 每个株系3个重复, 统计分析表明, 超表达株系L2和L5皮毛密度分别为1.77个 mm-2和3.26个 mm-2, 分别比野生型低51.24%和10.19% (图9), 说明超表达株系中表皮毛密度降低。另外 GhMYB0能够恢复拟南芥表皮毛缺失突变体 gl-1的表型(图8)。可以看出过表达 GhMYB0对拟南芥表皮毛的生成有抑制作用, 与前人对 GL1和 GaMYB23( GL1同源基因)的研究结果相符。继续对T3代35S::GhMYB0转基因拟南芥观察, 发现有一株系与野生型相比出现严重矮化的现象, 雄蕊明显短于野生型, 不能形成正常的果夹, 萼片上表皮毛数量与野生型相比明显增多(图10)。
 | 图8 GhMYB0转基因株系和野生型叶片表皮毛观察WT: 野生型拟南芥; gl-1: 拟南芥表皮毛缺失突变体。Fig. 8 Trichome of 35S::GhMYB0 transgenic lines and WTWT: Columbia; gl-1: the mutant of Arabidopsis. |
MYB转录因子是研究较为广泛的植物转录因子。在许多植物中克隆了MYB基因, 并对其功能进行了深入研究。近年来, 研究证明植物中MYB转录因子参与控制植物腺毛细胞的模式和形态建成。棉花纤维与拟南芥表皮毛两者都是单细胞, 均起源于表皮, 两者很可能有类似的发育机制[ 17, 18, 19, 20, 21]。Hülskamp等[ 22]在研究拟南芥叶表皮毛和根毛的基础上, 提出一个关于表皮毛模式形成的激活侧抑制模型(A-LI), 即 GL1编码的R2R3-MYB转录因子和bHLH类转录因子相互作用, 结合到下游基因启动子上, 激活下游基因在叶表皮细胞中表达, 来控制叶表皮细胞的特化[ 23, 24, 25, 26, 27], 负调控因子则通过与R2R3-MYB转录因子的竞争作用抑制表皮毛的生成[ 28, 29], Wang等[ 30]在亚洲棉中发现了类似的控制棉花纤维起始的激活侧抑制模型。因此在棉花中利用同源克隆得到与拟南芥表皮毛起始相关R2R3-MYB转录因子基因, 验证其功能是研究棉纤维发育的一个有效途径[ 31, 32, 33]。
本研究中, 雷蒙德氏棉D5基因组作为四倍体陆地棉D基因组的供体组, 氨基酸序列分析发现, 与GrMYB0相比, GhMYB0多了9个氨基酸, 并存在7个氨基酸的差异, 表明在棉花数百万年的进化史中, 一些功能基因发生了突变[ 34]; 另外, 雷蒙德氏棉胚珠表皮细胞不会特化成纤维原始细胞, 所以没有纤维形成, 同时由于纤维原始细胞的分化是由多基因形成的复杂基因网络来指引[ 35], 所以 GrMYB0是否在这一复杂过程中发挥调控作用有待进一步研究。其所编码蛋白与棉花GhMYB4以及大豆GmMYB185同源性最高, Loguercio等[ 8]系统进化分析也发现 GhMYB4与 GL1在同一进化支内。在本研究中, GhMYB0基因的拟南芥超表达株系叶片表皮毛密度减小, 与拟南芥中同源基因 Gl1以及亚洲棉中的R2R3-MYB转录因子GaMYB23的研究结果相符[ 30], 说明该基因过表达对拟南芥表皮毛的生成有抑制作用; 在 gl-1突变体中过表达该基因能够恢复突变性状, 表明 GhMYB0能够发挥 GL1在拟南芥中同样的调控作用。综上所述, 在促使表皮毛细胞特化机制中, GhMYB0发挥着一种精细的调控作用, 其表达量对于表皮毛细胞特化数量存在一定阈值, 野生型中过表达会抑制表皮毛的形成, 而在突变体内刚好可以互补 GL1的作用。另外野生型中过表达株系植株矮化, 花器官畸形, 育性降低, 推测目的基因的插入位点处于赤霉素受体AtGID1上, 导致该株系与赤霉素受体AtGID1的突变体表型相似[ 23], 使其育性下降, 该研究结果为筛选拟南芥不育突变体提供了种质资源。同时 GhMYB0在徐州142开花当天(0DPA)开始高调表达, 开花后20天(DPA)达到最大, 表明该基因对棉花纤维的起始和伸长都有一定作用, 为研究R2R3-MYB转录因子在棉纤维起始和伸长过程中的调控作用提供了证据, 目前正在通过转基因棉花进一步证实该结论.
从陆地棉徐州142中克隆了一个编码核定位R2R3-MYB转录因子的 GhMYB0。该基因包含2个外显子和1个内含子, ORF全长843 bp, 编码280个氨基酸。 GhMYB0在徐州142开花当天(0 DPA)开始高调表达, 开花后20 DPA达到最大; 超表达野生型拟南芥叶片表皮毛受到抑制, 突变体中过表达则可恢复其表型。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|