F-box蛋白参与细胞周期调控、细胞凋亡及信号转导等多种生命活动, 对维持植物正常生长发育a和介导非生物胁迫响应等过程发挥重要作用。谷子具有显著的耐旱耐瘠薄等特性, 本研究根据谷子转录组分析结果, 从525个F-box家族成员中鉴定出19个在干旱胁迫下表达量上调的F-box基因, 根据序列相似性将其分为6类, 同一类基因具有相似的内含子-外显子结构。染色体定位分析发现, 这些基因分别分布在谷子的8条染色体上, 其中, 第2条染色体上含F-box基因最多, 有6个。结构域分析结果表明, 19个F-box蛋白均含保守的F-box结构域, 而C端含FBD、WD40、FBA、ZnF、Kelch和LRR等结构域。启动子元件分析表明, 谷子19个F-box基因均含逆境应答元件, 其中, MYB和MYC元件的数量最多(9~78个), 说明这些基因对干旱应答反应可能主要受MYB、MYC转录因子调控。转录组分析结果表明,
F-box proteins play critical roles in plant growth and development and respond to abiotic stresses, involving in cell-cycle regulation, cell apoptosis and signal transduction. Foxtail millet has a character with drought-tolerance. We identified 19 up-regulated F-box genes from 525 F-box families under stress in foxtail millet based on the transcriptome sequencing and divided them into six subgroups, and the genes in the same subgroup had the same intron-exon structure. These genes were unevenly distributed on eight foxtail millet chromosomes, and there were six genes on chromosome 2, that gene number was more than that contained on other chromosomes. The analysis of conserved domain of 19 genes showed that all the protein sequences had a conserved F-box motif, and several other domain also existed in C-terminal including FBD, WD40, FBA, ZnF, and Kelch LRR. Putative
在适应干旱等非生物逆境胁迫中, 植物不断进化自身防御系统以减少水分损失或增加水分吸收等[ 1]。蛋白质的降解在植物逆境信号的响应、细胞信号转导等过程中发挥重要作用, 对于维持植物正常生长发育具有重要意义[ 2]。大多数蛋白通过泛素/26S蛋白酶体途径降解。泛素介导的蛋白质降解过程主要有3种酶参与, 包括泛素活化酶(E1), 泛素结合酶(E2)及泛素连接酶(E3)。在ATP供能的情况下, E1的半胱氨酸残基与泛素C末端的甘氨酸形成高能硫酯键, 使泛素激活, 被转移到E2的丝氨酸残基上, 随后E2和E3共同识别底物蛋白, 对其进行泛素化修饰。E3在泛素化-酶体途径的底物特异识别过程中起着关键作用。目前已经发现不同类型的E3[ 3], 主要的一种是Skp1-Cullin1-F-box (SCF)复合体, 由4个亚基组成, 分别为Cullin、SKP1、RBX1和F-box蛋白[ 4, 5]。每个F-box蛋白家族成员的N端都至少含1个大约40~50个保守氨基酸的F-box基序, 并与Skp1结合; C端是结构不同的蛋白-蛋白相互作用结构域, 包括亮氨酸拉链(LRR)、Kelch和WD40等。
F-box蛋白最早在cyclin F的N端被发现而命名[ 6], 广泛存在于真核生物中, 参与多种植物激素信号的传导、逆境胁迫应答等, 在调节光形态建成、光周期节律、花器官发育及自交不亲和中发挥着重要功能。植物中发现的第一个F-box蛋白是Unusual Floral Organs (UFO), 参与了植物花分生组织与花器官的建成[ 7, 8, 9, 10]。LKP1/ZTL、LKP2/FKL和FKF1可以调控光形态建成、调节生物钟的昼夜周期节律及开花时间[ 11, 12, 13, 14]。F-box蛋白还与植物的自交不亲和性相关, 许多植物的自交不亲和性由S位点控制, 研究表明, 与S位点相关的S-locus-F-box genes (SLF)能在花粉中特异表达[ 15, 16, 17, 18]。
许多F-box蛋白还参与激素的信号传导, TIR1是生长素受体蛋白, 与AUX/IAA发生相互作用, 导致AUX/IAA降解[ 19, 20, 21, 22]; EBF1过量表达引起植物对乙烯不敏感, ebf1和 ebf2突变株则表现出组成型乙烯反应, EBF1/EBF2两者之一发生突变都导致植株对乙烯反应增强, 而双突变体则表现出三重反应[ 23, 24]。赤霉素(GA)的信号通路也可以通过F-box蛋白(SLY1)调节[ 25, 26]。COI1编码F-box蛋白, 在茉莉酸(JA)信号通路中起关键作用[ 27, 28, 29]。
F-box蛋白参与非生物胁迫应答的调控, 如DOR1 (drought tolerance repressor 1)是S位点类F-box蛋白家族的基因, 在保卫细胞中表达, dor使ABA表达量上调, 气孔关闭, 从而增加抗旱性[ 30]。MAX2 (more axillary growth 2)对干旱响应明显, 与野生型相比, max2的保卫细胞对ABA敏感程度降低, 角质层变薄, 在MAX2突变的情况下, 与ABA合成、运输及代谢有关的基因表达量都降低[ 31]。
随着全球水资源短缺和气候变暖形势的日益严峻, 对作物的抗旱节水性进行遗传改良, 提高作物水分利用率, 已成为目前研究的一个热点。谷子具有耐旱、耐贫瘠等特点, 是一种抗逆性很强的作物。然而, 目前, 植物中关于F-box蛋白的研究主要集中在拟南芥、水稻、玉米中。植物已知功能的F-box蛋白只有30个, 其中22个来源于拟南芥(见附表), 谷子中尚未见报道。本研究对谷子进行了转录组测序分析, 筛选出19个在干旱胁迫下表达量上调的F-box基因, 对其基因结构、系统发育关系及染色体定位等进行分析, 并选择对干旱应答最强的基因 SiF-box18作了具体分析, 为进一步研究F-box家族的功能及 SiF-box18基因的功能提供了依据。
谷子Tie-8396种子由中国农业科学院作物科学研究所刁现民研究员课题组提供。谷子幼苗在温度22℃、相对湿度65%、光照周期16 h/8 h的条件下培养。生长3周的幼苗经干旱处理1周, 送华大基因进行转录组测序。
谷子( Setaria italica)基因组数据和cDNA数据均下载于谷子最新数据库版本Phytozome (http://www.phytozome.net/search.php)。利用SMART (http://smart.embl-heidelberg.de/)在线工具分析F-box蛋白结构域, 利用DOG 2.0[ 32]绘制19个F-box家族蛋白的功能结构域示意图。
使用MEGA5对19个谷子F-box基因进行多序列比对分析, 将分析结果用邻接法生成系统进化树, Bootstrap值设置为1000。利用GSDS在线工具(http:// gsds.cbi.pku.edu.cn/)制作谷子F-box家族基因外显子- 内含子结构示意图。
通过MapDraw程序显示每个基因在染色体上的位置, 得到各个基因在染色体上分布状况。采用Yang等[ 33]的方法, 判别同源基因, 即2个基因联配部分序列的长度要覆盖2个基因中较长序列的80%和联配部分序列的相似性大于70%。利用DNAMAN分析谷子中F-box家族基因中的重复片段, 并将分析结果标注在染色体上。从谷子基因组数据库 Phytozome (http://www.phytozome.net/search.php)中选取谷子F-box基因起始密码子上游2000 bp序列作为启动子区, 利用植物顺式作用元件数据库PLACE26.0 (http://www.dna.affrc.go.jp/PLACE/)分析基因的顺式作用元件。
根据预测的谷子 SiF-box18基因的CDS序列, 用Primer Premier 5.0设计引物F: 5′-ATG GCG GAG CGG AAG-3′和R: 5′-TCA CCA TAC TGG GAA CAA AGG-3′。用TRIzol试剂盒(TIANGEN, 北京)提取叶片总RNA, 用Primer Script (TaKaRa)反转录试剂盒获得cDNA为模板扩增 SiF-box18, 并将其连接到pEASY-Blunt载体上。
对生长3周的谷子幼苗分别进行干旱(6% PEG)、NaCl (100 mmol L-1)、茉莉酸(JA, 100 μmol L-1)、脱落酸(ABA, 100 μmol L-1)、赤霉素(GA, 100 μmol L-1)和水杨酸(SA, 100 μmol L-1)处理, 并于0、和24 h分别取样。利用TRIzol试剂盒(TIANGEN, 北京)提取总RNA, 分别利用6种胁迫下的谷子总RNA作为模板, 以SYBR Green染料法进行荧光定量Real-time PCR。用实时荧光定量PCR仪(ABI Prism 7300)进行PCR扩增。反应体系包括2× TaqPCR Master Mix (含荧光染料) 9 μL、10 μmol L-1引物各0.5 μL、ddH2O 9 μL和cDNA模板1 μL。反应条件为95℃ 5 min; 95℃ 15 s, 56℃ 20 s, 72℃ 30 s, 60℃ 30 s, 35个循环。PCR引物序列为F: 5′-GGA TGG AAA ACG GAG TCG-3′和R: 5′-TCA GGG TGA AAG TAG CGC-3′。以谷子 Actin基因( Si001873)为内标, 引物序列为Actin-F: 5′-GGC AAA CAG GGA GAA GAT GA-3′和Actin-R: 5′-GAG GTT GTC GGT AAG GTC ACG-3′。用2-∆∆Ct法计算各基因表达量。
采用In-Fusion (TaKaRa)单酶切技术( BamH I)构建SiF-box18::hGFP融合表达蛋白(F: 5′-TAT CTC TAG AGG ATC CAT GGC GGA GCG GAA G -3′, R: 5′-TGC TCA CCA TGG ATC CCC ATA CTG GGA ACA AAG G-3′)。取4周左右的拟南芥叶片切成细条, 放入15 mL新鲜酶解液中, 避光, 23℃抽真空酶解3 h, 制备原生质体。取融合表达的重组质粒转化原生质体[ 34]。12~16 h后于激光共焦显微镜下观察。
通过NCBI搜索, 谷子F-box基因家族有525个成员。通过谷子干旱转录组测序, 发现有19个F-box基因上调表达。用Pfam进行结构域分析发现这19个基因都具有完整的F-box结构域(图1), 并将这19个基因命名为 SiF-box1~ SiF-box19。这些F-box家族成员中蛋白最长有767个氨基酸, 最短的有155个氨基酸, 其等电点范围在4.90~11.48之间(表1)。
| 表1 谷子F-box家族基因的基本信息 Table 1 Basic information of F-box family genes in foxtail millet |
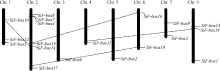
通过Pfam结构域分析显示, 19个谷子F-box蛋白家族均含有F-box保守域(图1)。每个F-box蛋白的N端均有一段保守的F-box基序, 而C端包含不同的蛋白-蛋白相互作用结构域(图2)。根据图中显示, 19个F-box蛋白中, 26.3%的蛋白只含有F-box结构域, 其他蛋白含有多个功能域, 包括Fibrinogen- like domain (FBD)、WD40 repeats (WD40)、F-box associated (FBA)、Zinc finger domain (ZnF)、Kelch repeats (Kelch)和Leucine-rich repeat (LRR)。例如, SiF-box1除了含有F-box基序外, 还包括FBA结构域; SiF-box16含有F-box、LRR和ZnF 3个结构域。有些蛋白不只包含1个F-box基序, SiF-box9与SiF- box13分别包含2个F-box基序, 而SiF-box15中有3个F-box基序。
对谷子19个F-box基因进行多重序列比对和系统进化分析后, 19个F-box基因划分为6类(图3)。内含子-外显子结构分析显示, 同一亚族的基因具有相似内含子-外显子结构。第I类包含 SiF-box6、SiF-box7、SiF-box2、SiF-box3和 SiF-box4, 这5个基因中, 没有或只包含1个内含子; 第II类中, SiF-box13和 SiF-box14含有3个内含子, 都有FBA结构; 第III类包含 SiF-box5、 SiF-box12、 SiF-box17和 SiF-box19, 其中, SiF-box5和 SiF-box12都含有kelch repeats; 第IV类只包含 SiF-box1一个基因, 没有内含子, 包含FBA结构; 第V类中, SiF-box8、 SiF-box9、 SiF-box15和 SiF-box16都包含F-box、LRR和ZnF结构域, 除 SiF-box8外, 其他3个基因都含有2个或2个以上内含子; 第VI类包含 SiF-box11与 SiF-box18, 只含有F-box基序, 没有其他结构域。
根据基因位置信息绘制了19个F-box家族基因在谷子染色体上的定位图(图4)。19个基因分布在8条谷子染色体上, 其中第8染色体上不含谷子F-box家族基因, 第2染色体上含F-box家族基因最多, 第1和第9染色体上都有3个F-box家族基因, 其他各染色体上含1~2个F-box家族基因。
为了确定可能的进化机制, 研究了基因的复制现象, 依据系统发育树及序列相似性, 发现了4对旁系同源基因, 分别为 SiF-box10与 SiF-box14, SiF-box15与 SiF-box16, SiF-box17与 SiF-box19, 以及 SiF-box11与 Si-Fbox18基因 。
目前发现的与干旱胁迫相关的元件主要有ABRE (ABA-responsive element)、DRE (dehydration- responsive element)、LTRE (low temperature responsive element)、MYB和MYC元件。本研究选择起始密码子上游2000 bp序列作为启动子区, 研究19个F-box家族基因启动子区元件。结果显示, 谷子F-box家族基因启动子区域均含多个MYB和MYC元件, 其中, MYB数量最少为9个, 最多为78个, MYC的数量12~40个。MYB和MYC都属于转录因子, 与干旱、ABA的诱导表达相关。此外, 在这19个F-box家族基因中, 除 SiF-box12外, 在启动子区均含ABRE作用元件; 63.2%含DRE作用元件; 84.2%含LTRE作用元件(表2), 说明F-box家族基因与植物的逆境胁迫应答具有很强的关联。
| 表2 谷子F-box家族基因启动子逆境相关顺式作用元件的数量分布 Table 2 Distribution of cis-acting elements in foxtail millet F-box gene promoters |
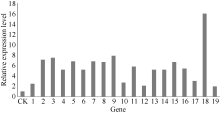
19个上调表达的谷子 F-box基因中, 表达量差异明显(图5), SiF-box1、 SiF-box4、SiF-box6、SiF- box10、SiF-box12、SiF-box17和 SiF-box19的相对表达量介于1~5倍; SiF-box2、SiF-box3、SiF-box5、SiF- box7、SiF-box8、 SiF-box9、SiF-box11、SiF-box13、SiF- box14、SiF-box15、SiF-box16、SiF-box17、SiF-box19基因的表达量介于5~10倍之间; 高于10倍的只有 SiF-box18基因。与对照相比 SiF-box18基因的表达倍数是16倍, 因此选择 Si-Fbox18基因进一步分析。
为了进一步解析非生物胁迫和外源激素对 SiF-box18基因表达的影响, 对谷子幼苗分别进行干旱、NaCl、茉莉酸(JA)、脱落酸(ABA)、赤霉素(GA)和水杨酸(SA)胁迫处理, 通过荧光定量PCR检测不同处理条件下 SiF-box18的表达量。如图6所示, 在不同处理条件下, SiF-box18基因的表达量在24 h均达到最高。干旱胁迫下的表达倍数最高达到42倍, GA处理条件下表达量最低, 其他胁迫条件下的表达倍数, NaCl为18倍, ABA为5倍, JA与SA均为10倍。
将SiF-box18构建到荧光表达蛋白GFP的N端。利用原生质体转化技术, 将SiF-box18-hGFP融合表达载体和空载体分别转入拟南芥原生质体, 在激光共聚焦显微镜下观察。绿色荧光蛋白在拟南芥原生质体中获得高效瞬时表达, 可以观察到对照GFP蛋白在细胞核、细胞膜、细胞质中均有绿色荧光信号; 而SiF-box18融合蛋白只在细胞核中有绿色荧光信号, 表明SiF-box18蛋白定位在细胞核中(图7)。
F-box蛋白是一个庞大的蛋白家族, 广泛存在于真核生物中。酵母、果蝇、人类、老鼠中分别有11、22、68和74个F-box蛋白成员; 而线虫中包含326个F-box蛋白家族成员[ 35]。植物中有大量的F-box蛋白存在, 拟南芥中大约有600~700个F-box基因[ 36, 37], 水稻中包含687个F-box基因[ 38]; 最近研究表明, 玉米中有359个F-box蛋白成员[ 39]。根据C端蛋白-蛋白相互作用结构域的不同, 可以把拟南芥F-box家族分为19个亚族[ 36, 37]; 水稻F-box家族分为10个亚族[ 38]; 玉米中分为13个亚族[ 39]。谷子F-box家族通过NCBI搜索, 发现有525个F-box基因, 进一步研究了其中19个受干旱诱导表达的F-box基因。
除N端保守的F-box基序外, 谷子F-box蛋白C端含有不同的蛋白-蛋白相互作用结构域, 例如, FBD、FBA、LRR、WD40、PPR和ZnF。本研究根据序列相似性, 将响应干旱胁迫的谷子19个F-box蛋白分为6类(图3)。第II类中, SiF-box13和SiF-box14包含FBA结构, FBA在泛素化中起重要作用, 可以识别靶蛋白; 第III类中SiF-box5和SiF-box12都含有Kelch repeats, Kelch是植物F-box蛋白特有的结构域[ 38]; 第IV只有SiF-box1一个蛋白, 包含FBA结构; 第V类中, 4个成员都包含F-box、LRR和FBD结构域, LRRs是Leu重复单元, 含有LRRs结构的蛋白一般与信号传导, DNA修复、重组、转录以及抗病等功能相关[ 40]; 第VI类含有F-box基序。此外, SiF-box6和SiF-box15蛋白还包含ZnF结构; 在谷子19个F-box家族中, 3个蛋白包含WD40结构, WD40结构存在于所有真核生物中, 在信号传导、细胞周期调控、细胞凋亡方面起着重要作用[ 41]。说明受干旱诱导表达的谷子F-box蛋白可能同样发挥着调控细胞周期、细胞信号传导等功能。
基因重复是基因组进化及基因家族扩大的重要因素。基因重复后会产生3种结果: (1)基因功能的缺失, 基因的2个拷贝中的一个拷贝由于突变失去功能; (2)产生新的功能, 基因的2个拷贝中的一个拷贝获得新的功能并通过自然选择被保留下来; (3)基因功能互补, 基因的2个拷贝都发生退化突变, 使得2个拷贝功能产生互补作用。本研究发现19个F-box基因有4对旁系同源基因, 这4对基因位于不同的染色体上, 可能是复制后功能产生了分化。
据报道, F-box基因不仅参与信号传导等生命活动[ 42], 还介导干旱等非生物胁迫响应。例如, 干旱条件下, dor转基因拟南芥植株表现出比野生型更强的抗性, 证明 DOR对干旱胁迫起负调控作用[ 30]; 在渗透胁迫和高盐胁迫下, DEL3 (EID1-like protein3)高效、快速表达[ 43]。本研究发现19个受干旱诱导表达的谷子F-box基因, 启动子区都包含多种非生物逆境相关元件, 而MYB和MYC的数量远远大于其他元件的数量, 说明F-box基因对干旱胁迫的响应主要通过MYB和MYC转录因子调控。
植物对干旱的响应是一个复杂的过程, 受不同分子和细胞通路的调控。根据干旱胁迫信号转导过程中, 胁迫相关基因的表达是否依赖ABA, 存在两种途径, 其中, ABA依赖型途径通过ABRE结合bZIP转录因子或者通过结合MYB/MYC转录因子激活下游基因的表达; 非ABA依赖型途径则由信号激活传感蛋白, 传递给DREB后结合DRE元件启动下游抗逆基因的表达。研究表明, 与干旱胁迫相关的F-box基因家族基因, 对ABA有不同程度的响应, 例如, ABA处理条件下, DOR表达量降低, 对ABA起负调作用[ 30]; 与对照相比, DEL3的表达量上升, edl3-1表达量下降[ 43]。在ABA胁迫下, SiF-box18基因表达量在24 h达到最高, 是对照的5倍(图6)。推测 SiF-box18基因可能通过依赖ABA途径参与谷子的干旱胁迫。
在干旱和高盐胁迫下, SiF-box18基因的表达量在24 h达到最高, 分别是对照的42倍和18倍(图6)。在植物外源激素GA、SA、JA、ABA处理下, SiF-box18基因表达量均有不同程度的增加(图6), 说明 SiF- box18基因可能参与了GA、SA、JA等植物激素的信号传导过程。 SiF-box18基因的启动子区包含1个ABRE、4个DRE、3个LTRE、45个MYB和14个MYC等多种非生物胁迫响应元件。说明 Si-Fbox18基因可能参与干旱、低温等多种非生物胁迫响应。
19个上调表达的谷子F-box基因位于8条染色体上, 其中SiF-box18蛋白被定位在细胞核中, 对干旱、高盐和ABA等都有响应, 推测 SiF-box18基因可能在非生物胁迫响应中起重要的作用。
植物中已知功能的F-box蛋白
Supplemental Table F-box proteins with known function in plants
| F-box protein | Gene identifier | Name | Organism | Function | Reference | |
|---|---|---|---|---|---|---|
| COI1 | At2g39940 | Coronatine Insensitive 1 | Arabidopsis | Jasmonate-Regulated defense responses, pollen fertility | [ 1] [ 2] [ 3] | |
| ORE9/ MAX2 | At2g42620 | Oresara 9/ More Axillary 2 | Arabidopsis | Regulation of leaf senescence, selective repression of axillary shoots | [ 4] [ 5] [ 6] | |
| TIR1 | At3g62980 | Transport Inhibitor Response 1 | Arabidopsis | Auxin receptor | [ 7] [ 8] [ 9] [ 10] | |
| AFB1/GRH1 | At4g03190 | Auxin signaling F Box protein 1 | Arabidopsis | Auxin receptor, plant growth and development | [ 11] [ 12] [ 13] | |
| AFB2 | At3g26810 | Auxin signaling F Box protein 2 | Arabidopsis | Auxin receptor, plant growth and development, glucose repression | [ 11] [ 12] [ 13] | |
| AFB3 | At1g12820 | Auxin signaling F Box protein 3 | Arabidopsis | Auxin receptor, plant growth and development | [ 11] [ 12] [ 13] | |
| AFB4 | At4g24390 | Auxin-signaling F-box protein 4 | Arabidopsis | Plant growth, development and innate immunity | [ 14] | |
| SON1 | At2g17310 | Suppressor Of nim1-1 | Arabidopsis | Defense responses | [ 15] | |
| UFO | At1g30950 | Unusual Floral Organs | Arabidopsis | Determination of floral meristem and floral organ identity | [ 16] [ 17] [ 18] [ 19] | |
| EID1 | At4g02440 | Empfindlicher Im Dunkelroten Licht 1 | Arabidopsis | Negative regulator in phytochrome A-specific light signaling | [ 20] [ 21] | |
| AFR | At2g24540 | Attenuated Far-Red Response | Arabidopsis | Positive regulator of phytochrome A-mediated light signaling | [ 22] | |
| EBF1 | At2g25490 | EIN3-Binding F box protein 1 | Arabidopsis | Negative regulator of ethylene-signaling, plant growth | [ 23] [ 24] | |
| EBF2 | At5g25350 | EIN3-Binding F box protein 2 | Arabidopsis | Negative regulator of ethylene-signaling, plant growth | [ 23] [ 24] | |
| ZTL/LKP1 | At5g57360 | Zeitlupe | Arabidopsis | Circadian rhythm, photomorphogenesis and flowering time | [ 25] [ 26] | |
| LKP2/FKL/ FKF1 | At2g18915 | Lov Kelch Protein 2 | Arabidopsis | Circadian clock regulation and flowering time control | [ 27] [ 28] [ 29] [ 30] [ 31] | |
| SLY1 | At4g24210 | Sleepy 1 | Arabidopsis | Positive regulator of Gibberellic acid signaling | [ 32] [ 33] | |
| SNE | At5g48170 | Sneezy | Arabidopsis | Positive regulator of Gibberellic acid signaling | [ 34] [ 35] | |
| CEG (AtSFL61) | At3g22650 | Cegenduo | Arabidopsis | Negative regulator of auxin-mediated lateral root formation | [ 36] | |
| DOR | At2g31470 | Drought Tolerance Repressor 1 | Arabidopsis | Negative regulate drought stress | [ 37] | |
| CFK1 | At5g42350 | COP9 INTERACTING F-BOX KELCH 1 | Arabidopsis | Controlling hypocotyl elongation | [ 38] | |
| FOA1 | AT3G17320 | F-box overexpressed/ oppressed ABA signaling | Arabidopsis | Play a negative role in ABA signaling. | [ 39] | |
| EDL3 | At3g63070 | EID1-like protein 3 | Arabidopsis | Negative acting factors in ABA nsignalling to degradation via the proteasome. | [ 40] | |
| GID2 | AB100246 | Gibberellin Insensitive Dwarf 2 | Oryza sativa | Positive regulator of Gibberellic acid signaling | [ 41] [ 42] | |
| D3 | AK065478 | Dwarf 3 | O. sativa | Control of tiller bud activity | [ 43] [ 44] | |
| FBK12 | OS03g03570 | F-box protein containing a kelch repeat motif | O. sativa | Delay in leaf senescence and germination, and increased seed size | [ 45] | |
| DDF1 | Dwarf and deformed flower 1 | O. sativa | Regulate vegetative growth and floral organ specification | [ 46] | ||
| MAIF1 | Os02g44990 | miRNAs regulated and abiotic stress induced F-box gene | O. sativa | Plays the negative role in response to abiotic stress | [ 47] | |
| FIM | S71192 | Fimbriata | Antirrhinum majus | Floral differentiation | [ 48] [ 49] | |
| AhSLF-S2 | AJ297974 | Self-incompatibility Locus-encoded F-box protein | A. hispanicum | Controls pollen function of S-RNase-based self-incompatibility | [ 50] [ 51] [ 52] | |
| SFB3 | AY571665 | S haplotype-specific F-box protein | Prunus avium | Male determinant of gametophytic self incompatibility | [ 53] [ 54] [ 55] | |
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|