第一作者联系方式: E-mail: shixh_1988@163.com
大豆胞囊线虫(soybean cyst nematode, SCN)严重危害世界大豆生产, Rhg4(resistance to Heterodera glycines 4)是控制大豆SCN抗性的2个主效位点之一。本研究针对 Rhg4 ( GmSHMT)上的2个单核苷酸多态性(single nucleotide polymorphisms, SNP)位点开发了快速、经济、简便易行的CAPS (Rhg4-389)和dCAPS标记(Rhg4-1165), 并用开发的2个标记鉴定了以大豆胞囊线虫应用核心种质为主的193份代表性抗感种质。结果表明, Rhg4-389和Rhg4-1165位点间存在显著连锁不平衡( P=0.0001, r2=0.87), 可形成4种单倍型。Rhg4-389-G/Rhg4-1165-T和Rhg4-389-C/Rhg4-1165-A为优势单倍型, 稀有单倍型Rhg4-389-G/Rhg4-1165-A和Rhg4-389-C/Rhg4-1165-T是在中国抗源中新发现的单倍型。结合193份种质对SCN 3号小种抗性鉴定分析发现, Rhg4-389-G和Rhg4-1165-T主要存在于抗病种质, 它们形成的单倍型对抗病种质鉴定效率可达94.1%。本研究开发了可用于辅助大豆SCN抗性鉴定且方便育种家利用的CAPS/dCAPS标记, 且用其摸清了应用核心种质等重要抗源在 Rhg4位点的“本底”, 为育种家有效利用这些优异抗源提供了重要信息。
Soybean cyst nematode (SCN, Heterodera glycinesIchinohe) is one of the most destructive diseases which largely suppressed soybean production worldwide. Rhg4 was one of two major resistant loci for SCN. In this study two nonsynonymous SNPs (Rhg4-389-G/C and Rhg4-1165-T/A) in GmSHMT were deduced to confer SCN resistance or susceptibility. One CAPS marker (Rhg4-389) and one dCAPS marker (Rhg4-1165) with the advantages of rapid, economic, simple and easy way to use were developed based on two nonsynonymous SNPs (Rhg4-389-G/C and Rhg4-1165-T/A), respectively. Then, a total of 193 soybean cultivars mainly from SCN-applied-core collection were genotyped by markers Rhg4-389 and Rhg4-1165. Linkage disequilibrium (LD) analysis indicated pairwise of Rhg4-389 and Rhg4-1165 was in significant LD ( P-value = 0.0001) with r2 of 0.87. Among four haplotypes detected in this panel, Rhg4-389-G/Rhg4-1165-T and Rhg4-389-C/Rhg4-1165-A were predominant; while both Rhg4-389-G/Rhg4-1165-A and Rhg4-389-C/Rhg4-1165-T were rare, which only discovered in the resistant cultivars from China. Rhg4-389-G and Rhg4-1165-T alleles mainly occurred in resistant cultivars, and almost coincided with Rhg4-389-G/ Rhg4-1165-T haplotype. Of 101 resistant cultivars with Rhg4-389-G/Rhg4-1165-T haplotype, 94.1% (95) were resistant to SCN. In this study, we not only developed convenient CAPS/dCAPS markers for marker assisted selection (MAS) breeding, but also dissected the genetic architecture of Rhg4 locus among SCN-applied-core collection. Hopefully, this study will provide helpful information for improving the utilization efficiency of resistant resources in soybean breeding.
大豆胞囊线虫病(soybean cyst nematode, SCN)是危害世界大豆(Glycine maxL.Merr.)生产的严重病害, 种植抗病品种、合理轮作非寄主作物是当前防治该病最经济有效的措施[1]。我国是大豆起源地, 拥有丰富的抗源, 国际上广泛应用的抗源如Peking (PI 548402)、PI 88788、PI 437654等(http://www.soybase.org/)皆来自我国。1986— 1990年, 大豆种质抗SCN鉴定协作组鉴定了我国1万余份大豆种质资源对大豆胞囊线虫1号、3号和4号生理小种的抗性, 筛选出一批免疫和高抗种质[2]。Ma等[3]以此为基础, 根据表型性状和SSR标记分析结果, 浓缩了国内外优异抗源的遗传多样性, 构建了蕴含丰富抗病等位变异的大豆胞囊线虫初选应用和应用核心种质。虽然百余份抗病资源被鉴定出来, 但我国大豆生产上应用的抗病品种仍然存在抗性单一、遗传基础狭窄的问题, 其原因是绝大多数优异抗源的遗传基础尚不清楚, 迄今只有北京小黑豆/哈尔滨小黑豆等少数抗源用于育种和生产[4]。因此, 有必要阐明这些抗源的分子遗传基础, 以促进抗源的有效利用。
大豆胞囊线虫病是由多基因控制的复杂数量性状, rhg1和Rhg4 (resistance to Heterodera glycines 1 and 4)抗性基因是控制大豆胞囊线虫病抗性的2个主效基因。其中, Rhg4基因(GmSHMT)已通过图位克隆获得, 该基因编码一种丝氨酸羟甲基转移酶(SHMT), 在一碳单位代谢过程中调控丝氨酸和甘氨酸相互转化, 突变体分析、基因沉默和转基因互补等实验皆证明该基因与抗性相关[5]。并通过28份大豆抗感资源Rhg4基因测序分析, 初步推断2个非同义突变SNP位点Rhg4-389(G/C)和Rhg4-1165(T/A)的碱基变化可能与大豆胞囊线虫抗性相关[5]。这2个位点在28份抗感资源中只形成Rhg4-389-G/Rhg4- 1165-T和Rhg4-389-C/ Rhg4-1165-A两种单倍型, 未发现发生位点间重组交换。其中携带Rhg4-389-G/ Rhg4-1165-T单倍型的6份(100%)大豆资源对SCN皆表现抗病, 携带Rhg4-389-C/Rhg4-1165-A单倍型的22份种质中, 18份(82%)对SCN表现感病。但因为鉴定资源数较少, 有必要扩大分析群体进行验证。
酶切扩增多态性序列(cleaved amplified polymorphic sequence, CAPS)标记是利用特异引物PCR与限制性酶切相结合而产生的一种检测SNP位点的DNA标记[6]。为了检测无限制性酶切位点的SNP位点, Michaels和Amasino[7]及Neff[8]在CAPS标记基础上, 开发了人工引入错配碱基的衍生酶切扩增多态性标记(derived cleaved amplified polymorphic sequence, dCAPS)。无论是CAPS还是dCAPS标记, 皆具有共显性、位点特异性、操作简单和成本低等特点, 已广泛用于作物的分子鉴定、基因定位、图位克隆和辅助育种等[9, 10, 11, 12, 13, 14, 15, 16, 17]。
本研究针对大豆抗胞囊线虫主要抗病基因Rhg4上发现的2个与抗性相关的非同义突变SNP位点分别开发CAPS和dCAPS标记, 鉴定国内外抗源的基因型, 阐明抗源在Rhg4位点的遗传背景, 并评价开发标记及单倍型在资源辅助鉴定中的效率, 为分子标记辅助大豆胞囊线虫抗病品种的选育提供标记和材料信息。
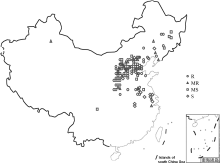
根据Ma等[3]和Chen等[18]文献报道, 筛选出以我国大豆胞囊线虫初选应用核心种质为主的150份抗源(3号生理小种)和43份来源广泛的感病资源, 共193份, 包括165份(85.5%)地方品种和16份选育品种。大部分供试材料(87%, 168)来自我国的东北(15份)、北方和黄淮海流域(139份)及南方(10份)(图1)。剩余25份资源分别来自日本(10份)、韩国(7份)、俄罗斯(2份)、美国(2份)、法国(1份)、澳大利亚(1份)、波兰(1份)和阿根廷(1份)[18]。
取每份种质3粒种子于温室育苗, 并单株繁种, 取其新鲜的三出复叶100 mg于2 mL离心管中(加钢珠), 速放入液氮冷冻, 用打样机捣碎叶片(2000 GENO/GRINDER)后按照快速DNA提取试剂盒(MBI Fermentas公司)的操作指南提取植物基因组总DNA保存于100 μ L去离子水中。所得DNA经1%琼脂糖凝胶电泳检测, 并使用紫外分光光度计(UV-Vis Spectrophotometer Q5000)测其浓度。
采用Riggs和Schmitt设计的大豆胞囊线虫生理小种鉴别模式[19], 2014年在黑龙江农业科学院田间病圃与温室2种环境条件下对193份大豆资源(其中包括5个鉴别寄主Pickett、Peking、PI 90763、PI 88788和Lee)[20]进行大豆胞囊线虫3号小种抗性鉴定。田间采用完全随机区组试验, 重复3次。田间种植行宽0.65 m, 行长1.50 m, 株间距0.05 m。温室采用盆栽鉴定, 每份种质3盆, 每盆5株。出苗30 d后, 对田间和温室的每份种质均随机选择长势相同10株进行鉴定, 数每株胞囊数, 计算每份鉴定种质的平均胞囊数。为了检测胞囊指数(female index, FI)差异, 每隔40行以中品03-5373作为抗病对照, 中黄13作为感病对照。以基于胞囊指数划分的抗病等级评估每份种质的抗性。胞囊指数FI=(鉴定品种每株平均胞囊数/感病对照品种的平均胞囊数)× 100%, 其中鉴别寄主的胞囊指数以Lee为感病对照品种, 根
据鉴别寄主的抗性确定生理小种, 其他193份大豆资源以中黄13为感病对照品种计算胞囊指数。以Schmmit等[21]提出的抗感指标为分级标准, FI< 10为抗病(resistant, R); 10≤ FI< 30为中抗(moderately resistant, MR); 30≤ FI< 60为中感(moderately susceptible, MS); FI=60+为感病(susceptible, S)。
从GenBank (http://www.ncbi.nlm.nih.gov/nuccore/JQ714083)下载Rhg4 (GmSHMT)基因序列(GenBank登录号为JQ714083, 来自抗病种质Forrest), 根据文献报道定位已知的2个候选SNP位点, 以已报道的与大豆胞囊线虫抗性相关的2个非同义突变位点(Rhg4- 389和Rhg4-1165)[5]为候选SNP位点, 分别开发CAPS和dCAPS标记。利用在线软件dCAPS Finder 2.0 (http://helix.wustl.edu/dcaps/dcaps.html)确认SNP位点酶切信息, 其中Rhg4-389位点存在限制性酶切位点, 开发CAPS标记; Rhg4-1165位点不存在限制性酶切位点, 利用反向互补序列, 人工引入一个错配碱基, 发现限制性酶切位点, 开发dCAPS标记, 分别选择特异、经济的内切酶Bsr B I和Hin f I作为候选标记所用酶。利用Primer 5.0软件[22]在候选CAPS/dCAPS标记位点两侧设计PCR引物(表1), 涵盖Rhg4-389位点的PCR产物预计长度为369 bp, Rhg4-389经Bsr B I酶酶切后理论上可获得237 bp和132 bp片段; 含有Rhg4-1165位点的PCR产物长度为219 bp, 经Hin f I酶酶切后理论上可获得196 bp和23 bp片段。
| 表1 CAPS/dCAPS标记的引物序列、PCR扩增和内切酶信息 Table 1 Summary information of CAPS/dCAPS markers developed |
以基因组DNA为模板, 反应体系20 μ L, 包括50 ng基因组DNA、10× PCR缓冲液, 2 mmol L-1 dNTPs, 2 μ mol L-1引物和1 U Taq聚合酶(全式金生物技术有限公司)。PCR在ABI (Applied Biosystems, 美国)公司的PCR扩增热循环仪上进行, 反应程序为95℃预变性3 min, 94℃变性30 s, 优化退火温度(表1) 40 s, 72℃延伸50 s, 38个循环, 最后72℃延伸8 min, 于4℃保存。对PCR产物采用1.5%琼脂糖凝胶电泳检测, 利用ABI 3730 (Applied Biosystems, 美国)对条带单一的PCR产物进行测序, 采用Multalin (http://multalin.toulouse.inra.fr/multalin/multalin.html)和DNAMAN软件[23]进行测序比对和分析。
酶切分析参照NEB (New England Biolabs)限制性内切酶操作指南, 酶切反应体系10 μ L, PCR产物5 μ L、3 U内切酶、1.5 μ L NEB缓冲液。将酶切反应体系放入37℃保温箱或水浴锅中, 30 min后取出, 采用1.5%琼脂糖凝胶电泳检测CAPS标记的酶切产物; dCAPS标记的酶切产物变性后采用7%变性聚丙烯酰胺凝胶电泳(PAGE)分离, 银染显色检测。
CAPS/dCAPS标记的抗病种质选择效率(%)= (携带某一等位变异的抗病种质数/携带该等位变异的总种质数)× 100%
使用TASSEL v4.0 (http://www.maizegenetics.net/)计算Rhg4-389和Rhg4-1165间的r2(squared allele frequency correlation, 等位基因频率相关系数平方)和D° (standardized disequilibrium coefficient, 标准平衡系数)[24]值, 评估2个SNP位点间的连锁不平衡(linkage disequilibrium, LD)水平。基于Fishers’ exact测验[25]检验位点间LD的显著性。
使用Microsoft Excel 2007进行表型次数分布与单倍型相关性卡方检验[26]。
用鉴别寄主Pickett、Peking、PI 90763、PI 88788检测鉴定结果显示, 黑龙江省农业科学院哈尔滨田间病圃和温室病土皆为3号生理小种(SCN3)(表2)。以Lee作为对照, 中品03-5373表现为免疫(FI为0), 中黄13表现为高感(FI=239)。193份大豆资源的FI (%)变化范围是0~251.34, 呈现连续分布。大多数种质(64.5%, 124份)表现抗病, 19份种质(6.5%)表现感病, 中抗和中感种质分别为26份(13.5%)和24份(12.1%)。在124份抗病和26份中抗种质中, 121份是地方种质, 主要来自我国大豆胞囊线虫初级应用核心种质(112份)。
| 表2 哈尔滨地区大豆胞囊线虫生理小种鉴定结果 Table 2 Determination of physiological races of soybean cyst nematode in some areas of Harbin |
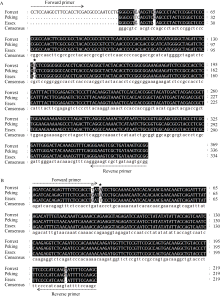
针对2个位点Rhg4-389和Rhg4-1165分别设计的引物, 对9份已知基因型[Peking、PI 90763、Forrest、PI 437654、PI 209332、PI 548316 (Cloud)、PI 88788、Essex、Williams 82]和4份未知基因型(元钵黑豆、赤不流黑豆、抗线1号和泰兴黑豆)的DNA进行PCR扩增, 均获得与目标片段长度大小相近的单一PCR产物。分别取Peking和Essex涵盖Rhg4- 389和Rhg4-1165位点的PCR产物进行测序并与Forrest参考序列比对分析显示, PCR产物皆为Rhg4基因目标片段, 不同种质间在目标SNP位置上存在碱基变化, 与预测一致(图2-A, B)。从图3-A可以看出, CAPS标记Rhg4-389 PCR扩增产物片段大小为369 bp, 等位变异为C; Bsr B I酶切后产生2个片段, 分别为237 bp和132 bp, 等位变异为G, 与预期一致(图3-A, 表1)。dCAPS标记Rhg4-1165产生的PCR片段经Hin f I酶切后理论上产生3个酶切产物, 由于其中一个23 bp的片段长度过小, 电泳时迁移速度快, 跑出PAGE胶而无法检测, 因此, 只检测到2种酶切产物。未经酶切反应的DNA片段大小为219 bp, 等位变异为T; 酶切反应的DNA片段为196 bp, 等位变异为A (图3-B, 表1)。已鉴定9份种质在2个SNP位点上的鉴定结果与已报道一致, 表明开发的CAPS/dCAPS标记可用于大豆种质资源基因型鉴定。
利用开发的CAPS标记Rhg4-389和dCAPS标记Rhg4-1165对193份大豆抗感种质进行基因型鉴定, 发现2个位点上不同等位变异的发生频率接近。Rhg4-389-G等位变异的发生频率为54.5%, Rhg4- 389-C等位变异为45.6%。Rhg4-1165-T等位变异发生频率为53.4%, Rhg4-1165-A为46.6%。Rhg4-389-G等位变异在105份检出种质中抗病种质有99份, 对抗病种质的鉴定效率为94.3%; Rhg4-1165-T等位变异在103份检出种质中抗病种质有96份, 对抗病种质的鉴定效率为93.2%, 表明Rhg4-389-G和Rhg4- 1165-T等位变异主要分布于抗病种质(表3)。携带Rhg4-389-C和Rhg4-1165-A等位变异的种质在不同抗感种质中均有分布, 其中携带Rhg4-389-C等位变异的感病种质有19份, 中感种质有23份, 占关联分析群体感病种质(包括MS和S, 共43份)的97.7%; 携带Rhg4-1165-A等位变异的感病种质有19份, 中感种质22份, 占感病种质(包括MS和S, 共43份)的95.3%, 表明感病种质主要携带Rhg4-389-C和Rhg4-1165-A等位变异。
Rhg4-389和Rhg4-1165间的物理距离为776 bp, 存在显著连锁不平衡(P< 0.0001), r2为0.87, D° 为0.96。进一步分析发现, 193份大豆抗感资源在2个SNP位点中共形成4个单倍型, 其中前人已报道[5]的Rhg4-389-G/Rhg4-1165-T和Rhg4-389-C/Rhg4- 1165-A为优势单倍型, 发生频率分别为52.3%和44.6%; Rhg4-389-G/Rhg4-1165-A和Rhg4-389-C/ Rhg4-1165-T为新单倍型; 其中Rhg4-389-G/Rhg4- 1165-A发生频率仅2%, 携带该单倍型的种质有小黑豆、小黑豆、黑豆、PI438489B; Rhg4-389-C/Rhg4- 1165-T发生频率仅1%, 携带该单倍型的种质有黑豆和邳县大紫花糙(表4)。表型次数分布与单倍型经卡方独立性检测显示P< 0.0001 (P=1.49E-36), 说明单倍型与表型极显著相关。携带Rhg4-389-G/Rhg4- 1165-T单倍型(101份)的资源中表现抗病的种质有95份, 该单倍型对抗病种质的鉴定效率为94.1%, 由于2个SNP位点间存在显著的连锁不平衡, 抗病单倍型与单个抗病等位变异对抗病种质的鉴定效率相近; 携带Rhg4-389-C/Rhg4-1165-A单倍型的抗病(包括R和MR, 共45份)和感病(包括MS和S, 共41份)种质数目相近, 但对抗病种质的鉴定效率却比较低, 为30% (表4), 推测这些携带感病单倍型的抗病种质具有其他抗病基因。
 | 图3 2个CAPS/dCAPS标记在13份大豆种质中的多态性分析A: Rhg4-389标记在13份大豆种质的多态性; B: Rhg4-1165标记在13份大豆种质的多态性。M: DNA marker DL100; 1~13: PCR产物的酶切产物, 引物及内切酶信息见表1; 1: Williams 82; 2: Peking; 3: PI 90763; 4: Forrest; 5: Essex; 6: PI 437654; 7: 元钵黑豆; 8: PI 209332; 9: PI 548316 (Cloud); 10: 赤不流黑豆; 11: PI 88788; 12: 泰兴黑豆; 13: 抗线1号; S: 抗病种质; R: 感病种质。Fig. 3 Polymorphism analysis of two CAPS/dCAPS markers in 13 soybean cultivarsA: Polymorphism of CAPS marker Rhg4-389 in 13 soybean cultivars; B: Polymorphism of dCAPS marker Rhg4-1165 in 13 soybean cultivars. M: DNA molecular marker (100-bp ladder); 1-13: digested products of PCR amplicons; the primers and enzymes informations are shown in Table 1; 1: Williams 82; 2: Peking; 3: PI 90763; 4: Forrest; 5: Essex; 6: PI 437654; 7: Yuanboheidou; 8: PI 209332; 9: PI 548316 (Cloud); 10: Chibuliuheidou; 11: PI 88788; 12: Taixingheidou; 13: Kangxian 1; S: susceptible varieties; R: resistant varieties. |
| 表3 不同等位变异对应的抗病种质次数分布及选择效率 Table 3 Frequency distribution and selection efficiency of different alleles on resistant materials |
| 表4 不同等位变异单倍型对应的抗病种质次数分布及选择效率 Table 4 Frequency distribution and selection efficiency of different haplotypes on resistant materials |
近年来, 许多研究利用分子标记对大豆胞囊线虫抗性基因进行了定位和克隆, 筛选出一些与抗病基因紧密连锁标记, 主要是与rhg1位点连锁的标记[27]。Mudge等[28]在SCN抗性位点rhg1 两侧发现SSR标记Satt038与Satt130, 鉴定SCN的准确率分别为95%和74%, 用2个标记联合鉴定的准确率为98%, 但无法将PI 88788或PI 209332等抗源与美国南部的一些感病种质区分开。Cregan等[29]用和rhg1共分离的标记Satt309和Sat_168 (位于rhg1的两侧), 发现Satt309能够区分出Peking (PI 548402)、PI 90763、PI 437654和绝大多数的感病种质, Sat_168能够将PI 88788和PI 209332与美国南部的感病种质如
Essex、Lee、Bragg区分开来, 但无法将感病品系Hutcheson、Noir-1与抗源PI 88788、PI 209332区分开, 2个标记联合鉴定可显著增加对杂交后代的鉴定效率。王文辉等[30]也利用Satt309对我国634份大豆种质进行分子标记辅助鉴定, 发现128 bp和134 bp等位变异对抗病资源的鉴定效率为72.46%, 其中免疫、双抗、三抗种质的鉴定效率分别达到77.27%、87.69%和100%。Li等[31]对8份抗感种质(6份抗源和2份感病种质)的候选基因rhg1测序, 根据发现的SNP位点开发经济简便的SNAP标记, 通过关联分析发现由2个非同义突变SNP位点689C> A和757C> T形成的单倍型可有效的区分抗病和感病资源。南海洋等[32]同样以rhg1基因序列为基础开发InDel标记rhg1-I4, 对SCN抗病种质鉴定效率为88.2%, 感病种质鉴定效率为100.0%, 与Satt309联合鉴定可提高检测效率。但是目前还没有针对主效抗病位点Rhg4开发的可用于分子标记辅助选择的有效标记。Liu等[5]图位克隆了Rhg4基因, 并在该基因编码区上发现2个非同义突变SNP位点Rhg4-389 (G/C)和Rhg4-1165 (T/A), 通过28份抗感种质的关联分析推测这2个SNP位点可能与大豆胞囊线虫抗性相关, 因为分析群体数量过少, 有必要在大规模群体上进行验证。本研究针对Rhg4-389和Rhg4-1165分别开发了CAPS和dCAPS标记, 通过对193份大豆抗感资源的鉴定和分析, 确认Rhg4-389和Rhg4-1165与大豆胞囊线虫抗性密切相关, 其中Rhg4-389 (94.3%)对抗病种质的鉴定效率稍好于Rhg4-1165 (93.2%)。Rhg4-389和Rhg4-1165间处于高度连锁不平衡状态, 抗感种质在2个位点的基因型具有协同性, 形成的抗病单倍型对抗病种质的鉴定效率为94.1%, 说明Rhg4-389或Rhg4-1165单独使用即可辅助SCN抗性的鉴定。
Rhg1和Rhg4是控制大豆胞囊线虫病抗性的主效基因。研究表明, 大豆胞囊线虫抗源中存在PI 88788- Type和Peking-Type两种类型材料, PI 88788-Type抗病种质的抗性仅由rhg1控制, Peking-Type种质由rhg1和Rhg4两个抗性基因控制[5, 33, 34, 35]。在本研究供试的来自大豆胞囊线虫核心种质的抗源中, 90%以上携带Rhg4抗病等位基因, 抗源以Peking-Type类型为主, 加大PI 88788-Type抗病种质在我国抗病育种中的利用可有效拓宽抗病品种的遗传基础。此外, 在标记辅助选择实践中, 应该结合使用rhg1和Rhg4相关标记联合对种质进行鉴定, 阐明SCN抗性在这2个位点的遗传基础, 以促进抗源利用。
随着分子生物学研究发展与技术手段提高, 数以百万计的大豆SNP被发掘[36, 37, 38], 为大豆遗传育种和分子进化研究提供了丰富的标记资源, 高通量的SNP基因分型技术(如Illumina公司的GoldenGate等)及应运而生的基因特异性SNP鉴定技术(如KASPar[39]、CAPS标记、SNAP[40]等)的使用大大促进了标记资源的利用。与自动化程度较高、对少量标记大群体基因型鉴定更经济有效的KASPar技术[41]相比, 本研究开发的CAPS和dCAPS标记, 不需要荧光实时定量PCR仪等设备, 利用琼脂糖和聚丙烯酰胺凝胶电泳就可以检测, 花费较低, 方便我国育种家利用。
针对大豆胞囊线虫主效基因Rhg4上的2个SNP位点, 开发了简便、经济的CAPS标记(Rhg4-389)和dCAPS标记(Rhg4-1165), 单标记抗病等位变异Rhg4-389-G (94.3%)和Rhg4-1165-T (93.2%)及它们组成的抗病单倍型(94.1%)对抗病种质的鉴定效率相近, 说明2个标记具有协同性, 皆可用于辅助SCN抗性鉴定。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|