第一作者联系方式: E-mail:hongxiaoxd@163.com
株型是决定水稻等作物产量的核心因素之一, 是品种选育的重要指标。本研究从粳稻品种Asominori的辐射诱变中分离出2个稳定的株型突变体, rad-1和 rad-2, 它们均表现出苗期弯曲生长、成熟期株高矮化、粒长变短、千粒重降低和产量下降等特征。等位性测验结合连锁分析证实 rad-1和 rad-2等位, 且位于水稻第7染色体上约230 kb的范围内。对定位区段的序列分析后确定 OsFH5为候选基因, 该基因呈现组成型表达, 编码水稻II型成蛋白。突变体 rad-1在 OsFH5基因第2个外显子上缺失8个碱基, 导致移码; 而 rad-2则在第14内含子上发生单碱基的变异, 发生异常剪切。两类突变最终均导致 OsFH5基因翻译提前终止, 产生截短的蛋白。相比野生型, rad-1和 rad-2在幼苗中 OsFH5基因的表达下调。细胞学研究表明, OsFH5基因的功能缺失会导致幼苗叶鞘细胞大小不均, 呈现不规则生长, 在成熟的颖壳中细胞显著变短。对生长素响应的ARF因子进行表达量检测发现, rad-2中一系列ARF成员表达均显著下调, 推测 OsFH5极有可能影响了植株对生长素的响应。
Plant architecture is a key element of crop yield and also taken as a criterion in breeding. In this study, two rice architecture determinant mutants, rad-1 and rad-2 were selected from Gamma-radiation induced mutants of rice cultivar Asominori. Each of them shows winding seedlings, dwarf plants and thereby decreased yield. It has been estimated that rad-1 and rad-2 are allelic to each other by allelism test and linkage analysis. After map-based cloning, the responsible gene for the mutations was restricted within a 230 kb region in chromosome 7. Sequence analysis revealed that OsFH5 gene, encoding a forming-like protein altered both in rad-1 and rad-2. An 8-bp deletion in exon 2 in rad-1 and a single nucleotide change in intron 14 in rad-2 made frame-shift reading and produced truncated proteins. OsFH5 gene exhibited constitutive expression in tested tissues. Compared with the wild type, prominent decline of OsFH5gene was observed in each mutant. In seedlings, loss-function of OsFH5 gene resulted in irregular cell growth in leaf sheath and shorter cells in inner husk, which therefore caused short grain and lower grain weight. By quantitative RT-PCR analysis, several auxin response factors were dramatically down-regulated in rad-2, which suggested that OsFH5 may affect the response to auxin in rice.
水稻是重要的粮食作物, 也是研究单子叶植物遗传和发育的模式植物。株型是影响水稻产量的一个重要指标, 对于生产具有重要的意义。20世纪60年代, 绿色革命的兴起实现了水稻产量的成倍增长, 这主要是通过培育半矮秆水稻品种以提高水稻抗倒伏性来实现的。水稻株型对于产量的影响还不仅仅取决于株高。20世纪80年代起, 育种家们相继提出了理想新株型的概念, 他们认为理想株型应满足耐肥、抗倒伏、生物量大且经济系数适宜的要求, 具体表现为根系发达、茎秆粗壮、穗粒数多且没有无效分蘖等。理想株型的提出为高产育种指明了方向, 引起了各国育种工作者的广泛重视, 相应的新株型育种计划得以开展。然而, 水稻理想株型的育种工作仍未达到预期目的, 这归根结底还是因为株型特征决定于遗传基础, 一个看似简单的表型往往由多种复杂的遗传机制共同调控, 因此, 只有深入了解株型形成的分子机制, 才能加快株型的改良。到目前为止, 关于株型相关基因的报道正逐步揭示株型形成的内在机制。
已克隆的株型相关基因主要集中在株高、分蘖、穗形等方面。Sd1是株高相关的基因, 它编码赤霉素(gibberellin, GA)合成途径中的关键酶GA20氧化酶(GA20ox)[1, 2]。该基因位点的不同突变均导致植株体内赤霉素含量降低, 引起植株半矮化。除Sd1外, 通过对一系列矮化及矮化多分蘖的突变体进行图位克隆, 发现了同样参与赤霉素途径的其他基因, 其中d35参与赤霉素的合成[3], 而d1、euil、gid1、gid2参与赤霉素的信号传导[4, 5, 6, 7]。关于赤霉素如何影响株高至今仍有许多没有解开的谜团, 而其他激素的加盟则让株高的调控机制变得更加错综复杂。d2、d11、brd1、brd2等基因被报道通过影响油菜素内酯(brassinosteroid, BR)的生物合成而导致矮化, 而d61则影响植株对BR的响应[8, 9, 10, 11, 12, 13]。另一类新型的激素独脚金内酯(Strigolactone, SL)也被报道作用于生长素的下游, 抑制腋芽的生长, 并调控株高的变化[14], 其中已克隆的d10、d17/htd1和d27参与SL的生物合成, 而d14和最新报道的d53参与了SL的信号转导, 这些基因的突变都会引起矮化多分蘖的表型[15, 16, 17, 18, 19]。
理想株型的另一方面重要研究就是分蘖, 同样通过突变体的遗传研究来了解分蘖调控的基因网络。如编码GRAS类型转录因子的MOC1基因, 过量表达MOC1能够引起分蘖能力的增加[20]。DLT同样编码GRAS类转录因子, 它通过BR的信号转导来影响分蘖, dlt突变体呈现出半矮秆、分蘖减少等表型[21]。Takeda等[22]对水稻中的OsTB1进行了研究, 该基因编码一个bHLH转录因子, 过表达后能极大地减少侧枝而不影响分蘖芽的数目, 说明该基因负调控侧枝的生长。在moc1突变体中, OsTB1表达量显著减弱, 说明OsTB1可能作用于MOC1的下游。2010年, Jiao等[23]利用一个少分蘖的突变体克隆了理想株型基因IPA1, 该基因不仅影响水稻的抗倒伏性和产量, 同时也抑制水稻的分蘖。除分蘖的数目外, 分蘖的角度也被认为影响着产量, TAC1和OsLIC1被报道影响着分蘖的角度, 其中TAC1的上调表达能够导致分蘖角度的增大[24], 而OsLIC1的表达则认为是受BR调控[25]。
相比于株高和分蘖, 穗粒数的多少更能直观地反映产量的高低。LAX1与SPA被报道共同调控腋芽的起始, 两者突变后植株既不能形成小穗枝梗, 也没有分蘖芽的形成[26]。LAX2与LAX1突变体的表型相似, 且LAX1与LAX2编码的蛋白能够形成二聚体。在穗畸形的突变体apo1中, 一次枝梗明显减少, 穗子缩短, 且穗粒数也明显减少。过表达APO1可同时增加一次枝梗数、二次枝梗数和穗粒数[27]。另一个重要的调控穗粒数的QTL Gn1a被克隆, Gn1a编码细胞分裂素氧化/脱氢酶OsCKX2, 能够降低植株体内细胞分裂素的含量。当Gn1a基因表达降低时, 细胞分裂素积累, 并促进花序分生组织的分化能力, 从而增加穗粒数[28]。
株型基因还包括了一些基本形态决定因子, 例如影响微丝聚合的成蛋白对株型起重要的决定作用, 而最新的研究报道认为基本形态决定因子通过影响生长素的极性运输来调控株型[29, 30]。总之, 植物株型建成是一个相当复杂的过程, 而目前已知的调控网络仍不够清晰, 进一步克隆基因并展开功能研究是促进机制研究的主要方法之一。本研究筛选到2个稳定的株型突变体(rice architecture determinant, rad-1和rad-2), 并对其进行图位克隆, 最终验证OsFH5基因是影响株型的关键因子。不同株型突变体的鉴定对于后续基因功能的研究具有重要的意义。
突变体rad-1和rad-2是粳稻品种Asominori经钴60辐射诱变后分离出来的, 经过多代自交后性状稳定遗传。将rad-1和rad-2与籼稻品种南京11杂交获得F1代, F1自交获得F2代分离群体用于定位研究。所有材料均于正季种植于江苏省南京市江宁区土桥镇南京农业大学试验基地, 种植和管理方法同常规大田。
将亲本及F2群体浸种催芽后均匀撒播于秧田, 8~10 d左右观察自然条件下野生型与突变体的表型差异, 并从F2群体中筛选极端突变表型个体, 用于基因的定位。成熟期, 各取10株突变体和野生型考察株高、分蘖数、千粒重等重要农艺性状, 其中株高、剑叶长宽、各节间长度、每穗粒数等均以主茎为考察对象。
利用水稻数据库(http://www.gramene.org/)已公布的576对SSR引物对Asominori和南京11进行筛选, 获得150对在2个亲本之间存在多态的分子标记, 分布于12条染色体上, 用于基因的初定位。根据籼稻9311与粳稻日本晴序列之间的差异, 利用Primer5.0软件设计引物, 开发SSR、Indel等标记, 用于基因的精细定位。引物设计的要求为18~25个碱基、GC含量50%~60%、避免特殊结构、扩增特异性强, 具体引物信息如表1。
| 表1 本实验所用引物列表 Table 1 List of primers used in experiment |
采集新鲜的叶片, 于液氮中碾磨至粉末状, 用SDS法提取基因组DNA, 溶解在适量ddH2O中, 用作PCR反应的模板。10 μ L反应体系含: 10× PCR缓冲液1 μ L, dNTPs (2 mmol L-1) 1 μ L, 正、反引物(2 μ mol L-1)各1 μ L, DNA 2 μ L (20~50 ng), Taq DNA聚合酶(2 U μ L-1) 0.2 μ L, ddH2O 3.8 μ L。PCR程序为: 95℃预变性5 min; 94℃变性30 s, 55~60℃退火30 s, 72℃延伸40 s, 34个循环; 72℃再延伸5 min。PCR产物经8%聚丙烯酰胺凝胶电泳检测及0.1% AgNO3溶液银染显色。
选取平均覆盖每条染色体的多态性分子标记, 对F2分离群体中具有极端表型的单株进行基因型鉴定, 寻找与突变表型相连锁的分子标记。同时, 根据极端个体中交换重组发生的位置, 确定基因的候选区段。在此基础上, 扩大F2分离群体, 进一步筛选表型明确的极端个体, 利用候选区段内加密的分子标记进行基因型鉴定。综合基因型与表型结果逐步缩小基因的范围。
根据精细定位的结果, 利用Gramene数据库(http://www.gramene.org/)对定位区间进行基因分析。选取与水稻生长发育相关的基因作为候选基因。设计基因特异性引物, 用高保真酶KOD (东洋纺)扩增候选基因的基因组及cDNA序列, 将PCR产物回收纯化后克隆到pEasy-Blunt (TransGen)载体, 挑选阳性克隆送至南京金斯瑞公司测序, 比较野生型与突变体中候选基因的序列差异。采用DNAMAN软件预测编码氨基酸的序列并比对。
选取苗期的叶鞘及成熟期的外颖组织, 在叶鞘外层及外颖内层轻轻涂上透明的指甲油, 待自然晾干后, 将印有细胞形状的薄膜揭下并粘于载玻片上, 光学显微镜下观察比较细胞的形状和大小。
采用植物总RNA提取试剂盒(TIANGEN)提取所有组织样品的RNA。以总RNA为模板, Oligo dT为引物反转录成cDNA。反转录所用试剂均购买至TaKaRa公司。
分别采集7 d和10 d野生型幼苗、幼根和成熟期的根、茎、叶、幼穗等部位, 提取RNA, 采用荧光定量的方法检测基因的表达模式。实时荧光定量PCR体系含: cDNA (10 μ g mL-1) 2 μ L、RNA free H2O 9.3 μ L、primer (5 μ mol mL-1) 1.2 μ L、Mix (Bio-Rad) 12.5 μ L, 共25 μ L。扩增程序为50℃ 2 min, 95℃ 10 min, (95℃ 15 s, 60℃ 1 min) 40个循环, 95℃ 15 s, 60℃ 1 min, 95℃ 15 s, 60℃ 15 s。以Actin作为内参基因, 按照2-Δ Δ CT计算基因在各组织样品中的相对表达量。
此外, 选取7 d野生型和突变体幼苗的cDNA样本, 对水稻中目前预测的25个ARF生长素响应基因进行定量, 分析基因突变对生长素响应的影响。ARF1~25对应的基因编号分别为Os01g0236300、Os01g0670800、Os01g0753500、Os01g0927600、Os02g0141100、Os02g0164900、Os02g0557200、Os02g0628600、Os04g0442000、Os04g0519700、Os04g0664400、Os04g0671900、Os04g0690600、Os05g0515400、Os05g0563400、Os06g0196700、Os06g0677800、Os06g0685700、Os06g0702600、Os07g0183100、Os08g0520500、Os10g0479900、Os11g0523800、Os12g0479400、Os12g0613700。 参考前人报道[31]的基因定量的引物, 其中对ARF9、10、11、12、13的引物做了更正, 详见表1。
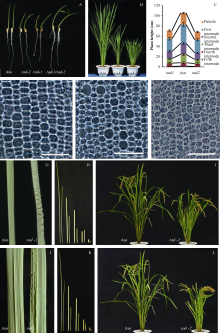
突变体rad-1和rad-2在萌发后10 d左右均表现出明显的弯曲生长, 且幼苗较野生型生长减缓, 并有明显的矮化现象(图1-A)。不仅如此, 幼苗的根部同样出现轻微波浪状弯曲(图1-A), 这说明突变对整个植株的形态都产生了明显的影响。随着幼苗的持续生长, 突变体的弯曲表型逐步减弱, 至分蘖期基本直立, 但株高矮化现象仍然明显, 且叶片呈现不同程度的褶皱(图1-B, G, J)。
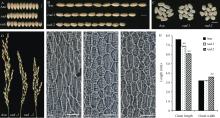
对成熟期株高测量发现, 野生型达(103.1± 3.2) cm, 而突变体rad-1和rad-2则分别为(68.0± 3.3) cm和(67.7± 2.1) cm, 呈现明显的半矮化(图1-I, L, 表2)。取植株的不同节间观察比较, 其中第一、二、三、五节间显著缩短, 而第四节间则没有差异(图1-C, H, K)。比较剑叶形态后发现, 剑叶宽无变化, 但长度显著下降(表2)。此外, 突变体rad-1和rad-2比野生型穗子短小, 其一次枝梗数、二次枝梗数和每穗粒数均显著下降(图2-D, 表2)。
尽管2个突变体植株形态较相近, 但在粒型上却存在明显差异。野生型粒长为(7.593± 0.024) mm, rad-1的粒长为(6.879± 0.027) mm, 而rad-2的粒长则更短, 为(6.081± 0.019) mm; rad-1与野生型的粒宽并无差异, 而rad-2较两者粒宽明显增加, 且糙米表面有褶皱, 并伴腹白表型(图2-A, B, C, H; 表2)。粒长的显著下降使得两个突变体的千粒重显著下降, 这足以说明株型的改变严重影响产量。
| 表2 野生型与突变体rad-1和rad-2农艺性状统计表 Table 2 Statistics data of agronomic traits in wild type and rad-1andrad-2 |
对幼苗期叶鞘的细胞学观测发现, 野生型幼苗的叶鞘细胞排列较规则, 大小相近, 而突变体中, 幼苗叶鞘表皮的细胞大小差异明显, 排列不规则, 说明苗期茎秆的扭曲可能是由于细胞形态变化造成的(图1-D, E, F)。此外, 对成熟期颖壳的内层细胞观察发现, 突变体颖壳细胞的长度明显小于野生型, 说明细胞长度缩短是突变体粒长减小的原因(图2-E, F, G)。
由于rad-1和rad-2在表型上相似, 将两者杂交, 并鉴定杂种F1的表型。rad-1/rad-2在苗期仍然表现出明显的茎秆弯曲表型(图1-A), 说明rad-1和rad-2互为等位突变。
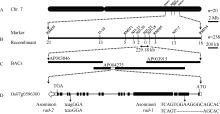
为了对突变的基因位点进行定位, 首先从rad-1/NJ11和rad-2/NJ11的F2群体中筛选出20个具有极端突变表型的单株, 利用覆盖12条染色体的多态性标记进行基因型鉴定。在第7染色体的分子标记RM351和RM505处观察到明显的连锁趋势。其中9个单株在分子标记RM455和RM351之间发生了交换, 将基因限定在RM455下游; 而其中4个单株在分子标记RM505和RM234之间发生交换, 将基因限定在RM234的上游。RM455和RM234标记之间的遗传距离为12.49 cM。为进一步精细定位基因, 对F2群体中218个极端个体进行基因型鉴定, 又获得24个在候选区段内存在交换的单株。利用加密的分子标记, 对37个交换单株进行鉴定, 发现2个单株在NJ7-29和NJ7-23之间发生交换, 将基因限定在NJ7-29下游, 而另一个单株则在NJ7-23和NJ7-18之间发生交换, 将基因限定在NJ7-18标记的上游。NJ7-29和NJ7-18之间的物理距离为229.18 kb (图3-A, B)。所有极端表型的交换单株在成熟期均表现出株高的矮化, 并且对其F2:3种子进行苗期鉴定, 均呈现稳定的茎秆弯曲, 这更明确了基因定位结果。
登陆Gramene数据库, 对定位区间内已预测的基因进行分析, 发现这一区域内存在一个编码植物II型成蛋白的基因OsFH5, 曾被报道参与微丝的聚合, 突变后引起苗期茎秆弯曲等相似表型[29], 因此我们将OsFH5作为候选基因。OsFH5基因含有19个外显子和18个内含子, cDNA全长5568 bp, 其编码的OsFH5蛋白含1627个氨基酸, 包括3个结构域, PTEN-like、FH1和FH2 (图4)。我们设计15对引物覆盖OsFH5的基因区域, 包括3 kb启动子区和10.48 kb编码区。对野生型Asominori和突变体rad-1和rad-2分别进行扩增、测序并拼接完整。对拼接序列分析显示, 与野生型相比, rad-1和rad-2在启动子区均无序列变异。而rad-1在ATG下游第1009个碱基处有8 bp的缺失, 该缺失位于第2外显子上, 直接导致突变基因移码, 编码的蛋白质在第124个氨基酸处提前终止, 3个结构域均缺失(图4)。在rad-2中, OsFH5基因在第14个内含子的最后一个碱基发生了G到A的替换。对rad-2中OsFH5基因所编码的cDNA克隆测序后发现, 内含子上G→ A的变化导致剪切方式的改变, 编码的氨基酸发生移码, 并在第1534个氨基酸处提前终止(图4), 该突变会造成FH2结构域缺失, 而PTEN-like和FH1结构域仍保持完整。根据以上结果推测, OsFH5基因的功能缺失是造成rad-1和rad-2突变体表型的原因。
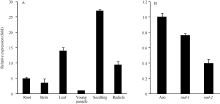
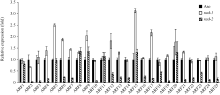
利用Real-time RT-PCR检测野生型的根、茎、叶、穗、幼苗和幼根中OsFH5基因的表达量发现, 该基因在各个组织中呈现组成型表达, 其中在幼苗中表达量最高。比较野生型与突变体rad-1和rad-2中OsFH5基因在幼苗期的表达量, 发现突变体中该基因的表达量都有下调, 这可能与该基因功能的缺失有关(图5-A, B)。
由于OsFH5基因功能的缺失, 观察到突变体在细胞生长水平中呈现出缺陷, 包括细胞长度减小, 细胞形态改变。在幼苗期, 突变体茎秆细胞出现不规则生长, Zhang等[30]研究猜测可能与细胞水平上激素的极性运输有关。为了证实这一想法, 我们比较了野生型与突变体中生长素和IAA响应基因的表达水平。有意思的是, 我们发现在突变体rad-2中, 生长素影响因子ARF2、ARF3、ARF10、ARF19等一系列基因均发生显著的下调, 而在rad-1中则无下调表达的趋势。
我们对株型突变体rad-1和rad-2与野生型的表型详细调查比较发现, rad-1和rad-2不仅能够在幼苗期造成茎秆的扭曲生长, 同时在成熟期较野生型株高明显降低, 一次枝梗数、二次枝梗数和每穗粒数减少, 并出现粒长的缩短, 说明突变的基因对于水稻株型的形成起决定性作用。通过突变体材料的图位克隆发现, 在rad-1和rad-2中, 编码植物II型成蛋白的OsFH5基因均发生了不同程度的突变, 造成结构域的缺失, 进而直接影响基因的功能。等位性测验也证实rad-1和rad-2为等位突变。据此, 我们证实OsFH5基因的变异是引起表型变化的内在原因。该基因呈现组成型表达, 而在突变体中表达量下调。以上结果说明OsFH5基因是株型的决定因子, 影响水稻生长的各个阶段, 并最终影响产量。
比较rad-1和rad-2的表型发现, 2个突变体在成熟期的表型并不完全一致, 主要表现在粒宽的差异, rad-2较野生型和rad-1籽粒更宽。OsFH5基因编码的是植物II型成蛋白, 包括PTEN-like结构域, 一个FH1结构域和FH2结构域, 其中PTEN-like结构域主要行使结合到叶绿体上的功能, 而后两者作用于微丝的聚合。在rad-1中, 突变导致翻译提前终止, 产生仅存124个氨基酸的截短蛋白, 即3个结构域都缺失, 蛋白完全丧失功能; 然而, 在rad-2中, 突变造成FH2结构域部分缺失, 由此产生的蛋白仍保留了完整的PTEN-like和FH1结构域, 这样的蛋白在体内或者仍能实现部分的功能, 或者以假基因的形式造成dominant-negative的效应, 干扰其他同源基因的功能。在已报道的等位突变体rmd/bui1中出现了穗轴弯曲的表型, 这在rad突变体中并没有明显的体现, 可能是遗传背景不同所致[29]。虽然以上突变体中突变的基因都相同, 但不同等位变异最终引起的表型却存在差异, 这其中的真正机制仍然未知。对于不同等位基因效应的深入研究可有助于进一步探讨各结构域的功能及基因与基因之间的相互作用。
通过对野生型和突变体苗期叶鞘和成熟期颖壳的细胞学观察发现, rad-1和rad-2苗期株高的弯曲和矮化主要是细胞呈现不规则而导致的。OsFH5蛋白具备在体外促使微丝、微管成束, 促进G-肌动蛋白成核和合成并对微丝进行封端等功能。拟南芥中一些微管蛋白的突变体, 包括tortifolia2、wave- dampened 2和spiral1等, 都曾被报道导致植株的扭曲, 其本质是微管、微丝走向的改变影响了细胞壁微纤维丝的排列, 最终改变了细胞的形状[32, 33, 34]。对rad突变体的细胞学观察发现, 突变体的细胞并没有发生扭曲, 说明茎秆弯曲并不是影响微管微丝的走向而引起。
Zhang等[29]对于rmd的研究指出, OsFH5突变后会影响生长素运输蛋白PIN的定位, 进而影响生长素的极性运输, 限制细胞的生长。我们的研究也发现rad-2中一系列ARF基因表达下调, 包括ARF2、ARF3、ARF10和ARF19等, 这一结果进一步证实了前人的报道。但在rad-1中并没有观察到ARF基因的表达量变化, 说明OsFH5基因不同类型的突变造成的影响是不同的。而OsFH5究竟如何调控生长素的极性运输还不得而知, 有待进一步研究。
rad-1和rad-2为2个等位的株型突变体, 候选基因定位在第7染色体上约230 kb的范围内, 其中OsFH5基因被证实在rad-1和rad-2中均发生突变, 导致蛋白截短。OsFH5基因呈组成型表达。OsFH5基因突变会导致茎秆细胞生长不规则, 颖壳内层细胞缩小, 引起苗期茎秆扭曲, 成熟期籽粒缩短, 产量下降。OsFH5基因可能影响植株对生长素的响应。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|