第一作者联系方式: E-mail:jing287346@163.com, Tel: 0311-87670626
重组型大豆花叶病毒(recombined soybean mosaic virus, SMV-R)是一种新SMV类型, 在我国多个大豆产区广泛流行。本研究对一个重组型SMV河北分离物(HB-RS)进行全基因组测序,比较与非重组型SMV在侵染4个大豆品种后病毒浓度积累的差异。结果显示, 除poly-A尾巴外, HB-RS (NCBI登录号为KR065437)由9993个核苷酸组成, 包含一个开放阅读框(open reading frame, ORF), 翻译后形成3202个氨基酸, 系统进化分析结果显示HB-RS分离物与另外两个重组型SMV分离物聚在一组。抗性鉴定结果显示, 4个品种对HB-RS和Sc6平均病情指数分别为59.5和60.5, 相同大豆品种对不同的株系(分离物)可能呈现不同的症状和抗性表现, 其中冀豆17对Sc6和HB-RS分别表现高抗和中抗, 表明大豆对SMV的抗性存在一定的株系(分离物)专化性。此外, HB-RS在4个品种中的浓度积累均高于Sc6, 在南农1138-2病毒浓度最高, 达522 U, 其次为五星1号(471 U)和冀黄13 (199 U), 最低为冀豆17, 仅90 U。说明HB-RS在寄主体内更具有生存适应性, 不同品种对SMV存在抗性差异。冀豆17可作为抗性品种和亲本进一步推广。
Recombined Soybean mosaic virus (SMV-R), which is prevalent in many soybean production regions, is a novel type of SMV. To clarify the characteristics of recombinant SMV structure and pathogenicity of SMV-R, we sequenced the whole genomes of a SMV-R isolate (HB-RS) from Hebei province of China, and compered the difference of SMV infection and accumulation in four soybean cultivars with SMV-R. The results showed that besides the poly-A tail, HB-RS consists of 9993 nucleotides, encoding only one open reading frame and 3202 amino acids. Phylogenetic analysis showed that the HB-RS isolate was clustered with other two recombined SMV isolates. Resistance identification results showed that the average disease index of four cultivars resistant to HB-RS and Sc6 was 59.5 and 60.5, respectively. The same soybean cultivar had different symptoms and resistance levels to different strains (isolates), Jidou 17 showed high resistance to Sc6 and moderate resistance to HB-RS. These results indicated that soybean resistance to SMV exists strain (isolate) specialization. Additionally, the pathogenicity test showed that accumulation of HB-RS in four cultivars was higher than that of Sc6, indicating that HB-RS is more adaptable to the host plants. The host with highest accumulation of HB-RS was Nannong 1138-2, with 522-fold of reference virus accumulation, and the following was Wuxing 1 (471 U), Jihuang 13 (199 U), and Jidou 17 (only 90 U), suggesting that HB-RS has a more survival adaptability in the soybean host. However different resistance levels to HB-RS were observed in various soybean cultivars and Jidou 17 could be used as a resistant cultivar in production or parents in further breeding.
大豆花叶病毒(soybean mosaic virus, SMV)病是全球大豆产区分布最广的病毒病害之一, 在我国各大豆产区均有发生, 严重影响大豆的产量与品质。目前, 针对SMV病害没有有效的化学防控方法, 主要以抗病育种为主。
SMV是单链正义RNA病毒, 属于Potyvirus属成员, 该属是已知植物病毒中最大的属, 占总数的20%以上[1, 2]。SMV全长约9600个核苷酸(nt), N端有一共价键连接的金属蛋白Vpg, C端有一个Poly(A)尾巴, 整个基因组只有一个开放阅读框架(open reading frame, ORF), 翻译后产生一个多聚蛋白前体[3], 随后被病毒基因组编码的3种蛋白酶(P1, HC-Pro, NIa-Pro)切割加工, 形成10个不同功能的成熟蛋白, 从5° 到3° 依次为P1、HC-Pro、P3、6K1、CI、6K2、NIa-VPg、NIa-Pro、NIb和CP。此外, 在P3编码区内部由于移码翻译形成一个与P3融合的P3N-PIPO蛋白[4]。
重组进化分析发现重组病毒在Potyvirus属中非常常见, 尤其P1编码区是发生重组的热点区域[4, 5]。SMV也易与其他种类病毒复合侵染发生重组, 形成新的病毒类型。例如分析半夏上分离出的SMV-P分离物全序列, 推测它可能是由SMV与芋花叶病毒(dasheen mosaic virus, DsMV)在P1区域重组而来, 但在寄主范围上与SMV有较大差异[6]。通过比较西瓜花叶病毒(watermelon mosaic virus, WMV) Pk分离物与其他相近病毒分离物在P1蛋白的N-和C-, HC-Pro, 以及5° UTR区域后分析认为WMV-PK分离物是由SMV和花生条纹病毒(peanut strip virus, PStV)或菜豆普通花叶病(bean common mosaic virus, BCMV)在P1区域重组而来[7]。此外, 类似重组事件在BCMV和WMV等病毒中也有报道[8, 9, 10, 11, 12], 重组后的病毒在其寄主的范围上或致病力上均有改变。
最先从重庆获得的SMV分离物中发现并鉴定了重组型SMV[13], 该病毒是由SMV和BCMV在P1编码区重组而来, 在西南、东南沿海及黄淮北部地区均有广泛分布[14]。重组型SMV是在大豆上被发现的, 对我国大豆生产存在潜在危害, 到目前为止对重组型SMV的抗源筛选工作和侵染特性研究还未开展, 出现我国大豆抗病育种工作的盲区。
本研究在发现重组型SMV的基础上, 对来源于河北省大豆主产区的一个重组型SMV进行全基因组测序及其结构分析, 明确其在系统进化中的分类地位; 利用4个大豆品种对2个不同类型SMV分离物进行抗性鉴定, 观察病毒在大豆植株中的积累, 明确重组型SMV潜在的危害, 为抗病育种提供借鉴。
所用SMV重组型分离物采自河北省中部石家庄大豆产区, 试验前期在田间采集具有典型花叶症状的大豆叶片, 用锡箔纸包好于液氮中速冻, -80℃冰箱中储存备用。将病毒分离物接种至大豆品种南农1138-2, 繁殖并保存。利用之前报道的引物DeF: 5′ -AAATTAAAACWACTCATAAAGACAA-3′ 和DeR: 5′ -ATCGCTCATCAAACAGTAAACC-3′ 确认病毒类型[13]。对其中一个确认的重组型SMV分离物进行全基因组测序, 并命名为“ 河北重组型SMV分离物(HB-RS)” 。
用于病毒积累分析的4个品种分别是南农1138-2 (感病对照)、冀豆17、冀黄13和五星1号, 其中南农1138-2由南京农业大学智海剑教授提供, 其余3个品种由本研究所提供。
利用植物RNA提取试剂盒(DP419, 天根)提取含有病毒叶片的总RNA, 利用反转录试剂盒AMV3.0 (DRR019A, TaKaRa, 日本)按说明书合成cDNA。由北京六合华大公司合成扩增引物(表1)[13]并测序。每个克隆保证6个以上, 以确保序列的准确性。参照Yang等[13]方法制备病毒接种液及接种大豆植株, 接种后用缓冲液冲洗, 并取其中一片真叶以液氮冷冻备用, 大约14 d后待对照品种南农1138-2完全发病后, 取同时期同部位4个品种的上位叶片, 利用荧光定量方法监测, 使用的试剂为SYBR Premix ExTaq II (DRR820A, TaKaRa), 仪器为Bio-Rad IQ5, PCR反应体系和反应程序均按说明书操作。
| 表1 本研究中所用到的引物 Table 1 Primers used in the study |
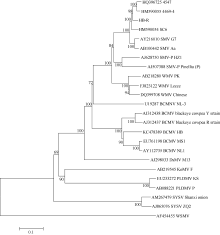
试验中用到的22条与SMV亲缘关系较近的全序列分别为BCMV分离物blackeye cowpea Y strain (AJ312438)、blackeye cowpea R strain (AJ312437)、MS1 (EU761198)、NL1 (AY112735)和HB (KC478389); BCMNV分离物 NL-3 (U19287); SMV分离物Aa (AB100442)、G7 (AY216010)、4547 (HQ396725)、4469-4 (HM590055)和Sc6 (HM590054); SMV半夏分离物Pinellia (AJ507388)和HZ1 (AJ628750); DsMV分离物M13 (AJ298033); WMV分离物Pk (AB218280)、Lecce (FJ823122)和Chinese (DQ399708); PLDMV分离物KS (EU233272); SYSV分离物Shanxi onion (AM267479); KoMV分离物F (AB219545); PLDMV分离物P (AB088221), 以及聚类分析中用于外组成员的病毒分离物WSMV (AF454455)。以上序列均从http://www.dpvweb.net/notes/families.php网站上获得。对测序获得的重组型SMV序列HB-RS利用SeqMan软件(DNAStar, Inc.)拼接, 并对每个差异碱基位点进行人工校正。利用DNAMAN[15]比对分析病毒序列相似性。利用MEGA4.1软件中的Neighbour-Joining方法构建系统发育进化树, 用UPGMA[16]和MP[17]两种方法同时验证结果, BootStrap值为1000; 隐藏可信度小于50%的值。
主要调查包括发病率、症状类型、病级, 在此基础上计算病情指数。参照智海剑等[19]的单株病级标准。其中, 坏死症状对大豆生产有较大影响, 因此在本研究中作为感病症状。品种抗性按如下标准分类, 即病情指数0为免疫材料(SY); 1~20为高抗(HR); 21~40为中抗(MR); 41~60为中感(MS); 61~80为感病(S); > 80为高感(HS)。

每个样本和内部参照均用3个重复反应孔扩增。通过比较Ct的方法进行基因表达水平的相对定量分析, Δ Ct是看家基因的归一化, 目的是消除样品间模板量的差异, Δ Δ Ct是基准品的归一化(如正常的样品VS处理样品中, 正常样品即为基准品), 用Δ Δ CT = (CTTarget- CTtubulin) Timex-(CTTarget- CTtubulin) Time0计算分析数据, 用SigmaPlot处理显示。
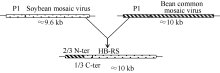
重组型SMV分离物的全序列克隆测序拼接后提交NCBI的GenBank数据库, 登录号为KR065437。除poly-A尾巴外, HB-RS由9993个核苷酸组成, 将全序列提交NCBI进行Blast显示, 与之前报道的SMV分离物4469-4和4547同源性最高, 分别为95%和94% (数据未显示)。与相近病毒种序列相比, HB-RS分离物的5° UTR和2/3 N-P1编码区与BCMV同源性最高(> 85%), 其余编码区及3° UTR区域均与SMV有极高的核苷酸同源性(> 96%), 因此推测HB-RS分离物是由SMV和BCMV在P1编码区重组而来的(图1)。HB-RS包含一个开放阅读框(open reading frame, ORF), 从第132个核苷酸位置开始翻译, 根据序列推测翻译后形成3202个氨基酸, 与以往公布重组型SMV分离物一致, 但比非重组型SMV分离物多出135个或136个氨基酸。HB-RS翻译后形成10个蛋白的预测酶切位点与重组型SMV分离物4469-4及非重组型SMV分离物Sc6均完全一致(表2)。
| 表2 HB-RS多聚蛋白切割位点的预测和比较 Table 2 Predicted and comparison of polyprotein cleavage sites for HB-RS |
图2显示, HB-RS分离物与SMV大豆上的分离物聚在一组, 而SMV半夏上的分离物聚在相对较远的一组, BCMV的blackeye cowpea来源的分离物也与其他来源的分离物各自聚在一组, 表明不同寄主物种来源的病毒存在一定的分化性, 但与其他近缘种病毒相比亲缘关系仍最近。HB-RS除与重组型SMV分离物4547和4469-4亲缘最近外, 相比较国外鉴定的SMV分离物G7和Aa, 国内来源的分离物Sc6与HB-RS也显示出较近亲缘关系, 这可能跟来源地相近有关。WMV聚类于SMV和BCMV两组之间, 表现出其自SMV和BCMV衍生的特性, 亲缘上与SMV更接近, 表明在重组进化中来源于SMV的血缘更多一些。
选取4个大豆品种对2个SMV分离物进行抗性鉴定, 结果显示4个品种接种HB-RS后整株症状均以花叶为主, 接种叶无症状(表1)。其中, 除冀豆17对HB-RS表现中抗外, 其余3个品种均为感病。南农1138-2和五星1号对Sc6表现高感, 植株出现严重的花叶、黄化和坏死等症状, 接种叶上出现局部坏死斑。但冀豆17和冀黄13对Sc6分别表现为高抗和中抗, 整株症状以轻花叶为主, 接种叶上无明显坏死斑点。4个品种对HB-RS和Sc6平均病情指数接近, 分别为59.5和60.5, 4个品种对HB-RS病情指数比较接近, 对Sc6呈现两极分化。研究还显示, 相同大豆品种对不同的株系(分离物)可能呈现不同的症状反应和抗性表现, 表明大豆对SMV的抗性存在一定的株系(分离物)专化性。
| 表3 抗性鉴定结果 Table 3 Resistance identification results |
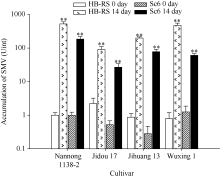
选取上述4个大豆品种接种HB-RS和Sc6进行病毒浓度积累对比分析。分别以接种HB-RS和Sc6后0时间点取样叶片的含毒量作为初始病毒浓度, 将其作为1个单位(Unit)计算, 检测植株完全发病后(14 d)病毒累积量。图3表明, 感病对照品种南农1138-2在接种2个SMV分离物完全发病后, 病毒浓度累积均在100 U以上。而其余3个大豆品种在接种Sc6以后其14 d的浓度积累均未超过100 U, 其中最低的为冀豆17, 浓度仅约为27 U。但接种HB-RS后, 除冀豆17外, 其余3个品种病毒浓度均超过100 U, 其中最高的为南农1138-2, 病毒浓度达到了500 U以上, 其次为五星1号, 浓度也达到了470 U以上。而整体上HB-RS在4个大豆品种中病毒积累浓度均显著高于Sc6, 但从症状上分析, Sc6侵染寄主后容易形成坏死斑, 而HB-RS则主要以轻花叶为主。以上结果表明, HB-RS在大豆植株内更具有侵染性, 冀豆17可以作为抗性品种加以推广。
SMV是大豆生产上的重要病原物之一, 国内外的学者对SMV开展了广泛研究, 目前对SMV病害的防控主要以培育抗病品种为主。重组是病毒变异的一种方式, 病毒变异可导致原有品种抗性丧失。韩国历史上就出现过SMV变异株系致当地主栽品种Kwanggyo丧失抗性, 几乎绝产。重组型SMV是SMV的一个变异种, 在我国多个大豆产区均广泛分布[14]。因此, 明确重组型SMV的侵染特性及致病力尤为重要, 对今后防控病害的蔓延和流行具有重要意义。
坏死属于植物一种抗病反应机制, 可将部分病毒限制在坏死细胞周围, 防止进一步扩散。极端的坏死反应是系统性坏死, 某些特定大豆受特定SMV侵染后也会出现系统坏死反应。这种抗性机制在实际生产中会造成巨大产量损失, 因此育种上将此反应作为感病考虑。试验中南农1138-2和五星1号受Sc6侵染后, 接种叶和上位叶均表现出坏死反应, 表现出较为严重的危害, 从育种角度上划分属于高感品种。Pasin等[18]通过突变PPV的P1蛋白编码区, 发现P1属于负调控基因, P1功能的改变或缺失会导致植株症状减弱同时病毒复制能力增加。试验中HB-RS与Sc6在侵染南农1138-2和五星1号后引起的症状上具有明显差异, HB-RS在4个品种中病毒积累的浓度也均明显高于Sc6, 表现出较强的侵染力, 推测这种症状和侵染力的差异可能跟2个分离物在P1编码区的差异有关。
此外, 大豆对侵染力强的株系并不一定表现更差的抗性, 这是因为植株症状反应是病毒与寄主互作的结果, 育种过程中指的品种抗性主要根据症状反应和产量损失来界定。某些抗性机制不一定对生产有利, 如系统性坏死抗病反应, 不同的界定标准使大豆品种的抗性与株系本身的强弱没有必然的关联。尽管如此, HB-RS的高侵染特性仍值得我们关注和建立针对性的预警机制, 以防其进一步发生变异。
完成了对HB-RS分离物的全序列测定。HB-RS由9993个核苷酸组成, 包含一个开放阅读框, 翻译后形成3202个氨基酸, 是一个典型的重组型SMV分离物。4个大豆品种中冀豆17抗性最好, 对HB-RS和Sc6分别表现抗病和高抗。HB-RS在4个大豆品种上显示出比Sc6更强的侵染性, 但在冀豆17植株体内浓度积累均较低, 因此冀豆17可作为抗性品种推广或用作抗病育种亲本材料。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|