小麦穗部性状与单株产量密切相关。本研究以小麦骨干亲本燕大1817与优良品系北农6号衍生的269个重组自交系为材料, 通过在北京和河北石家庄的2年田间试验数据, 利用本实验室已构建的高密度SNP和SSR遗传连锁图谱进行穗长、穗粒数和穗粒重QTL定位。采用完备复合区间作图法共检测到29个穗部性状加性效应QTL, 其中10个穗长QTL分布于1B、2D、3A、3B、4A、5A、5B、6A和7D染色体上, 解释的表型变异率为2.96%~9.63%, QSl.cau-4A.2在所有5个环境中均能被检测到, 解释的表型变异为5.89%~9.62%, 另有7个QTL能在2个或2个以上环境中被检测到; 8个穗粒数相关QTL分布于1A、3A、3D、4A和5B染色体上, 解释的表型变异为4.06%~11.17%, 为单个环境QTL。11个与穗粒重相关QTL分布于1A、1B、2A、2D、3A、4D、5A、5B和6B染色体上, 解释的表型变异为2.79%~16.12%, 其中 QGws.cau-1B、QGws.cau-3A和 QGws.cau-6B.2在2个或者2个以上环境中能被检测到。另外, 鉴定出6个分布于1A、2D、3A、4A和5B染色体上的QTL富集区段。
Spike length (SL), grain number per spike (GNS), and grain weight per spike (GWS) are important spike traits associated with yield in wheat. In this study, quantitative trait loci (QTLs) for spike traits were mapped using an available high-density SNP and SSR genetic linkage map developed from a recombinant inbred line (RIL) population of Yanda 1817 × Beinong 6. Using phenotypic data on two locations (Beijing and Shijiazhuang) in two years (2011-2012 and 2012-2013 growing seasons), 29 QTLs for SL, GNS and GWS were detected by inclusive composite interval mapping (ICIM) (LOD ≥ 2.5). Among which, 10 QTLs for spike length were mapped on chromosomes 1B, 2D, 3A, 3B, 4A, 5A, 5B, 6A, and 7D with phenotypic variations ranging from 2.96% to 9.63%. A stable and major QTL associated with SL, QSl.cau-4A.2, was detected in all the environments with phenotypic variations ranging from 5.89% to 9.62%. Eight QTLs for GNS were found on chromosomes 1A, 3A, 3D, 4A, and 5B with phenotypic variations from 4.06% to 11.17%. However, they were all environments sensitive and detected in a single environment. Eleven QTLs for GWS were mapped on chromosomes 1A, 1B, 2A, 2D, 3A, 4D, 5A, 5B, and 6B with phenotypic variations ranging from 2.79% to 16.12%. The QTL QGws.cau-6B.2was identified in three environments and the favorable allele was contributed by Beinong 6. The stable QTLs on chromosomes 3A, 4A, 6B, and 7D identified in this study may serve as target sites in marker-assisted selection of spike related traits in wheat breeding program.
小麦穗长、穗粒数和穗粒重是影响产量的重要农艺性状, 解析其遗传机制对小麦遗传改良有重要意义。小麦穗部相关性状是多基因控制的数量性状, 遗传基础复杂, 且易受环境影响[1]。国内外研究者利用不同群体进行小麦穗部相关性状的QTL定位已有诸多报道。Bö rner等[2]和Jantasuriyarat等[3]利用以Opata85和人工合成小麦W-7984为亲本构建的RIL群体(又称国际作图群体, ITMI)在多个环境中定位了穗长、穗粒数、粒重等农艺性状QTL。Cui等[4, 5]、姚琴等[6]和Ma等[7]分别利用不同的小麦重组自交系群体, 对穗部相关性状进行了QTL定位, 并分析了穗部性状间的遗传关系。Sourdille等[8]、Heidari等[9]和张坤普等[10]利用小麦DH群体定位了控制穗部相关性状的QTL。BC2F3群体[11]、人工合成大穗多粒小麦[12]和F2群体[13]也被用来研究穗部相关性状的遗传基础。此外, Zhang等[14]利用中国微核心种质, 通过全基因组关联分析, 发现了一些和籽粒性状相关基因位点。
近年来, 小麦上开发了基于Illumina技术平台的9K[15]和90K[16]基因芯片, 鉴于其高通量且易于实现自动化分析的特点, 被广泛应用于小麦SNP多态性检测, 极大地提高了遗传连锁图谱的密度。前人进行小麦重要农艺性状的QTL定位多是利用SSR和STS等标记, 迄今为止尚未有利用高密度SNP遗传连锁图谱进行小麦穗部性状QTL定位的报道。本研究利用SNP构建的高密度遗传连锁图谱, 可以提高作图精度, 缩小QTL置信区间, 利于为QTL精细定位和克隆奠定坚实的基础。
燕大1817是我国小麦育种骨干亲本之一, 由原燕京大学作物改进场从山西省地方品种平遥小白麦系选而成, 以抗逆性而著称。北农6号是北京农学院上世纪90年代育成的具有高产潜力的小麦品系。燕大1817和北农6号在籽粒形态及大小、株高、穗长、穗粒数及穗粒重等性状上差异显著, 且后代遗传变异丰富, 是研究小麦农艺性状重要基因/QTL比较理想的材料。
本研究以燕大1817和北农6号组配得到的重组自交系群体为材料, 利用本实验室通过SSR和Illumina 9k iSelect SNP chip构建的高密度遗传连锁图谱(Wu等, 未发表数据)对穗长、穗粒数和穗粒重进行QTL定位, 旨在为解析小麦穗部性状QTL及其遗传效应和分子标记辅助选择育种奠定基础。
燕大1817和北农6号杂交(燕大1817为母本, 北农6号为父本)经单籽粒传法得到269个F10~F11重组自交系(RIL)群体。2011— 2012和2012— 2013年度, 分别在北京市中国农业大学上庄实验站和河北省石家庄市高邑县原种场种植该RIL群体及其亲本, 采用随机区组设计, 常规管理。2011— 2012年度为2行区, 2012— 2013年度4行区, 行长2 m, 行距0.25 m, 3次重复。其中2011— 2012年度, 北京试点又分为未接种白粉菌和接种白粉菌两种环境。
小麦成熟后, 从每个小区随机选10个代表性单株调查穗长, 然后取10个植株的主穗混合脱粒调查穗粒数和穗粒重, 各性状取3次重复的平均值用于数据分析和QTL定位。2011— 2012年度石家庄试点未调查穗粒数和穗粒重。
利用Microsoft Excel和SPSS 20.0软件对穗长、穗粒数、穗粒重进行平均值、标准差及相关性的统计分析。利用SAS9.3软件分析各性状的广义遗传力(h2), 遗传模型为h2=VG/VP。根据本实验室已构建的包含2431个SNP和128个SSR标记的高密度遗传连锁图谱(Wu等, 未发表数据), 利用基于逐步回归的完备复合区间作图法[17]的IciMapping 4.0软件(http://www.isbreeding.net/)的BIP功能进行QTL定位, 步移速度为1.0 cM, P值临界值为0.001。以LOD=2.5为统计检测阈值, 即当LOD值大于2.5时认为该处存在一个真实有效的QTL[18]。
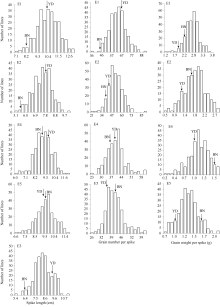
2个亲本穗部性状差异较大, 燕大1817在穗长和穗粒数上为高值亲本, 而北农6号在穗粒重上为高值亲本; 但2013年河北省高邑县原种场燕大1817的穗长和穗粒数低于北农6号, 与其他几个环境下的结果不一致(表1和图1)。RIL群体在穗长、穗粒数和穗粒重3个性状上变异幅度大, 其中穗长呈现典型的正态分布, 穗粒数和穗粒重的表型稍微有些偏离(图1)。各性状在所有环境下都存在明显的双向超亲分离现象, 表明为多基因控制的数量性状, 适于进行QTL定位。穗长的广义遗传力较高, 为95.30%, 而穗粒数和穗粒重的广义遗传力低于穗长, 分别为54.77%和51.36% (表1)。
| 表1 燕大1817/北农6号RIL群体在不同环境下穗部相关性状的变异 Table 1 Phenotypic performance and distribution of spike related traits in the RILs of Yanda 1817/Beinong 6 at different environments |
3个穗部性状在所有环境中都存在正相关。穗长、穗粒数和穗粒重的平均值之间存在显著相关, 且穗粒重和穗粒数的相关性最高, 相关系数为0.435, 穗长和穗粒数相关系数为0.197, 而穗长和穗粒重相关系数为0.125。穗粒数和穗粒重在4个环境下都存在显著相关, 相关系数0.404~0.680。穗长和穗粒数之间, 只有2012年北京2个环境表现显著正相关, 穗长和穗粒重在北京的3个环境中表现显著相关性(表2)。
| 表2 RIL群体穗长、穗粒数和穗粒重在不同环境中的相关系数 Table 2 Correlation coefficients between spike length (SL), grain number per spike (GNS), and grain weight per spike (GWS) in the RIL population in different environments |
5个环境中共检测到29个控制穗部性状的QTL, 其中10个在2个或者2个以上环境中被检测到。这些QTL分布于除1D、2B、4B、5D、6D、7A和7B以外的14条染色体上, 以5B染色体上的QTL最多, 为6个; A、B、D基因组分别有12、11和6个; 第1至第7部分同源群上依次有4、3、6、4、8、3和1个(表3和图2)。
| 表3 不同环境下检测到的控制穗部相关性状的QTL Table 3 QTLs for spike traits detected in different environments |
2.2.1 穗长QTL 5个环境中共检测到10个控制穗长的QTL, 位于1B、2D、3A、3B、4A、5A、5B、6A和7D染色体上, 解释表型变异2.96%~9.63%。有7个QTL能够在2个或者2个以上的环境中被检测到, 其中QSl.cau-3A和QSl.cau-4A.2在所有环境中均被检测到, 其增加穗长的等位效应分别来自于北农6号和燕大1817 (表3)。QSl.cau-4A.1和QSl.cau-7D在除2012年北京接种白粉菌环境外的4个环境中被检测到, QSl.cau-1B、QSl.cau-2D和QSl.cau-5B在2个环境中被检测到, 除QSl.cau-2D增加穗长的效应来自于燕大1817外, 其余都来自北农6号。仅在一个环境下被检测到的穗长QTL有3个, 其增加穗长的效应均来自燕大1817 (表3)。
2.2.2 穗粒数QTL 4个环境中共检测到8个与穗粒数相关的QTL, 分布于1A、3A、3D、4A和5B染色体上, 表型变异解释率为4.06%~11.17%, 以QGns.cau-4的表型贡献率最大。这些QTL都只在一个环境中被检测到, 且其中有6个是在2012年北京不接种白粉病的环境中能被检测到(表3)。
2.2.3 穗粒重QTL 4个环境中检测到11个控制穗粒重的QTL, 分布于1A、1B、2A、2D、3A、4D、5A、5B和6B染色体上, 单个位点可解释2.79%~ 16.12%的表型变异。其中, QGws.cau-6B.2在3个环境中被检测到, 其表型贡献率为5.70%~7.41%, 增效加性效应来自北农6号; QGws.cau-1B和QGws.cau-3A在2个环境中被检测到; 其余8个QTL只在一个环境中被检测到, 包括表型贡献率最大(16.12%)的QGws.cau-5B.2其加性效应来自北农6号, 可增加穗粒重0.16 g (表3)。
本研究中2013年河北环境中燕大1817的穗长小于北农6号, 其余环境条件下1817的穗长均大于北农6号(表1), 表明穗长易受环境因素的影响。在2012年北京、2013年北京环境下, 燕大1817的穗粒数高于北农6号, 但在2013年河北环境下, 燕大1817的穗粒数低于北农6号(表1), 这可能与河北高邑2013年春季温度较低、气温回升慢等因素有关, 因为春季低温导致不育小穗数增多, 穗粒数和穗粒重明显降低[19, 20]。2013年小麦将近成熟时降雨使小麦成熟加快, 同时大风也使部分家系倒伏, 导致籽粒欠饱满, 穗粒重低于常年水平, 其广义遗传力偏低。
在不同的研究中, 由于使用的标记及群体不同, 其结果很难用于直接比较, 但仍可提供一些参考和例证。本研究定位的部分穗长、穗粒数和穗粒重QTL与前人定位的QTL区间位置相同或邻近。不同的定位群体和实验环境所定位到的同一性状的某些QTL具有很好的一致性, 这些位点在不同来源的群体间是保守的, 为预测重要QTL在染色体上的位置进而实现QTL精细定位和克隆奠定基础[15]。
QSl.cau-1B位于标记wsnp_Ex_rep_c66255_ 64400455和Xbarc81之间(表1), Bö rner等[2]也在该区域发现了控制穗长的QTL。QSl.cau-2D与分子标记Xcfd51和Xgwm296紧密连锁, 王瑾等[12]也检测到与Xcfd51紧密连锁的控制穗长的QTL位点; Heidari等[9]和Ma等[7]都在标记Xgwm261附近检测到控制穗长的QTL, Somers等[21]通过整合SSR图谱, 发现Xgwm296和Xgwm261是紧密连锁的。本研究在多个环境中检测到4AS和4AL上控制穗长的QTL (QSl.cau-4A.1和QSl.cau-4A.2), 在QSl.cau-4A.1[2, 12]和QSl.cau-4A.2[2, 3, 4]相近位置也被报道存在控制穗长的QTL, 说明这是2个稳定的QTL。QSl.cau-5B位于5B染色体上118 cM的位置, Cui等[4]在5B染色体125 cM位置检测到一个控制穗长的QTL, 但由于邻近分子标记不同, 无法判断是否为同一位点。在7D上检测到的QSl.cau-7D位于标记Xbarc128和Xwmc221之间, 与Ma等[7]定位的QSpl.nau-7D位置相近。QSl.cau-3A在所有环境中稳定存在, 但是在该区段未见同类报道, 可能是本研究特有的位点。
由于受环境影响较大, 本研究定位的8个控制穗粒数的QTL都只能在一个环境中被检测到。QGns.cau-3D.1位于分子标记Xgwm314与Xwmc656之间, 与Huang等[11]报道的穗粒数QTL的标记Xgwm161位置相近。QGns.cau-4A是8个QTL中表型贡献率最大的一个, 该位点与Bö rner等[2]检测到的QTL位于相近位置。
关于小麦粒重相关QTL, 大多数研究考察千粒重指标, 而穗粒重QTL定位的研究报道较少。本研究中, QGws.cau-4D与Xcfd71紧密连锁, Cui等[5]在相同位置也检测到穗粒重QTL。根据连锁分子标记, 我们初步判断, 位于Xwmc719附近的穗粒重QTL [5]与本文的QGws.cau-1B可能是同一基因; 与Xbarc124紧密连锁的QTL [9]、与Xwmc28紧密连锁的QTL [9]可能分别与本文的QGws.cau-2D (与Xcfd51紧密连锁)和QGws.cau-5B.2是同一基因。本研究发现3A和6B上存在2个稳定QTL区段, 但未见类似报道。
本研究在1A、2D、3A、4A和5B染色体上发现QTL簇或QTL富集区, 控制一个或多个穗部性状, 表现出一因多效或基因紧密连锁效应, 高度相关的性状间有一些共同的QTL[10, 13]。其中, 1A上的QTL簇与穗粒数和穗粒重相关, 王瑾等[12]也在1A染色体上发现与小穗数和穗粒数相关的QTL簇; 2D上的QTL富集区同时控制穗长和穗粒重, 张坤普等[10]在该区段也检测到同时与籽粒产量、穗粒数、穗长、小穗着生密度相关的QTL; 5BL上的QTL区域, 同时定位到了与穗长、穗粒数和穗粒重相关的QTL; 4AL上的位点同时与穗长和穗粒数相关, 且效应值较高; 3A和5B上各有一个与穗粒数和穗粒重同时相关的位点, 这与穗粒数和穗粒重的表型值相关性最高是一致的。
共检测到29个控制穗长、穗粒数和穗粒重的QTL。其中10个在2个或2个以上环境中被检测到, 同时发现6个QTL富集区, 为进一步解析穗部相关性状遗传机制及分子标记辅助选择育种奠定了基础。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|