磷脂酰乙醇胺结合蛋白(phosphatidyl ethanolamine-binding proteins, PEBP)基因家族广泛存在于真核生物中, 在被子植物中主要起着促进或抑制开花和控制株型的作用。利用亚洲棉( Gossypium arboreum, A2)和雷蒙德氏棉( Gossypium raimondii, D5)的基因组数据库, 分别搜索到8个棉花PEBP同源基因, 都包含4个外显子和3个内含子, 编码的蛋白都存在PEBP家族的保守基序和关键氨基酸位点, 表明二倍体棉花中至少存在8个PEBP家族基因。进化分析表明, 8个PEBP基因分属于3个亚家族, 含 FLOWERING LOCUS T( FT)-like亚家族1个、 TERMINAL FLOWER 1 ( TFL1)-like亚家族5个(包括3个 TFL1和2个 BFT)、 MOTHER OF FT AND TFL1 ( MFT)-like亚家族2个。实时荧光定量PCR分析陆地棉( Gossypium hirsutum) 8个PEBP基因在根、茎、叶、幼苗顶端分生组织、花、胚珠和25 d的纤维组织中的表达, 表明 FT1在叶片中表达量最高, 其次在纤维、胚珠和花中; MFT1在各组织中均表达, 但在纤维中表达量最高, 其次是花和叶片中, 而 MFT2以在叶片中表达为主; TFL1a、 TFL1b和 TFL1c均在根中表达量最高, 但 TFL1c在叶片、花和胚珠中也有相对较高的表达; BFT1和 BFT2在叶片中表达量最高, 但除幼苗顶端分生组织外, BFT1在其他各组织中的表达明显高于 BFT2。这些结果表明, PEBP家族基因在棉花的生长发育中可能具有不同的功能。
The phosphatidylethanolamine-binding proteins (PEBP) widely exist in eukaryotes. In angiosperms, PEBP family genes play important role in promoting or inhibiting flowering, as well as plant architecture control. Eight PEBP genes were identified from diploid cotton Gossypium arboreum (A2) and Gossypium raimondii (D5) genome database, respectively . All the PEBP genes of cotton contained four exons and three introns, and their encoded proteins contained a conserved PEBP motif and critical amino acid sites of PEBP family, which indicated there were at least eight PEBP genes in diploid cotton. Phylogenetic analysis showed that eight cotton PEBP genes comprised three subfamilies: FLOWERING LOCUS T ( FT)-like containing one gene, TERMINAL FLOWER 1 ( TFL1)-like containing five genes including three TFL1 and two BFT genes, and MOTHER OF FT AND TFL1 ( MFT)-like containing two genes. The expression patterns of eight Gossypium hirsutum PEBP family genes in root, stem, leaf, shoot apical meristem, flower, ovule and 25 days post-anthesis (DPA) fiber were determined with quantitative Real-time reverse transcription PCR (qRT-PCR). The results showed that FT1 transcript was preferentially expressed in leaf and secondly in fiber, ovule and flower. MFT1 expressed in all the tissues, with the highest expression level in fiber, then in flower and leaf, while MFT2 transcript was preferentially expressed in leaf. TFL1a, TFL1b, and TFL1c expressed mainly in root, and TFL1c also expressed in leaf, flower and ovule. Expression of BFT1 and BFT2 were present mainly in leaf, and that of BFT1 in the other six tissues except for shoot apical meristem (SAM) were higher than that of BFT2. Expression analysis revealed that eight PEBP genes in cotton have different expression patterns, showing their different functional roles in regulation of cotton development.
磷脂酰乙醇胺结合蛋白(phosphatidyl ethanolamine-binding proteins, PEBP)家族基因因含有进化上较为保守的乙醇胺结合域而得名, 广泛存在于动、植物及酵母中[1, 2, 3]。在植物中, PEBP家族主要分为FLOWERING LOCUS T(FT)-like, TERMINAL FLOWER 1(TFL1)-like和MOTHER OF FT AND TFL1 (MFT)-like 3个亚家族[4]。其中, MFT-like是FT-like和TFL1-like亚家族的祖先, 在苔藓和石松植物中没有FT和TFL1同源基因, 但分别含有4个和2个MFT同源基因, 表明FT/TFL1-like是伴随着种子植物的进化而出现的[5]。MFT-like的主要功能与苔藓植物的配子囊、孢子体的发育以及种子发育和萌发有关, 而在种子植物由营养生长向生殖生长的转变过程中, FT/TFL1-like基因起着重要的作用[5, 6, 7, 8]。
在拟南芥基因组中, 包含6个PEBP家族基因, 分为3个亚家族, 其中FT和TWIN SISTER OF FT (TSF)属于FT-like, TFL1, BROTHER OF FT AND TFL1 (BFT)和ARABIDOPSIS THALIANA CENTRORADIALIS(ATC)属于TFL1-like, MFT属于MFT-like亚家族[8]。对FT和TFL1基因的功能研究得相对较为清楚, 但其他基因的功能还需进一步研究。FT基因编码的蛋白被认为是植物的成花激素(Florigen)[9, 10, 11]。目前研究认为, FT蛋白主要在叶片合成, 经过韧皮部到顶端分生组织, 与bZIP转录因子FLOWERING LOCUS D (FD)蛋白结合, 促进下游开花身份相关基因APETALA1(AP1)等表达, 进而促进植物开花[12]。但FT-like基因存在着功能冗余或相反的现象, 在南瓜(Cucurbita moschata)、龙胆(Gentiana triflora)和苹果(Malus × domesticaBorkh.)等植物中都存在2个FT同源基因, 且对开花均具有促进作用[13, 14, 15]; 在烟草(Nicotiana tabacum)中存在4个FT同源基因NtFT1、NtFT2、NtFT3和NtFT4, 在短日照条件下只有NtFT4能够促进开花, 其他3个FT同源基因均对开花有抑制作用[16]; 在甜菜(Beta vulgaris)中有2个FT同源基因相互作用调节开花, 其中BvFT2是FT的功能保守基因, 促进开花, 而BvFT1抑制开花, 并且其表达下调对甜菜春化反应起着关键作用[17]。TFL1-like与FT-like的序列相似性很高, 但二者功能正好相反, TFL1-like抑制开花并维持花序分生组织的无限生长, 例如金鱼草(Antirrhinum majus)的CEN基因和拟南芥(Arabidopsis thaliana)的TFL1基因[18, 19]。FT/TFL1-like的这种功能差异主要由2个关键氨基酸位点不一致造成的, FT中为Tyr85, 而TFL1中为His88[20]。此外, FT/TFL1-like第4外显子上14个氨基酸片段LGRQTVYAPGWRQN和三联体LYN, 也对FT/TFL1-like的功能相反起着重要作用[21]。
棉花是一种重要的纤维作物, 农业生产中所栽培棉花的种类为四倍体的陆地棉(Gossypium hirsutum)和海岛棉(Gossypium barbadense), 它们都是在进化过程中由A基因组供体— — 亚洲棉(Gossypium arboreum, A2)和D基因组供体— — 雷蒙德氏棉(Gossypium raimondii, D5)种间杂交形成的[22]。对棉花PEBP家族基因的克隆和功能分析, 有利于解析棉花生长发育及开花的分子机制, 并利用这类基因调节棉花的开花时间, 对提高棉花产量和纤维品质具有重要的理论指导意义。近年来, 本实验室一直开展棉花PEBP家族基因的鉴定和功能分析, 前期从陆地棉中克隆了一个编码成花素的同源基因GhFTL1 (本研究中该基因被命名为GhFT1), 在拟南芥中过量表达可以促进早花[23]。从海岛棉中克隆了2个MFT-like同源基因GbMFT1和GbMFT2, 二者在萌发的种子中表达量较高, 并且GbMFT2受ABA诱导[24, 25]。Argiriou等[26]从陆地棉中分离了2个TFL1- like基因GhTFL1a和GhTFL1b, 二者主要在根中表达。由于栽培棉是异源四倍体, 基因组较为复杂, PEBP家族基因的研究远远落后于其他植物。随着亚洲棉和雷蒙德氏棉基因组测序的完成, 为在全基因组水平上分析棉花PEBP家族基因提供了可能[27, 28]。本研究利用二倍体棉花基因组数据库分析棉花A2、D5基因组中的PEBP家族成员, 并利用棉花丰富的EST数据库查找PEBP基因转录本, 利用我们前期对棉花PEBP基因研究的成果, 结合发表的相关文献, 分析它们在陆地棉的不同组织中的表达特征, 为进一步研究棉花PEBP家族基因的功能奠定了基础。
2014年4月25日, 将陆地棉(G. hirsutum)品种新陆早33种植于石河子大学试验农场。采集播种后40 d的根、茎、真叶和棉花顶端, 在解剖镜下剥去叶片将幼苗顶端分生组织分离出来; 采集开花当天的花、胚珠以及25 DPA (day post-anthesis)的棉桃, 将纤维从棉桃中剥离。将所有棉花组织立即浸没于液氮中, -80℃保存备用。
分别以拟南芥的6个PEBP家族基因FT (GenBank登录号为AF152096.1)、TSF (GenBank登录号为AF152907.1)、MFT(GenBank登录号为AF147721.1)、TFL1 (GenBank登录号为U77674.1)、ATC (GenBank登录号为AB024714.1)和BFT (GenBank登录号为NM_125597.1)的开放阅读框(ORF)序列为查询序列(query), 利用植物比较基因组学Phytozome V.9.1 (http://www.phytozome.net/)门户网站, 选择物种G. raimondii, 在Blast program中选择TBlastX程序, 其他为缺省值, 搜索棉花D5基因组数据库, 选择比对结果中E值小于E-10的序列, 并根据数据库给出的功能注释(functional annotations)筛选PEBP基因。用Pfam (http://pfam.sanger.ac.uk/)程序对基因编码的蛋白序列进一步鉴定[29], 参数设置为缺省值。利用NCBI (http://www.ncbi.nlm.nih. gov/)的BlastN工具(参数设置为缺省值)比对分析棉花D5基因组PEBP家族基因与GenBank数据库中其他PEBP基因的相似性, 并根据比对结果给基因命名。
利用NCBI的TBlastX工具, 同样分别以拟南芥的6个PEBP家族基因的ORF为query, 选择全基因组鸟枪(whole-genome shotgun contigs, wgs)数据库, Organism限制为G. arboreum, 其他为缺省值, 搜索棉花A2基因组上的PEBP家族序列, 选择比对结果中E值小于E-10的序列, 并用MegAlign程序与D5基因组上的PEBP家族基因逐一比对分析; 选择Expressed sequence tags (EST)数据库, Organism限制为Gossypium, 查找棉花EST数据库中的所有PEBP家族基因的EST序列。
利用ScanProsite (http://prosite.expasy.org/)在线工具搜索棉花PEBP家族蛋白的保守结构域。运用Clustal X2程序比对蛋白序列。利用MEGA 5.1进行拟南芥和棉花PEBP家族蛋白的系统进化分析[30], ClustalW比对蛋白氨基酸序列, 邻位相连法(Neighbor-Joining, NJ)构建进化树, BootStrap参数设置为1000次重复, 使分支结果更为可靠。
采用杭州博日科技有限公司(BIOER) Biospin多糖多酚植物总RNA提取试剂盒提取新陆早33棉花不同组织的总RNA, 方法参照试剂盒说明书。利用Supermo III M-MLV反转录酶(北京百泰克)合成cDNA第1链。
根据上面分析二倍体棉花PEBP家族基因的序列, 结合已经从四倍体棉花中克隆的PEBP家族基因的序列设计实时荧光定量PCR (quantitative Real- time RT-PCR, qRT-PCR)分析的引物(表1), 内参基因为棉花的Ubiquitin7 (UBQ7, GenBank登录号为DQ116441.1)。以棉花不同组织的cDNA为模板, 采用北京康为世纪有限公司FASTSYBR Mixture (With ROX)试剂盒, 利用7500 Fast实时荧光定量PCR仪(Life Technologies, Foster City, CA, USA)进行基因表达量的检测。检测每份样品目的基因和内参基因的Ct值(循环阈值), 每份样品3次重复。
| 表1 本研究所用的引物 Table 1 PCR primers used in this study |
qRT-PCR总体系为10.0 μ L, 其中cDNA 30 ng, 2 × FASTSYBR混合物5.0 μ L, 基因特异正向引物(10 μ mol L-1) 0.2 μ L, 反向引物(10 μ mol L-1) 0.2 μ L, 用RNase-Free H2O补足到10.0 μ L。采用二步法PCR, 反应程序为95℃预热10 min, 95℃ 15 s, 60℃ 1 min, 共40个循环, 熔解曲线阶段为仪器默认程序。采用2-Δ Ct法分析试验数据, 先分别计算出每组的Δ Ct= Ct目的基因-Ct内参基因, 再根据Δ Ct值求出2-Δ Ct以及标准误, 使用Microsoft Excel 2010软件处理数据。
通过比对分析G. raimondii (D5)基因组数据库, 共得到8个PEBP家族基因, 它们分别位于第1、第4、第6、第7、第9染色体上(表2)。Blast比对分析8个PEBP家族基因的CDS序列, 其中转录序列Gorai.004G264600.1与本实验室克隆的陆地棉GhFT1 (GenBank登录号为HM631972.1)[23]的相似性为100%; Gorai.009G174600.2与GbMFT1 (GenBank登录号为KC513744.1)[24]的相似性为98%, 它们之间仅有几个碱基差异, 可认为是同一个基因; Gorai.006G192300.1与GbMFT2 (GenBank登录号为KF739071.1)[25]的相似性为100%, 说明是MFT2基因; Gorai.001G121800.1与GrTFL1a (GenBank登录号为EU026438.1)[26]的相似性为100%, 与GhTFL1a[26]的相似性为99%, 为TFL1a基因; Gorai.006G155800.1与GrTFL1b (GenBank登录号为EU026439.1)[26]的相似性为100%, 与GhTFL1b[26]的相似性为99%, 为TFL1b基因; Gorai.009G403800.1与GrTFL1b的相似性只有59.7%, 可能是一个新的TFL1基因, 因此被命名为GrTFL1c; Gorai.004G120400.1和Gorai.007G010800.1与其他物种的BFT基因相似性较高, 例如它们所编码的蛋白与大豆GmBFT (GenBank登录号为NM_ 001249668.1)所编码的蛋白序列相似性分别为82%和83%, 它们之间的核苷酸序列相似性为85%, 说明是2个不同的BFT基因, 因此被命名为GrBFT1和GrBFT2。
虽然G. arboreum (A2)基因组草图已经测序完成, 在GenBank中只能搜索到A2基因组的散弹(wgs)序列。通过比对, 从A2基因组中获得了8个wgs序列, 通过和相应D5基因组的PEBP基因比较, 获得了A2基因组的8个PEBP家族基因(表3)。
在棉花的EST数据库中, 也只从陆地棉、海岛棉和亚洲棉中搜索到8个属于棉花PEBP家族的EST序列(表4)。Blast比对分析表明, JK805297.1、ES826802.1和JG852183.1为GhFT1的EST序列, ES800631.1、DW499267.1和BQ410064.1为GbMFT1的EST序列, DW506990.1和DW506935.1为GbMFT2的EST序列, 另外5个棉花PEBP家族基因未能找到相关的EST序列。
综上所述, 在棉花A2、D5基因组中均存在8个PEBP家族基因, FT1同源基因只有1个, TFL1同源基因有5个, MFT同源基因有2个(表2和表3)。而目前的棉花EST数据库中只存在8个PEBP家族基因高度相似序列, 分别对应GhFT1、GbMFT1和GbMT2基因(表4)。因此, 在棉花中可能存在8个PEBP家族基因。并且在四倍体棉花中除TFL1c、BFT1和BFT2没有相关的克隆报道外, 其余5个基因均己克隆(表2和表3)。
| 表2 雷蒙德氏棉(G. raimondii, D5)基因组中的PEBP家族基因 Table 2 PEBP family genes in G. raimondii (D5) genome |
| 表3 亚洲棉(G. arboreum, A2)基因组中的PEBP家族基因 Table 3 PEBP family genes in G. arboreum (A2) genome |
| 表4 棉花PEBP基因家族EST序列信息 Table 4 EST sequences of PEBP family genes in cotton |
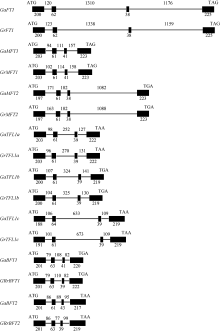
分别比较亚洲棉和雷蒙德氏棉8个PEBP家族基因的ORF及ORF相对应基因组区域的序列, 分析这8个基因的外显子和内含子结构。结果显示和拟南芥PEBP基因结构相似, 棉花的8个PEBP基因均由4个外显子和3个内含子组成, 并且差异主要在内含子部分(图1)。说明棉花PEBP家族基因的结构在进化上十分保守, 可能具有类似的功能。
PEBP蛋白家族中通常含有一个长度为23个氨基酸的保守基序[FYL]-x-[LVM]-[LIVF]-x-[TIVM]- [DC]-P-D-x-P-[SNG]-x(10)-H。将拟南芥中的6个PEBP蛋白家族成员, 即AtFT (GenBank登录号为AAF03936.1)、AtTSF (GenBank登录号为AAF03937.1)、AtTFL1 (GenBank登录号为AAB41624.1)、ATC (GenBank登录号为BAA75931.1)、AtBFT (GenBank登录号为NP_201010.1)和AtMFT (GenBank登录号为AAD37380.1), 与棉花PEBP蛋白家族GhFT1 (ADK95113.1)、GbMFT1 (AGJ98454.1)、GbMFT2 (AIE37919.1)、GhTFL1a (ABW24970.1)、GhTFL1b (ABW24964.1)、GrTFL1c、GrBFT1和GrBFT2氨基酸序列比对分析(图2), 表明棉花PEBP蛋白中都存在PEBP保守基序和决定FT/TFL1功能的关键氨基酸位点Tyr85 (Y)/His88 (H), 该位点在GhFT1中为Y, 在棉花TFL1-like中均为H; 同样, 另外一个关键氨基酸Gln140 (Q)/Asp144 (D), 该位点在GhFT1中为Q, 在棉花TFL1-like中均为D。
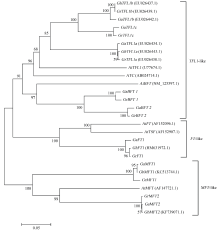
将A2基因组中推测的8个、D5基因组中推测的8个及已经报道的5个四倍体棉花的PEBP家族基因, 与拟南芥6个PEBP基因构建进化树, 结果表明, 棉花PEBP家族被明显地分为3个分支, 即FT-like、TFL1-like和MFT-like亚家族(图3)。
qRT-PCR分析表明, 8个PEBP基因在陆地棉的根、茎、叶、幼苗顶端分生组织、花、胚珠和25 d的纤维等7个组织具有完全不同的表达模式(图4)。FT1在叶片中的表达量最高, 在花、胚珠和25 d的纤维中也有较高的表达, 但在根和幼苗顶端分生组织中几乎不表达(图4-A); MFT1在各组织中均有表达, 但在25 d的纤维中表达量最高, 其次是花和真叶(图4-B); MFT2在真叶和幼苗顶端分生组织中具有较高的表达(图4-C); TFL1a、TFL1b和TFL1c均在根中的表达量最高(图4-D, E, F), TFL1a在茎秆和SAM中有弱的表达, TFL1b在其他组织中几乎检测不到, 但TFL1c在真叶、花和胚珠中也有较高的表达; BFT1和BFT2在所检测的7个棉花组织中均有表达, 且在叶片中的表达量最高, 但除幼苗顶端分生组织外BFT1在其他6个组织中的表达明显高于BFT2 (图4-G, H)。
目前, 全基因组测序及数据挖掘是生物学研究的热点领域之一。二倍体棉花G. arboreum (A2)和G. raimondii (D5)测序工作的完成, 为利用全基因组数据研究棉花中基因家族的分类、序列特点、新基因的克隆、功能预测以及异源四倍体棉花序列分析等提供了方便。本研究利用G. raimondii (D5)基因组数据库, 查找到棉花中可能存在的8个PEBP基因, 它们分别位于第1、第4、第6、第7和第9染色体上(表2)。与G. raimondii相一致的是, 在G. arboreum(A2)的wgs基因组序列数据库中也只找到8个PEBP基因, 分别位于第1、第3、第10、第11和第12染色体上(表3)。结构分析表明, 棉花8个PEBP基因不仅具有植物PEBP家族基因类似的基因结构(图1), 并且蛋白序列中含有PEBP家族蛋白的保守基序; 在FT/FTL1-like中, 决定促进或者抑制开花的关键氨基酸位点在棉花中也是一致的(图2), 表明植物PEBP家族基因功能在进化上具保守性。
进化分析表明, 棉花中的PEBP家族基因也分为3个亚家族。其中, FT-like只有1个FT1基因; TFL1-like包括TFL1a、TFL1b、TFL1c、BFT1和BFT2共5个基因; MFT-like包括MFT1和MFT2两个基因(图3)。在拟南芥[8]、南瓜[13]、龙胆[14]、苹果[15]、烟草[16]等植物基因组中均存在多个FT-like基因, 并且存在功能冗余或相反的现象, 但在二倍体G. arboreum和G. raimondii基因组中只存在1个FT-like基因, 与我们前期在四倍体G. hirsutum中克隆的一个GhFT1基因一致[23]。值得注意的是, 在四倍体棉花中可能还存在其他FT-like基因, 但是目前只发现1个GhFT1。拟南芥基因组中存在3个TFL1-like基因[8], 且均抑制开花, Argiriou等[26]在四倍体G. hirsutum、二倍体G. arboreum和G. raimondii中均克隆到了2个棉花TFL1-like基因TFL1a、TFL1b, 但TFL1c、BFT1、BFT2尚无相关研究报道。迄今为止, 除了在苔藓和石松植物中分别含有4个和2个MFT同源基因外, 在多数种子植物(如大豆[5]、小麦[7]、拟南芥[8]等)中, 只发现1个MFT-like基因, 其功能多与种子萌发有关, 我们前期在四倍体G. barbadense中克隆了GbMFT1和GbMFT22个MFT-like基因, 与二倍体G. arboreum和G. raimondii基因组中的2个MFT-like一致[24, 25]。
根据GenBank中统计结果(2013年1月1日), 棉花EST数目共有464 601条(http://www.ncbi.nlm. nih.gov/genbank/dbest/dbest_summary), 包括G. hirsutum 297 522条、G. arboreum 64 087条、G. raimondii63 577条。但在棉属EST数据库中, 只检索到8个PEBP家族基因EST序列, 分属于FT1、MFT1和MFT2基因(表4)。分析原因, 一方面由于EST主要是通过cDNA文库大规模随机测序获得, 有很多组织的cDNA文库少有构建, 就很难获得组织特定表达的基因; 另外由于基因表达量很低, 一些基因在表达量很低的组织中很难被检测到, 使EST数据库中很少有这些基因的EST序列信息。
与我们前期的研究[23]一致, 除了在根中很难检测到GhFT1基因的表达外, 在叶片、茎秆、花、胚珠等棉花不同组织中都有表达, 但在叶片中的表达最高(图4-A)。在拟南芥中过量表达GhFT1可以明显地促进早花[23], 暗示着棉花FT1基因可能起着开花促进作用, 并与拟南芥AtFT基因作用模式一样[9, 10, 11], 在叶片中转录为mRNA并合成FT1蛋白, FT1蛋白再由韧皮部经过长距离运输到顶端分生组织, 激活下游信号, 促进植物开花。GhMFT1在花、叶片、纤维中有较高的表达(图4-B), 与GbMFT1[24]的表达类似; GhMFT2在叶和SAM中表达相对较高(图4-C)。近年来的研究表明, 高等植物中MFT-like基因调控种子的发育[5, 6, 7, 8, 24], 我们前期的研究也表明G. barbadense的GbMFT1和GbMFT2在萌发的种子中高表达, 并且GbMFT2基因的表达受ABA诱导[24, 25]。TFL1a、TFL1b均在根中的表达量最高(图4-D, E), 其基因表达模式和Argiriou等[26]研究结果相似, 但它们在根中的分子机制及功能并不十分清楚[26]。TFL1c除了在根中表达量最高外, 在真叶、花和胚珠中也有较高的表达(图4-F), BFT1和BFT2均在叶片中表达较高(图4-G, H), 并且它们编码的蛋白都具有类似TFL1的关键氨基酸位点His (H)和Asp (D), 推测其除了可能具有抑制开花的作用以外, 可能还具有其他重要的功能。棉花的8个PEBP基因, 除了TFL1a、TFL1b外, 均在叶片中高表达, 表明TFL1a、TFL1b在分生组织的维持和分化过程中可能不受光周期的直接诱导; 而FT1、MFT1、MFT2、TFL1c、BFT1、BFT2这6个PEBP基因的调节可能受光周期的诱导。
总之, 在二倍体棉花基因组A2和D5中均存在8个PEBP家族基因, 其中5个与已知的四倍体棉花PEBP基因序列一致, 且这8个PEBP基因在四倍体陆地棉中均有表达, 表明棉花PEBP基因家族在二倍体中数目较为保守, 但四倍体陆地棉由A2与D5基因组进化而来, 四倍体棉花也可能具有A2与D5两套的PEBP家族基因, 同时也不排除四倍体棉花PEBP基因家族在进化过程中由于重复基因间的相互作用只有一组二倍体基因组(A2或D5)来源的PEBP基因表达[31]。因此, 在四倍体棉花中究竟存在多少个PEBP基因, 相应的基因序列与二倍体有什么不同, 以及它们在棉花发育过程中的功能需要进一步的克隆研究。本研究为进一步解析棉花发育及开花的分子机制提供了研究基础, 将来利用这类基因调节棉花的开花时间, 对提高棉花产量和纤维品质具有重要的理论指导意义。
在棉花二倍体G. arboreum(A2)和G. raimondii(D5)的基因组上存在8个PEBP家族基因, 它们所编码的蛋白存在一个长度为23个氨基酸的PEBP保守基序; 棉花的8个PEBP家族基因包括4个外显子和3个内含子, 并分属3个亚家族, 即1个FT-like, 2个MFT-like, 5个TFL1-like (包括3个TFL1和2个BFT基因); 8个PEBP家族基因在棉花不同组织中具有不同的表达模式, 表明它们在棉花分生组织决定中可能起完全不同的作用。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|