DNA甲基化作为一种表观遗传调控方式, 在植物体生长发育中起重要作用, 而甲基转移酶I (DNA methylase I, MET1)在甲基化过程中起主要作用。本研究克隆到5个油菜 BnMET1同源基因, 并比较了白菜 BrMET1、甘蓝 BoMET1和油菜 BnMET1同源基因的进化及其表达模式。结果显示, 白菜和甘蓝 MET1同源基因在进化中较为保守, 而油菜 BnMET1基因结构发生较大改变; 部分油菜 BnMET1同源基因表达发生沉默, 而激活表达的 BnMET1表达模式较其亲本白菜和甘蓝直系 MET1同源基因发生明显的改变。上述结果表明, BnMET1同源基因通过改变基因结构以及表达模式影响油菜组织中 BnMET1基因剂量, 从而调节油菜的生长发育。
Methylation in genomic DNA, a way of epigenetic regulation, plays an important role during the growth and development of plant. As allotetraploid oilseeds plant, Brassica napus (AACC, 2 n = 38) originated from the natural cross between Brassica napus(AA, 2 n= 20) and Brassica oleracea (CC, 2 n=18), containing duplicate genes. Here, we isolated and characterized five BnMET1s, which are orthologous gene of Arabidopsis AtMET1 acted as transferase in DNA methylation. Our results showed that there were conspicuous variations in gene structures of BnMET1s compared with its orthologos of BrMET1and BoMET1, leading to more abundant divergences in coding region of BnMET1s. Moreover, we found that some divergences among BnMET1 paralogous genes were derived from its progenitor orthologous genes of B. rapa or B. oleracea. Furthermore, the transcription analysis indicated that partial BnMET1 paralogs were silence, and the expression patterns of the activated BnMET1 were altered in contrast to its BoMET1 orthologs in B. oleracea and BrMET1 orthologs in B. rapa. Taken all these together, we speculated that duplicate BnMET1s regulate the development process of B. napus with a certain gene dosage kept by altering their gene structures and spatio-temporal expression patterns.
DNA甲基化作为调控植物生长发育的一种重要方式, 其作用机制是对基因DNA序列中胞嘧啶进行甲基化修饰, 从而影响基因的表达(基因被激活或者被沉默)来调控植物的生长发育[1, 2]。DNA甲基化在不同物种、不同时空、不同器官之间表达的变化是植物正常生长发育所必需的[3, 4]。目前, 多种植物甲基化酶基因被克隆并进行了详细的功能分析, 但是对于大多数为多倍体的农作物其甲基化酶研究相对滞后。因此, 对多倍体甲基化酶基因的克隆和研究, 有助于阐明多拷贝甲基化酶基因对多倍体作物生长调控的机制。
DNA甲基化是新生DNA链胞嘧啶(C)被甲基化酶共价甲基修饰的过程[5]。植物细胞中甲基化酶较多, 根据甲基化酶的结构和功能差异, 可将植物细胞中的DNA甲基化酶分为MET1甲基转移酶、染色质甲基化酶和结构域重排甲基转移酶3类[6]。MET1甲基转移酶主要是对新生DNA链中重复和单拷贝序列进行甲基化修饰[7], 从而维持基因组DNA序列中的甲基化水平[8]。现已在诸多植物中分离鉴定到MET1及其同源基因[9]。第一个植物MET1甲基转移酶基因由Finnegan等[10]在拟南芥中分离得到。拟南芥ATMET1突变体植株具有矮小, 开花延迟等表型[11, 12], 这些表型主要是由于甲基化酶突变使得特殊位点的甲基化缺失导致。研究发现 ATMET1突变体中开花控制因子FWA缺少甲基化导致开花延迟[13]。在需春化作用的小麦中也发现同样的现象, 从小麦克隆的5个甲基化酶基因, 在春化小麦中的表达受到推迟或抑制[14], 表明小麦营养生长向生殖生长转化过程与甲基化相关。从烟草克隆的甲基转移酶NtMET1, 其反义转基因导致烟草基因组甲基化水平下降, 导致植株的形态建成受到影响[15]。从草莓基因组克隆的2个MET1基因, FaMET1a和FaMET1b, 体外繁殖试验表明其表达水平与草莓基因组整体甲基化水平具有一定相关性[16]。此外, 植物甲基化酶MET1还参与植物的应激反应。研究发现, 玉米甲基转移酶ZmMET1对低温处理后的植物部分组织DNA甲基化水平降低具有抑制作用[17]。从冷处理水稻材料表达谱发现OsMET1表达水平显著上升[18]。
甘蓝型油菜(Brassica napus, AACC)是我国最重要的油料作物之一, 是由白菜(Brassica rapa, AA)和甘蓝(Brassica oleracea, CC)自然杂交进化形成的异源四倍体作物[19]。白菜和甘蓝基因组测序表明, MET1基因在白菜和甘蓝基因组中均有3个旁系同源基因[20, 21]。最近, 甘蓝型油菜基因组测序表明甘蓝型油菜基因组中有6个BnMET1旁系同源基因[22]。这些BnMET1同源基因在油菜基因组形成和进化过程中, 通过何种方式调控加倍后同源基因的表达, 维持一定水平的BnMET1基因剂量, 影响油菜的生长发育, 目前不得而知。本研究克隆油菜基因组BnMET1同源基因, 通过分析其与白菜和甘蓝MET1直系同源基因的进化关系以及在不同器官的表达模式, 揭示多倍体油菜中多拷贝BnMET1基因协同表达调控油菜生长发育的模式。
利用拟南芥AtMET1(Arabidopsis thalianaMET1, AtMET, At5g49160)蛋白序列在白菜数据库BRAD (http://brassicadb.org/brad/)进行BlastP和基因注释检索, 获得白菜和甘蓝MET1基因。以白菜和甘蓝MET1基因CDS序列为模板, 分别设计油菜BnMET1基因同源克隆引物(表1)。
| 表1 克隆BnMET1基因所用引物 Table 1 Primers used for cloning BnMET1 genes |
克隆所得的BnMET1DNA序列通过基因在线预测GENESCAN (http://genes.mit.edu/GENSCAN.html)获得BnMET1基因相应的蛋白序列和基因结构, 白菜和甘蓝MET1蛋白序列从BRAD数据库下载。利用MEGA软件构建基因进化树。利用在线GSDS 2.0 (GSDS, http://gsds.cbi.pku.edu.cn/)软件构建基因结构图。
将油菜中双11、白菜型油菜TN004和羽衣甘蓝种植在试验田中, 每个材料种植3行, 每行种植10穴, 每穴1株, 株行株距为20 cm × 40 cm。植株将抽薹开花时, 采集幼嫩叶片; 植株开花时, 标记开花时间, 收集开花后7 d (7 days after flower)、14 d和21 d角果, 从每个植株剪取1~2个角果, 十株角果混在一起; 同时从每株采取1~2个花蕾, 十株花蕾混在一起, 即每个材料具3个生物重复。田间所采集的样品用液氮速冻, 然后转入-80℃冰箱保存待用。
使用Plant RNA Mini Kit (Watson Biotechnologies) RNA提取试剂盒, 按其操作说明提取组织总RNA。取4 μ L RNA, 加入96 μ L DEPC-H2O, 振荡混匀, 用紫外分光光度计测定260 nm波长OD值, 其数值即为RNA浓度。
取1~2 g总RNA, 加DEPC水至10 μ L。加入Oligo (dT)151 μ L和10 mmol L-1 dNTPs 1 μ L, 混匀。放入PCR仪中65℃ 5 min, 然后取出置冰上, 快速加入 5× 缓冲液4 μ L、RNase inhibitor 1 μ L、0.1 mol L-1 DTT 2 μ L、5 U L-1反转录酶H 0.5 μ L, 在PCR仪上进行反转录反应(cDNA合成), PCR程序为42℃ 50 min; 45℃ 10 min; 50℃ 10 min; 70℃ 15 min。完成反应后加1 μ L 10 mg mL-1 RNase, 于37℃放置30~60 min。将分装产物保存于-80℃冰箱待用。
为了区别每个油菜、白菜和甘蓝MET1同源基因的表达差异, 根据MET1同源基因CDS序列特异性, 设计每个MET1同源基因特异对引物; 用BnACTIN、BrACTIN和BoACTIN作为油菜、白菜和甘蓝的内参(表2)。
实时荧光定量PCR使用Promega qRT-PCR mix, 反应体积20 μ L, 含qPCR SYBR Green Master Mix 10 μ L、上下游引物各加0.5 μ L、合成的cDNA经10倍稀释后的8.5 μ L, 实时定量PCR反应条件为95℃ 5 min; 95℃ 10 s, 58℃ 30 s, 65℃ 10 s; 40个循环。每个反应3个技术重复。
| 表2 完成基因表达分析所用引物 Table 2 Primers used in qRT-PCR for determiningMET1 expression |
利用拟南芥AtMET1蛋白序列在白菜数据库BRAD 进行BlastP, 获得白菜(B. rapa)和甘蓝(B. oleracea)最大相似性的基因各3个, 即Bra039939、Bra030191和Bra010026; Bol045816、Bol031719和Bol016616。进一步在白菜和甘蓝数据库中通过基因
注释方式检索白菜和甘蓝MET1基因, 获得白菜和甘蓝的MET1基因各3个, 与利用拟南芥蛋白比对结果相一致。把获得的白菜MET1基因分别命名为BrMET1-1、BrMET1-2和BrMET1-3; 甘蓝MET1基因分别命名为BoMET1-1、BoMET1-2和BoMET1-3。
油菜是白菜和甘蓝基因组杂交形成的, 由于目前尚无BnMET1相关核酸序列, 因而以白菜和甘蓝MET1基因序列为模板设计油菜BnMET1基因同源克隆引物。为获得BnMET1基因组DNA序列, 了解基因编码区结构变异, 以油菜中双11品种基因组DNA为模板, 进行PCR扩增(图1)。扩增片段经测序, 序列利用基因在线预测GENESCAN (http://genes. mit.edu/GENSCAN.html), 最终获得5个BnMET1基因的全长DNA序列, 长度分别为5290、5289、7106、1041和6389 bp。这5个BnMET1基因分别被命名为BnMET1-1、BnMET1-2、BnMET1-3、BnMET1-4和BnMET1-5。
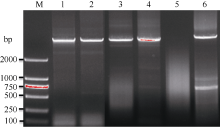 | 图1 扩增油菜BnMET1基因 M: DL2000 DNA marker, 左边数值为DNA条带相应分子量; 1~6: 同源克隆引物对(见表1)扩增产物BnMET1-2、BnMET1-1、BnMET1-4、BnMET1-3、BnMET1-6和BnMET1-5。Fig. 1 Amplified BnMET1 gene sequences from B. napus genomic DNA M: DL2000 DNA marker, of which the molecular weight of each DNA band is showed at the left of the gel, the lanes 1-6 on the gel from left to right are PCR amplified products (BnMET1-2, BnMET1-1, BnMET1-4, BnMET1-3, BnMET1-6, and BnMET1-5) with primer pairs listed in Table 1. |
为了解所获得的BnMET1基因是否源自白菜和甘蓝基因组MET1基因, 克隆获得的BnMET1基因DNA序列通过基因在线预测GENESCAN获得BnMET1基因相应蛋白序列。将5个BnMET1蛋白序列以及白菜和甘蓝MET1蛋白序列利用MEGA6.06软件进行分析, 获得BnMET1、BrMET1和BoMET1蛋白与拟南芥AtMET1蛋白进化树。
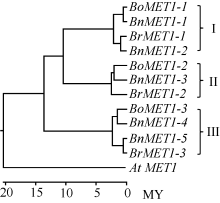
BnMET1蛋白聚簇表明, 每个BnMET1蛋白由相似性对比均能比对到与之相对应的BrMET1和BoMET1蛋白, 形成一对一的相似配对, 并聚成直系同源蛋白簇(图2): BoMET1-1与BnMET1-1相似配对, BrMET1-1与BnMET1-2相似配对, 这4个蛋白聚成一簇形成直系同源蛋白簇(I); BoMET1-3与BnMET1-4相似配对, BrMET1-3与BnMET1-5相似配对, 这4个蛋白聚成一簇(III); BoMET1-3与BnMET1-4, 由于BnMET1-6未克隆到, 缺少与BrMET1-2对应的油菜直系同源基因, 因而该簇直系同源蛋白仅有3个(II)。上述结果表明本研究所克隆的BnMET1为油菜真实MET1基因, 也表明油菜基因组进化上是由白菜和甘蓝基因组杂交而来的。
蛋白序列进化树表明, 白菜、甘蓝和油菜MET1与拟南芥MET1处于不同的分支, 分支节点表明AtMET1与BrME1T、BoMET1和BnMET1分化发生在大约2000万年前。白菜、甘蓝和油菜MET1明显分为3簇(图2)。每簇均包含白菜、甘蓝和油菜MET1基因。然而分化时间比较, 第III簇的直系同源基因分化时间明显较第I簇和第II簇早, 大约发生在1300万年前, 而I和II簇分化大约发生在1000万年前(图2)。说明白菜和甘蓝基因组形成前, 该3个MET1直系同源基因已经存在于古老的十字花科植物基因组。后续形成白菜和甘蓝物种基因组后, 3个直系同源基因变异分化过程是独立进行的。图2端支节点表明第I簇和第Π 簇中BrMET1直系同源基因变异分化较BoMET1早, 然而第III簇中BoMET1- 3直系同源基因变异分化却比BrMET1-3早。
基因结构变异分析表明, 基因结构上白菜和甘蓝MET1基因非常相似。每对白菜和甘蓝MET1直系同源基因均含有相同数目的外显子和内含子。此外, 除了BrMET1-2和BoMET1-3的基因长度具有明显的差异外, 其余基因长度基本相同。然而, 与白菜和甘蓝MET1直系同源基因结构比较, BnMET1基因结构已经发生明显的改变。BnMET1基因外显子较其两亲本MET1基因减少, 因而BnMET1蛋白较白菜和甘蓝MET1分子量变小(图3)。
根据前述蛋白进化聚类分析图2, BrMET1-1和BoMET1-1基因与BnMET1-1和BnMET1-2相近, 为直系同源蛋白。基因结构上, 与拟南芥AtMET1基因结构相近的BrMET1-1和BoMET1-1基因, 均具有11个外显子, 10个内含子。然而与其直系同源的BnMET1-1和BnMET1-2基因, 仅有6个外显子, 5个内含子。基因结构图表明(图3), BnMET1-1和BnMET1-2基因外显子发生了突变, 改变了基因的mRNA拼接位点, 导致这2个基因的外显子数目减少。
 | 图3 BnMET1、BrMET1、BoMET1和AtMET1外显子和内含子结构 结构图由在线GSDS 2.0软件显示, 图中黑色柱状表示外显子, 黑线表示内含子, 每个基因的外显子和内含子按照序列长度比例绘制, 图底为序列长度标尺。Fig. 3 Exon/intron structure of BnMET1, BrMET1, BoMET1, and AtMET1 Black boxes denote exons within coding regions and the black lines connecting them represent introns. The length of boxes and lines represents the size (bp) of the corresponding exon and intron, respectively, and the length ruler shows at the bottom of the figure. The gene structures without the untranslated regions (UTRs) were constructed using the Gene Structure Display Serve tool 2.0 (GSDS, http://gsds.cbi.pku.edu.cn/). |
此外, BnMET1-4和BnMET1-5基因和与直系同源的BoMET1-3和BrMET1-3基因之间变异较大。BnMET1-5基因在第2个外显子处发生了突变, 导致相对应的BrMET1-3基因外显子区域变成了BnMET1-5基因内含子区域。而BnMET1-4基因仅具有BoMET1-3基因后端4个外显子区段。因而, BoMET1-3基因前端区段可能变成了BnMET1-4基因的启动子区。这种改变可能导致BnMET1-4基因的时空表达发生改变, 从而影响BnMET1基因作用的时空模式。
上述结果表明, 油菜在进化过程中基因序列发生了剧烈的改变, 导致源自双亲的MET1直系同源基因序列及其结构发生明显的改变, 可能影响BnMET1基因的生物学功能及其作用方式。
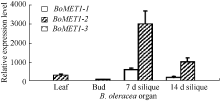
采用实时定量PCR (qRT-PCR)检测了白菜3个BrMET1旁系同源基因在白菜叶片、花蕾、7 d角果、14 d角果和21 d角果等器官中的表达水平。发现在这些器官中, 3个BrMET1旁系同源基因的相对表达水平差异较大(图4)。其中, BrMET1-1是主要表达者。BrMET1-1在叶片和7 d角果器官中表达水平较高, BrMET1-2在叶片和角果中低水平表达, 而BrMET1-3基本不表达。
在白菜3个器官中, 3个BrMET1旁系同源基因在花蕾器官中均不表达。BrMET1在早期角果器官中表达水平最高, 随着角果的发育, BrMET1表达水平迅速降低, 到14 d时, BrMET1-1表达水平降低约10倍, 至21 d时基本不表达。BrMET1这种表达模式与其功能相适应, 甲基化酶主要调控基因组DNA序列的甲基化水平, 从而调控基因的表达。白菜受精完成后, 从胚胎发育开始到胚胎逐渐形成各种组织期间, 需要对控制胚胎早期发育相关基因进行调控, 通过DNA甲基化方式控制这类基因的表达是最快速、最经济的调控方式之一。
与此相似, 3个BoMET1旁系同源基因在叶片、花蕾、7 d和14 d角果等3个器官中表达水平差异明显(图5), 这种差异模式与BrMET1旁系同源基因相似。3个BoMET1旁系基因中, 仅有BoMET1-2基因在角果发育到7 d时具高水平表达, 而BoMET1-1表达水平较低, BoMET1-3则不表达。在甘蓝3个组织中, 3个BoMET1旁系同源基因在花蕾器官中基本不表达, 与白菜的结果类似。BoMET1-2基因在角果发育早期, 在7 d时表达水平最高, 随后迅速降低, 到角果发育14 d时, BoMET1-2基因表达水平降低了2倍。在检测的3个器官中, 发育7 d的角果中BoMET1基因表达水平最高, 其次在叶片中, 然后是发育14 d的种子, 最后是花蕾, 这种表达模式与白菜类似。
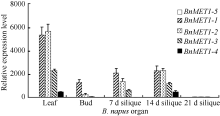
qRT-PCR结果显示, 5个BnMET1旁系同源基因在不同器官的表达模式与白菜和甘蓝MET1直系同源基因不同(图6)。BnMET1-5与其直系同源BrMET1-3基因在各自的植物器官中均不表达。与BrMET1-3直系同源的BoMET1-3基因在所检测的甘蓝器官中也不表达。然而, 与BoMET1-3直系同源的BnMET1-4在叶片和14 d角果中却被激活低水平表达。说明BnMET1旁系同源基因的表达模式由于基因组的加倍发生改变。所检测的3个油菜器官中, BnMET1-1和BnMET1-2基因在油菜叶片器官中的表达水平最高, 其直系同源基因BoMET1-2和BrMET1-1基因, 在甘蓝和白菜叶片器官表达水平相也对较高。另外, BnMET1-2在油菜叶片表达水平与BrMET1-1在白菜叶片中表达水平相近, 但是BoMET1-2在叶片器官表达水平较BnMET1-1低, 说明BoMET1-2基因融合到油菜基因组后其表达活性被增强。然而在角果发育过程中, BnMET1-1和BnMET1-2基因的表达水平明显较其BoMET1-2和BrMET1-1基因的表达水平低。上述结果说明在油菜生长发育过程中部分旁系同源基因被沉默或降低表达水平, 从而使器官中维持一定水平的基因剂量。
在所检测的白菜和甘蓝各器官中, 角果发育7 d时MET1基因表达水平最高。随着角果的发育, MET1基因表达水平急剧降低。然而在油菜角果发育过程中, 角果发育到7 d和14 d时, BnMET1基因表达水平较高, 随后表达水平才急剧降低, 说明BnMET1表达时间较白菜和甘蓝MET1延长。
DNA甲基化在植物生长和环境应激过程中起重要作用[6], 尽管DNA甲基化不改变基因的碱基序列, 但能影响所修饰基因的表达, 从而改变基因行使功能的模式[7, 8]。油菜是异源多倍体油料作物, 由白菜和甘蓝通过自然杂交加倍而成。克隆分析油菜基因组甲基化基因对了解甲基化基因调控油菜生长调控具有重要意义。本研究从油菜基因组中克隆到5个BnMET1基因, 生物信息分析发现5个BnMET1基因在油菜基因组形成稳定、进化演变过程中较其亲本白菜和甘蓝MET1基因已发生变异。表达模式分析发现白菜、甘蓝和油菜的直系同源基因在相同器官的表达模式已改变, 在所检测的器官中, 部分同源基因在不同的器官中沉默。表明油菜BnMET1同源基因通过差异表达控制基因剂量, 进而影响油菜的生长发育。
通过基因组比对和系统进化分析表明十字花科植物的分化大约发生在3800万年左右, 芸薹属植物(包括白菜和甘蓝)大约在1700万年前从古老的十字花科植物分化而来, 而白菜与甘蓝分化发生在大约800万年前[25]。白菜和甘蓝物种分化前, 芸薹属植物基因组已发生三倍化事件[20, 23]。因而, 在白菜和甘蓝基因组中分别具有3个MET1同源基因。白菜和甘蓝基因组测序表明[20, 21], 在白菜和甘蓝基因组中均有3个MET1同源基因, 基因号分别是Bra039939、Bra030191和Bra010026; Bol031719、Bol045816和Bol016616。基于油菜为白菜和甘蓝杂交形成的四倍体作物, 甘蓝型油菜基因组测序表明甘蓝型油菜基因组中有6个MET1旁系同源基因, 即BnaA04g 13310D、BnaC04g35390D、BnaA06g29420D、BnaC 07g27300D、BnaA09g42250D、和BnaC08g34700D[22]。本研究仅克隆到5个BnMET1同源基因, 未克隆到的BnMET1同源基因是由于在油菜进化的过程中, 其DNA序列发生改变, 导致其不能匹配根据亲本白菜BrMET1直系同源基因序列所设计的引物, 从而无法扩增。甘蓝型油菜测序品种为欧洲栽培油菜Darmor-bzh。中国甘蓝型油菜源自欧洲, 经国内育种工作者转育成目前国内的栽培品种。通过BnMET1 DNA序列比对可以了解不同地域性的人工选育对油菜BnMET1选择效应。但目前Darmor-bzh基因组DNA序列数据尚未释放, 不能获得Darmor-bzh基因组BnMET1DNA序列, 无法与本研究所获得的中双11 BnMET1序列比较。
甘蓝型油菜演化历程较短, 约在5000~10000年前形成[19]。因而, 在油菜基因组其基因的变异应较其亲本白菜和甘蓝变异小。本研究所克隆的5个BnMET1基因, 蛋白序列分析表明油菜直系同源蛋白与其亲本间的变异较小, 而白菜和甘蓝亲本基因组中3个旁系同源蛋白的变异却较大, 表明古老十字花科植物基因组三倍化事件较油菜形成的时间早。然而基因结构分析发现BnMET1基因结构较白菜和甘蓝直系同源MET1变异大, 而白菜和甘蓝直系同源MET1之间的变异却小。此外, MET1蛋白聚类分析也表明白菜和甘蓝3个MET1直系同源基因间的基因结构变异较小, 由序列相似性可明显形成同源配对关系, 即BoMET1-1与BnMET1-1, BrMET1-1与BnMET1-2, BoMET1-2与BnMET1-3, BoMET1-3与BnMET1-4, BrMET1-3与BnMET1-5。说明油菜基因组在稳定的过程中, 通过改变部分BnMET1同源基因的结构达到调控BnMET1同源基因功能的作用, 从而维持油菜正常生长发育。然而, 改变后的BnMET1同源基因功能是否发生改变, 有待进一步验证。本研究克隆的BnMET1同源基因为后续深入分析其功能差异奠定序列基础。
拟南芥基因组中AtMET1仅有一个拷贝(At5g49160), 而白菜和甘蓝基因组中均有3个MET1基因, 油菜有6个BnMET1基因, 不难想象如果油菜6个BnMET1基因同时表达, 且表达水平与拟南芥AtMET相同, 那么油菜各器官中BnMET1基因剂量是拟南芥的6倍, 是白菜和甘蓝的2倍, 如此高剂量的BnMET1, 必定影响油菜的正常生长。研究表明, 多倍体植物部分同源基因以沉默或活化的方式使同源基因差异表达, 以此方式控制组织中同源基因的剂量水平[24, 25], 本研究利用实时定量PCR结果也支持此结论。在白菜和甘蓝的组织中3个MET1旁系同源基因, 仅有一个同源基因在各组织器官中主要表达。在油菜中也发现同样的现象, 所克隆的5个油菜BnMET1旁系同源基因中仅有2个在油菜各组织中主要表达, 其余3个仅低水平表达。说明油菜在基因组进化和稳定的过程中通过改变BnMET1的基因结构以及旁系同源基因的表达模式来维持油菜各组织中BnMET1的基因剂量水平, 从而保证油菜的正常生长发育。然而, 这种调控模式以及作用的机制及其普遍性有待进一步研究。
在油菜基因组中克隆到5个BnMETI旁系同源基因。在油菜基因组进化和驯化过程中, 其基因结构较其亲本白菜和甘蓝基因组中MET1直系同源基因已发生明显的改变, 部分同源基因拼接位点突变导致外显子数目减少, 阅读框变短, 使得蛋白分子量相应变小。此外, BnMETI旁系同源基因在不同器官的表达模式随着进化和驯化较亲本白菜和甘蓝发生改变。由此可见, 油菜BnMETI基因表达模式的改变, 影响油菜各器官中BnMETI基因的剂量水平, 从而影响油菜器官的形成与油菜的生长发育。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|