科农9204是一个兼具高产和氮高效的候选小麦骨干亲本, 其遗传背景复杂, 携带冀麦38、小偃5号、绵阳75-18、小偃693和矮丰3号的遗传成分。利用221个PCR标记和89个DArT标记, 绘制了科农9204的全基因组基因型图谱。在2DL上, Xmag3596- Xmag4089区段与增加千粒重和籽粒含氮量的QTL紧密连锁; 在4BL上, Xcnl10与增加穗粒数、降低株高和穗茎长的QTL紧密连锁; 在6BS上, Xcnl113和 Xwmc756均与降低株高、穗茎长和穗下节间长的QTL紧密连锁。这些标记在科农9204衍生后代的传递率均为100.0%。利用已报道的关联性标记检测科农9204基因型在衍生后代的传递情况, 与增加穗粒数相关的1个优异等位基因位点在衍生后代中的传递率为71.6%; 与增加千粒重相关的4个优异等位基因位点的传递率均为100.0%; 与根部性状相关的4个基因位点中, 3个传递率为100.0%。这些与重要农艺性状相关位点, 科农9204基因型在其衍生后代中有很高的传递率, 在很大程度上与其对应的优异的农艺性状密不可分。科农9204染色体区段上存在的重要QTL可能是其成为候选骨干亲本的遗传基础。
Kenong 9204 (KN9204), a wheat cultivar with high yield potential and high nitrogen use efficiency (NUE), has a diverse genetic basis containing genetic materials of Jimai 38, Xiaoyan 5, Mianyang 75-18, Xiaoyan 693, and Aifeng 3. In this study, the genotypic map of KN9204 was released, which embraced 221 PCR-derived markers and 89 DArT markers. On chromosome 2DL, the region of Xmag3596- Xmag4089 harbored QTLs for increasing thousand-kernel weight and grain nitrogen content. On chromosome 4BL, Xcnl10 was close to the QTLs for increasing kernel number per spike and decreasing plant height and spike exsertion. On chromosome 6BS, Xcnl113and Xwmc756were closely linked with QTLs for decreasing plant height, spike exsertion and peduncle length. These markers had the transmissibility of 100.0% in the derivatives. The transmissibility of KN9204 elite genotypes was analyzed by known associated markers. The percentages of marker transmissibility from KN9204 to its derivates were 71.6% for one locus associated with kernel number per spike, 100.0% for four loci associated with thousand-kernel weight and 100.0% for three out of four loci associated with root traits. The high transmissibility of KN9204 genotypes on these loci might attribute to the excellent agronomic traits of KN9204. The important chromosomal regions harboring QTLs for elite agronomic traits are deduced to be the genetic basis of KN9204 serving as a candidate backbone parent.
在农作物新品种选育过程中, 一些优良作物品种(系)表现出良好的丰产性、抗病性和广适性, 其作为杂交亲本具有配合力高、优良性状遗传力强等优点, 并能衍生出许多具有重要应用价值的新品种(系), 这类材料被称为骨干亲本[1]。骨干亲本对其衍生品种的产量、品质、适应性、抗病性等方面具有较大贡献, 对于提高作物育种水平、促进作物品种的更新换代有重要作用, 因而从某种意义上讲, 作物育种历史也就是骨干亲本利用的历史, 对骨干亲本的遗传研究越来越引起育种家的重视。
利用分子标记解析重要染色体区段/位点的遗传效应可以揭示骨干亲本形成的遗传基础。张学勇等[2]研究发现, 我国黄淮海、西南冬麦区和西北春麦区的大部分骨干亲本携带矮秆基因Rht8。Christopher等[3]用SSR标记分析澳大利亚小麦Cook和大麦Triumph关键染色体区段在其衍生后代的遗传规律, 发现一些高频率选择位点与重要农艺性状相关。在中国小麦骨干亲本欧柔、胜利麦、碧蚂4号、矮孟牛、周8425B上也发现很多高频率选择位点与重要性状相关[4, 5, 6, 7, 8, 9]。徐鑫等[10]利用1B染色体上的17对SSR引物探讨小麦骨干亲本洛夫林10号中1BL/1RS染色体在14个代表性品种的遗传特征, 同时分析了洛夫林10号中1B染色体的重要区段在后代中的遗传特征。
科农9204由中国科学院遗传与发育生物学研究所农业资源研究中心, 以石6021 (冀麦38, 阿芙乐尔衍生后代)为母本, SA502 (八倍体小偃麦× 普通小麦杂种后代)为父本杂交选育而成, 于2003年通过国家农作物品种审定, 属半冬性、中早熟品种, 遗传基础丰富, 耐瘠薄, 氮肥利用效率高[11, 12, 13, 14]。据不完全统计, 利用科农9204为亲本已培育出4个小麦新品种(科农1093、科农199、科农1006和冀5265), 而且还有14个新品系正在参加国家或河北省冬小麦区域试验, 其中科农2009已经完成河北省生产试验(待审)。主要衍生品种或品系的系谱见附图1。4个科农9204衍生品种中, 科农1093、科农199、科农1006由本课题组选育, 冀5265由河北省农林科学院粮油作物研究所选育。其中, 科农1093为强筋小麦品种, 科农199、冀5265通过国家审定。
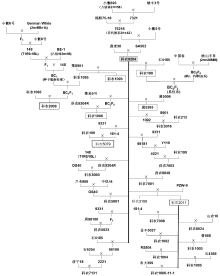 | 附图1 科农9204及其衍生后代系谱 虚框表示正在参加区域试验或生产试验的品系; 实框表示已经审定的品种。Supplementary Fig. 1 Pedigrees of Kenong 9204 and its derivatives Contents of the text boxes with dashed borders indicate the lines that are under regional trial. The text boxes with the solid borders describe the varieties that have been authorized. |
本实验室已经完成科农9204和京411的RIL群体(KJ-RIL)分子标记遗传连锁图谱的构建[15]和8个环境下产量、氮效率相关性状的QTL分析(见附表1)。本研究利用均匀分布于KJ-RIL遗传连锁图谱上的分子标记对科农9204原始亲本进行基因型值分析, 绘制科农9204全基因组基因型图谱。结合KJ-RIL群体QTL检测结果及前人报道的关联性标记, 追踪科农9204关键染色体区段在其衍生后代中的传递规律。
| 附表1 本实验室在2DL、4AL、4BL、6BS上发现的重要染色体区段和QTL簇 Supplementary Table 1 QTL clusters on chromosomes 2DL, 4AL, 4BL, and 6BS identified by our laboratory |
科农9204的5个原始亲本(矮丰3号、小偃693、绵阳75-18、冀麦38和小偃5号), 由中国农业科学院作物科学研究所小麦种质资源课题组李立会研究员提供, 科农9204及其611个衍生后代(见附表2)由本课题组保存和创制, 其中574个衍生系用于关键染色体区段的传递分析, 37个衍生系用于聚类分析。
选用221对均匀分布于KJ-RIL遗传连锁图谱的G-SSR、EST-SSR和STS引物进行PCR扩增, G-SSR和STS引物序列及其相关信息从GrainGenes网站(http://wheat.pw.usda.gov/)获得, EST-SSR引物序列参考Cui等[16]的文章。利用已报道的关联标记[17, 18, 19, 20, 21, 22, 23, 24, 25, 26], 对科农9204及其574个衍生系进行扫描。所有的引物均由生工生物工程(上海)股份有限公司合成。
采用SDS-酚法[27]从植株幼叶中提取DNA。PCR总体积为25 μ L, 含2 mmol L-1 MgCl2, 1× PCR buffer, 200 mmol L-1dNTPs, 1 U TaqDNA聚合酶, 正、反向引物各10 ng (25 ng μ L-1), 模板DNA为90 ng。在PCR Thermal Cycler (Applied Biosystems, 基因有限公司)中进行降落PCR (Touchdown PCR)。反应程序为94℃变性4 min, 15个循环的复性温度降落程序, 每个循环为94℃变性45 s, 65℃复性50 s (每循环降低1℃), 72℃延伸55 s; 30个循环的普通PCR, 即94℃变性40 s, 50℃复性40 s, 72℃延伸40 s; 最后72℃延伸5 min, 并于15℃保存扩增产物。将扩增产物加入适量的溴酚蓝(产物和溴酚蓝体积比为5∶ 1)混匀, 在6%聚丙烯酰胺非变性凝胶(39∶ 1)上稳压120 V电泳, 银染显色, 然后用凝胶成像系统(基因有限公司)照相观察。
参照https://www.triticarte.com.au/cgi-bin/order/login.pl公布的方法提取DNA。DArT标记分析数据由澳大利亚Diversity Arrays Technology Pty Ltd.提供。
PCR扩增结果按0-1赋值, 每对引物在相同迁移率处有带记为1, 无带记为0; DArT标记按照杂交信号强弱或有无记为0或1。分子标记结果最后形成0、1矩阵, 利用NTSYSpc2.10e软件计算遗传相似性系数, 用UPGMA法进行聚类分析。
结合KJ-RIL分子标记遗传连锁图谱[15], 选择均匀分布于全基因组的221个PCR标记(包括G-SSR、EST-SSR和STS标记)和89个DArT标记对科农9204及5个原始亲本进行基因型扫描, 绘制科农9204基因型图谱。将科农9204与矮丰3号、小偃693、绵阳75-18、冀麦38和小偃5号相同的基因位点依次记为1、2、3、4和5, 与5个亲本均不同的基因位点记为6, 与2个或2个以上亲本相同的基因位点记为7。以上基因位点用不同颜色表示, 利用GGT2.0绘制科农9204染色体基因型图谱。
221个PCR标记和89个DArT标记分析结果表明, 科农9204及其5个原始亲本、37个衍生后代共43份材料之间的相似系数在0.50~1.00之间; 在0.75水平, 大部分材料被划入7个类群(图1), 并且聚类结果与系谱关系基本一致。由科农5001的5个系选姊妹系与科农9204、科农9204S及科农9204的原始亲本冀麦38归为类群I; 普通小麦-冰草-偃麦草杂交后代系选科农3106和科农2011的5个系选姊妹系归为类群II; 科农199的2个姊妹系归为类群III; OS40与科农9204R杂交衍生的科农5003的5个系选姊妹系归为类群IV; 科农9204与西农9848杂交衍生的科农7001的4个系选姊妹系归为类群V; 科农5023的4个系选姊妹系归为类群VI; 含有黑麦品种German White的1R染色质的科农1006和科农2009, 与小麦-黑麦(German White)杂交后代选系9204R归类群VII。
结合KJ-RIL分子标记遗传连锁图谱, 选择均匀分布于全基因组的221个PCR标记和89个DArT标记, 绘制了科农9204基因型图谱(图2)。在全基因组水平, 科农9204染色体遗传物质构成中, 34.5%来自冀麦38, 24.3%来自小偃5号, 10.3%来自绵阳75-18, 8.6%来自小偃693, 而矮丰3号的遗传物质仅占2.1%。另外, 4.9%的染色体区段与5个原始亲本均不一样, 为等位变异片段; 15.2%的染色体区段与2个以上亲本相同, 暂且无法确定其亲本来源。
根据8个环境下KJ-RIL群体的QTL检测结果, 发现在2DL、4AL、4BL和6BS染色体区段上存在与产量、氮效率性状相关的重要QTL簇(见附表1), 利用这些区段上的标记对科农9204及其574个衍生后代进行基因型值扫描。在2DL上共发现7个重要标记, 其中区段Xmag3596-XIn10附近存在增加千粒重(QTkw)和籽粒含氮量的QTL (QGnc), 其在衍生后代中的传递率为100.0%。4AL上共有2个重要标记, 其中Xgpw2331附近存在降低穗下节间长的QTL (QPl), 其在衍生后代中的传递率为45.6%; Xgpw7543附近存在增加千粒重和缩短抽穗期的QTL (QHd), 其在衍生后代中的传递率为70.7%。4BL上共有5个重要标记, 其中Xcnl10附近存在增加穗粒数(QKnps)、降低千粒重及株高(QPh)和穗茎长(QSe)的QTL, 其在衍生后代中的传递率为100.0%。6BS上共有3个重要标记, 其中区段Xcnl113-Xwmc756附近存在降低株高、穗茎长和穗下节间长的QTL, 其在衍生后代中的传递率为100.0% (表1)。
| 表1 重要染色体区段在衍生后代中的传递 Table 1 Transmission of important chromosomal regions in derivatives |
利用前人报道的关联性标记, 对科农9204及其574个衍生后代进行扫描, 科农9204扩增片段大小及其在后代中的传递见表2。在Xgwm131位点上, 科农9204没有扩增出优异等位基因片段, 其在衍生后代中的传递率仅为32.6%; 在Xcfe273位点上存在增加穗粒数的优异等位基因, 其在衍生后代中的传递率为71.6%; 在Xwmc17、Xgwm234、Xcfa2257和Xcfa2234位点上存在增加千粒重的优异等位基因, 其在衍生后代中的传递率均为100.0%; 在Xgwm332和Xgwm294位点上存在与茎长和株高相关的等位基因, 科农9204基因型在衍生后代中的传递率分别为100.0%和69.0%。在Xcfa2257和Xcfa2040上存在与根部性状相关的等位基因, 科农9204基因型在衍生后代中的传递率为100.0%。
| 表2 关联性标记及其在衍生后代中的传递 Table 2 Transmission of associated markers in derivatives |
骨干亲本的形成是控制多个优良性状基因的组合及优化和协调表达的综合结果。冀麦38、小偃5号、绵阳75-18、小偃693和矮丰3号5个亲本的遗传物质在科农9204中的传递比例分别为34.5%、24.3%、10.3%、8.6%和2.1%, 与理论值有所偏差, 主要是人工选择的结果, 因为在实际的育种过程中, 为了得到理想的育种材料, 育种家会对感兴趣的性状优先选择, 从而使某些优良基因聚合, 并传递到杂交后代中。如在2DL存在提高千粒重的QTL, 来源于冀麦38和绵阳75-18; 在6BS存在降低株高的QTL, 来源于冀麦38和矮丰3号。这些增效染色体区段不仅被科农9204继承, 而且在其衍生后代中也表现较高的传递率, 说明这些染色体区段在育种过程中被强烈选择。
用骨干亲本之所以能选出众多优良品种, 除其本身具有优良性状基础外, 控制其重要农艺性状的基因组区段必须具有较强的遗传传递能力。本研究中, 科农9204的4个重要染色体区段上存在大量与产量、氮效率性状相关的QTL, 如千粒重、穗粒数和籽粒含氮量的增效QTL及降低株高的QTL等, 且这些QTL区段在衍生后代中优先传递, 是科农9204作为直接和间接亲本发挥作用的重要遗传基础。
科农9204的重要区段在衍生后代中的传递表明, 距离QTL位点越近的标记在后代中的传递率越高, 越向两侧传递率越低[28], 如2DL区段中Xmag3596、Xbarc228和XIn10与千粒重相关的QTL紧密连锁, 其在后代中的传递率均为100.0%, 距离该QTL 1.8 cM的Xmag3947在后代中的传递率为95.6%, 距离该QTL 4.1 cM和9.1 cM的Xswes61和Xcfd233传递率更低, 分别为82.9%和62.5% (表1)。4BL区段与株高等QTL紧密连锁的标记Xcnl10在衍生后代中的传递率为100.0%, 距离QTL较远的标记Xmag2055和Xmag4087的传递率则相对较低(表1)。在4AL区段, Xgpw2331附近存在降低穗下节间长的QTL, Xgpw7543附近存在增加千粒重和缩短抽穗期的QTL, 在衍生后代中的传递率都不高, 可能是由于这两个标记与QTL之间的距离太远, 该区段还需要进一步加密, 找到与该QTL紧密连锁的分子标记。以上结果与张学勇等[2]所述的选择牵连效应相符, 即育种家在选择优异性状时利用优异等位基因, 同时造成这些基因附近区域的遗传多样性降低, 即连锁不平衡。
Shoemaker等[29]认为植物育种实际上反映了育种家对农艺性状表型的“ 操纵” , 高产、抗病和优质等性状一直受到强烈选择。肖永贵等[9]对周8425B的研究表明, 与农艺或抗/耐环境胁迫的表型存在关联的遗传位点/片段在育种过程中得到强烈选择。李俊等[30]研究表明, 在利用川6415选育小麦新品种川麦42的过程中, 与每平方米穗数、产量、每穗粒重等目标性状相关的染色体区段被强烈选择而以单元型的形式保留下来。本研究中, 与优异等位基因紧密连锁的标记位点/区段在衍生后代中传递率均较高, 而与非优异等位基因连锁的位点Xgwm131-3B, 在衍生后代中的传递率仅为32.6%, 其余67.4%的材料均含有优异等位基因。上述结果表明, 在育种过程中我们一方面对重要农艺性状的优异等位基因片段进行强烈选择, 同时对非优异等位基因位点进行改良。
绘制了科农9204全基因组基因型图谱, 揭示了其重要染色体区段/位点在衍生后代中的传递规律: 与重要QTL紧密连锁的标记在衍生后代中的传递率为100.0%, 越往两侧传递率越低。优异等位基因在衍生后代中有较高的传递率。上述重要染色体区段/位点可能是科农9204成为候选骨干亲本的重要遗传基础。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|




