第一作者联系方式: E-mail: cricaas@163.com
WRKY转录因子广泛参与植物抗逆胁迫反应, 全面分析挖掘小麦A染色体组供体物种乌拉尔图小麦中的WRKY转录因子, 对进一步挖掘分析六倍体小麦WRKY转录因子的分子功能具有重要意义。本研究通过生物信息学分析, 在乌拉尔图小麦基因组数据中鉴定得到62条全长WRKY转录因子, 其中14条能被准确定位在染色体上, 并发现2对WRKY转录因子发生了复制; 通过进化树分析, 发现62条WRKY转录因子被分为8个小亚族, 且小亚族内部的基因结构相对保守。通过荧光定量分析所选基因在非生物胁迫下的表达模式, 筛选到2条在不同非生物胁迫下均上调表达的WRKY转录因子, 为进一步分析其功能打下基础。
WRKY transcription factors have been found to be involved in the processes responding to various abiotic and biotic stresses in plants. The knowledge on WRKY transcription factors in Triticum urartu (AA) will facilitate the function study of WRKY transcription factors in hexaploid wheat ( Triticum aestivum, AABBDD). In this study, 62 WRKY transcription factors with full length of coding sequence (CDS) were selected from Triticum urartu genome through bioinformatic analyses, in which 14 could be located on specific chromosomes and two pairs had been duplicated. These WRKY transcription factors were divided into eight subgroups by phylogenetic analysis and the exon-intron structure in individual subgroups was relatively conserved. Two WRKY transcription factors were selected for function validation, and their expressions consistently increased under diverse abiotic stresses by qRT-PCR assay.
转录因子是一类重要的调控因子, 可通过与目标基因启动子特定序列结合, 激活或抑制靶基因的转录表达, 从而调控生命活动进程。WRKY转录因子含有一段大约60个氨基酸的WRKY保守结构域, 在其结构域N-末端具有高度保守的WRKYGQK氨基酸序列, 其基因家族可分为3类, 其中I类有两个WRKY结构域, 且有CX4-5CX22-23HX1H的锌指结构; II类具有一个WRKY结构域和同I类的锌指结构; III类具有一个WRKY结构域和CX7CX23HX1C的锌指结构。通过对不同物种WRKY转录因子的功能分析, 发现它们参与植物对非生物胁迫的应答、生物胁迫反应以及激素信号转导等各种生命活动。
1994年, Ishiguro等[1]首次在马铃薯中发现WRKY转录因子, 随后在拟南芥、水稻、大豆、棉花中均发现大量的WRKY转录因子并进行了进化分析与功能验证。Eulgem等[2]在拟南芥中首次挖掘了61条WRKY转录因子, 并将其命名为AtWRKY1至AtWRKY61; 随后, Yu等[3]在拟南芥中克隆了一个受病原体感染和水杨酸诱导的WRKY转录因子AtWRKY62; 之后, Dong等[4]通过对拟南芥基因组数据的查找并整合Eulgem等和Yu等数据, 在拟南芥中发现72条WRKY转录因子, 并分析了其进化和表达模式。Jiang和Deyholos[5, 6]通过对不同时间盐胁迫下拟南芥根组织的芯片分析, 发现AtWRKY25和AtWRKY33在盐胁迫下显著上调表达, 并且这2个基因在拟南芥中过表达的转基因株系耐盐能力明显提高。Zou等[7]发现AtWRKY34在拟南芥花粉粒中特异表达, 并可能通过CBF信号级联反应负调控拟南芥成熟花粉粒对低温的敏感性。同时, 在拟南芥对病原体侵染的应答调控以及调节种子发育等途径中, 均发现AtWRKY转录因子发挥重要作用[8, 9]。Xie等[10]首先发现81个水稻WRKY转录因子, 并发现OsWRKY24、51、71和72等可被脱落酸(ABA)诱导表达; 随后Ross等[11]分别在粳稻和籼稻中发现98条和102条WRKY转录因子, 并进行了详细的进化分析; Ramamoorthy等[12]整合前人的结果, 并以其为参照, 与粳稻基因组数据进行查找分析, 获得103条水稻WRKY转录因子, 发现其中54条OsWRKY转录因子在非生物胁迫、激素诱导下存在不同的表达模式。对水稻WRKY转录因子的分子功能研究发现, 等位基因OsWRKY45-1和OsWRKY45-2在ABA信号途径中发挥不同作用, OsWRKY45-1干扰的转基因株系对ABA的敏感性增强, 而OsWRKY45-2干扰株系对ABA的敏感性降低, 使耐盐性得到改善[13]。Wu等[14]对OsWRKY11研究发现, 其可被热处理诱导表达, 其转基因株系的耐热性和抗旱性显著增强。此外, 在水稻中还发现许多WRKY转录因子参与到调节植物抗病与激素信号转导途径之中[15, 16]。在大豆和棉花中也以拟南芥WRKY转录因子为参照, 分别得到133条和116条WRKY转录因子, 并对其进行了进化分析[17, 18]; 在Retama raetam[19]、Thlaspi caerulescens[20]和大麦[21]等物种中也已发现诸多与非生物胁迫相关WRKY转录因子。
到目前为止, 在小麦核酸数据中(http://www. ncbi.nlm.nih.gov/nucleotide?term=txid4565[Organism])共发现76条WRKY转录因子, 40条具有全长CDS, 但只对其中少数进行了深入的功能分析[22, 23], 进一步深入挖掘小麦全长WRKY转录因子具有重要的意义。乌拉尔图小麦(Triticum urartu)是六倍体小麦A染色体组供体, 其基因组测序的完成为深入挖掘小麦WRKY转录因子序列提供了重要的数据资源[24]。本研究通过分析乌拉尔图小麦基因组数据, 深入挖掘其中全长WRKY转录因子序列, 并分析进化树、基因结构、染色体定位、基因复制和表达模式, 为进一步研究其分子功能打下基础。
根据文献报道, 分别从TAIR (The Arabidopsis Information Resource, http://www.arabidopsis.org/)和RGAP (Rice Genome Annotation Project, http://rice. plantbiology.msu.edu/)下载拟南芥和水稻WRKY转录因子的编码区序列(CDS)和蛋白序列[4, 12], 并根据Ling等[24]的报道, 下载乌拉尔图小麦基因组数据中的CDS和蛋白序列数据库。将拟南芥、水稻WRKY转录因子分别与乌拉尔图小麦的CDS和蛋白序列进行Blastn和Blastp比对, 进一步在乌拉尔图小麦基因数据中调取值小于0.001的相似序列。将所调取基因的蛋白序列分别在Pfam (http://pfam.sanger.ac. uk/search#tabview=tab1)和SMART (http://smart. embl-heidelberg.de/)平台进行多重序列比对和Hidden Markov Models (HMM) 查找确定WRKY转录因子[25, 26], 进一步手动去除非全长WRKY转录因子序列后, 即得乌拉尔图小麦全长WRKY转录因子。
根据保守结构域所在位置, 调取拟南芥、水稻和乌拉尔图小麦WRKY转录因子的保守结构域, 将所得保守结构域进行ClustalW分析, 并应用MEGA5.10构建进化树。
将所得WRKY转录因子进行Blastp比对分析, 当两个转录因子的覆盖度超过较长基因的70%、相似度大于70%, 且仅发生一次复制事件时, 认为该对基因发生了基因复制[27]。采用禾本科植物所应用的r = 6.5× 10-9计算复制年限[28], 计算公式为T = Ks/2r, 其中非同义突变频率(nonsynonymous, Ka)、同义突变频率(synonymous, Ks)通过PAL2NAL (http:// www.bork.embl.de/pal2nal/#RunP2N)计算获得[29]。
依据乌拉尔图小麦基因组的数据信息[24], 分析乌拉尔图小麦WRKY转录因子所在的染色体位置, 并通过MapInspect (http://www.plantbreeding.wur.nl/ uk/software_mapinspect.html)绘制染色体定位图。
根据基因组数据信息, 获得乌拉尔图小麦WRKY转录因子的DNA和CDS序列, 进一步将其通过GSDS (Gene Structure Display Server, http://gsds. cbi.pku.edu.cn/)分析WRKY转录因子的基因结构[30]。
1.4.1 材料及其处理 以六倍体小麦(Triticum aestivum)栽培种矮抗58为试验材料, 挑选饱满一致的种子, 将其在0.1% HgCl2溶液中消毒6 min, 用去离子水冲洗干净后, 摆放在铺有滤纸的培养皿中, 并在人工智能气候箱(18± 1℃, 相对湿度75%± 5%, 光照12 h)中培养至第2片幼叶萌发; 选取长势一致的幼苗转至Hoagland营养液, 分别进行多种胁迫处理。4℃低温处理6、12和24 h后取样; 营养液中分别加入5%、10%、15%和20% PEG模拟干旱胁迫, 处理24 h取样; 营养液中分别加入0.5%、1.0%、1.5%和2.0% NaCl进行盐胁迫, 处理24 h取样; 以正常生长的幼苗为对照。将各时间点样品的根和叶片组织迅速置液氮中, 保存在-80℃冰箱待用。
1.4.2 总RNA的提取和反转录 采用TRIzol试剂盒(TaKaRa)提取小麦根和叶组织的总RNA, 并采用PrimeScript RT reagent Kit (TaKaRa), 按说明书反转录合成cDNA。
1.4.3 引物设计和荧光定量 应用Oligo6在WRKY转录因子非保守结构域设计荧光定量引物(表1), 由生工生物工程(上海)有限公司合成。反应体系25 μ L, 包括12.5 μ L SYBR Green PCR Master Mix、9.5 μ L灭菌水、2 μ L cDNA、0.5 μ L正向引物、0.5 μ L反向引物。扩增程序为95℃预变性10 min; 95℃变性10 s, 60℃退火35 s, 72℃延伸35 s, 40个循环。每个样品3个重复。以18S为对照, 用2-Δ Δ Ct法计算相对表达量。
| 表1 荧光定量引物及其序列 Table 1 The primers for qRT-PCR |
在拟南芥和水稻中已分别报道了72条和103条WRKY转录因子[4, 12], 进一步在TAIR和RGAP中下载拟南芥和水稻WRKY转录因子的全长CDS和蛋白序列, 应用本地Blast, 将所得拟南芥和水稻WRKY转录因子的CDS和蛋白序列与乌拉尔图小麦基因组数据分别进行相似性比对, 获得相似序列。调取相似序列全长蛋白, 进一步经Pfam和SMART验证之后共获得78条WRKY转录因子, 去除非全长CDS序列之后, 最终获得62条乌拉尔图小麦全长WRKY (TuWRKY)转录因子, 其CDS长度为234~2163 bp, 编码蛋白长度在77~720个氨基酸之间, 等电点在4.84~5.30之间, 分子量为17 986.1~ 177 827.1 Da (表2)。
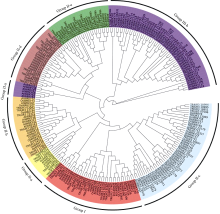
为了进一步分析所得TuWRKY转录因子的分子功能, 调取拟南芥、水稻和乌拉尔图小麦WRKY转录因子的保守结构域, 应用保守结构域序列构建进化树。结果显示, 62条TuWRKYs转录因子被分为Group I、Group II-a、Group II-b、Group II-c、Group II-d、Group II-e、Group III-a和Group III-b共8个亚族中, 其中Group I、Group II和Group III分别包含12、37和13条TuWRKY转录因子(图1)。
通过分析查找拟南芥和水稻WRKY转录因子的分子功能, 发现OsWRKY12、22、51、53以及AtWRKY40、47、58与植物抗病、防御等生物胁迫应答响应有关; OsWRKY72以及AtWRKY23、50、51、55、75与生长素、脱落酸等激素信号转导途径相关; OsWRKY11、90以及AtWRKY21、33、34与植物耐盐、抗旱等非生物胁迫应答响应相关。根据TuWRKY转录因子所属进化树位置, 推断TuWRKY转录因子TRIUR3_02376、06407、16802、23877、27575、28409、28514可能与生物胁迫密切相关; TuWRKY转录因子TRIUR3_06231、16697、23179、29978可能参与到激素信号转导、激素调控等生物学途径之中; TuWRKY转录因子TRIUR3_21369、22206、24481、35037可能与植物对非生物胁迫应答途径密切相关。
| 表2 本研究所用62个TuWRKY转录因子基本信息 Table 2 Details of 62 TuWRKYs used in this study |
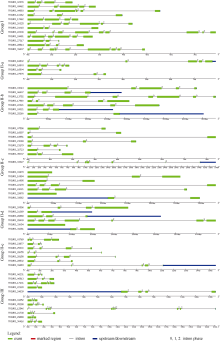
利用GSDS在线工具, 对62条TuWRKY转录因子的DNA序列及其CDS序列进行基因结构分析, 发现大部分TuWRKY转录因子具有2、3或4个内含子, 此外有3条TuWRKY转录因子分别具有0、5和6个内含子, 也有少数TuWRKY转录因子具有8或9个内含子(图2)。综合分析不同亚族TuWRKY转录因子的基因结构, 发现每个亚族TuWRKY转录因子的基因结构相对保守, 但其亚族内部基因结构也存在一定的差异。Group I中TuWRKY转录因子的基因结构差异相对较大, 其中分别有3条TuWRKY转录因子具有2、3和5个内含子, 1条具有4个内含子, 2条具有8个内含子。其他亚族中, 大部分TuWRKY转录因子具有4个以下的内含子, 但也有部分亚族内有少数TuWRKY转录因子具有9个或6个内含子。由此说明, 在物种进化过程中, 植物为了适应复杂的环境变化形成了内含子的多样性。
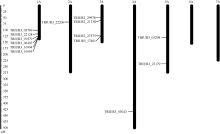
根据乌拉尔图小麦基因组测序数据, 查找每个TuWRKY转录因子所属染色体骨架在染色体中的位置, 结果显示, 只有14条能被准确地定位到染色体上, 1~5A染色体上分别有6、1、4、1和2条(图3)。在1A染色体底部有一个高密度TuWRKY转录因子局域, 进一步根据Hulob等[31]筛选基因簇标准, 发现在1A染色体底部和3A染色体底部各有一个基因簇, 分别是TRIUR3_10994、10995和TRIUR3_ 27575、17401。
根据Wei等[27]分析基因复制所选参数分析TuWRKY转录因子, 发现只有2对TuWRKY转录因子(TRIUR3_18951和TRIUR3_10995; TRIUR3_16802和TRIUR3_16803)发生了基因复制, 其中TRIUR3_16802和TRIUR3_16803位于同一个染色体骨架中发生了串联复制。2对基因的复制年限显示, TRIUR3_16802和TRIUR3_16803在54百万年前(million years ago, Mya)发生基因复制;
TRIUR3_18951和TRIUR3_10995在24 Mya发生基因复制。Ka/Ks比值是反映基因复制发生正向或纯化选择的一个指标, 当其比值大于1时表明基因在复制过程中发生了正向选择, 当其比值小于1时表明基因在复制过程中发生了纯化选择[32]。分析发现2对复制基因的Ka/Ks比值均小于1 (表3), 说明2对基因在复制过程中有害变异被净化, 从而保持了基因的功能。
| 表3 TuWRKY基因复制分析 Table 3 Analysis of TuWRKY gene duplication |
选取4条与非生物胁迫或ABA有关的TuWRKY转录因子, 验证其在不同程度低温、盐、干旱胁迫的小麦幼苗根和叶片组织中的表达模式。在低温胁迫下, TRIUR3_21369在小麦幼苗的根组织中显著上调表达, TRIUR3_16697、35037在小麦幼苗的根和叶片组织中均显著上调表达; 在盐胁迫下, TRIUR3_ 16697、22206在小麦幼苗的根组织中显著上调表达, TRIUR3_35037在小麦幼苗的根和叶片组织中均显著上调表达; 在干旱胁迫下, TRIUR3_35037在小麦幼苗的叶片组织中显著上调表达, TRIUR3_16697在小麦幼苗的根和叶片组织中均显著上调表达(图4)。结果表明, 这4条WRKY转录因子均与非生物胁迫密切相关。
WRKY转录因子是一类重要的调控因子, 广泛参与植物对非生物胁迫、生物胁迫的应答响应以及激素信号转导等各种生命活动, 并在其他许多物种中进行了深入的挖掘分析。Wu等[33]首次在小麦中克隆了15条小麦WRKY转录因子的cDNA序列, 并对其进行了的表达模式分析, 随后Niu等[23]以WRKY转录因子的保守结构域为参照与小麦的EST进行Blast比对, 并将相似序列拼接共获得43条小麦WRKY转录因子。综合他们的研究结果, 相对小麦庞大的基因组数据来说, 小麦WRKY转录因子的挖掘工作还远远不够。我们也进一步以拟南芥和水稻WRKY转录因子的CDS和蛋白序列为参照, 与小麦现有的EST和mRNA数据进行Blast比对分析, 共发现76条小麦WRKY转录因子, 其中只有40条具有全长CDS序列, 与其他物种相比小麦现已发现的WRKY转录因子的数量相对较少。
本研究第一次以最新测序完成的乌拉尔图小麦基因组数据为资源, 筛选到62条全长WRKY转录因子, 通过对其保守结构域分析发现, 其中11条基因具有两个WRKY保守结构域, 并且均属于Group I, 与拟南芥和大豆等物种相似[4, 17], 但分析OsWRKY转录因子发现具有2个WRKY保守结构 域的OsWRKY61和OsWRKY63属Group II-e, 表明Group I与Group II-e的WRKY转录因子较为接近。染色体定位信息是通过分析基因所属染色体骨架所在的染色体位置而获得的, 但通过分析发现许多单一的染色体骨架被定位到不同的染色体上[24], 例如scaffold81850被分别定位到1AS和3AL上, 这也使得仅有少数染色体骨架能被准确地定位到染色体上。
为了进一步验证所得WRKY转录因子的是否为新发现基因, 我们进一步将所得转录因子序列与小麦已知的全长WRKY转录因子进行比对分析, 发现其中仅有7条TuWRKY转录因子与小麦已知WRKY转录因子的覆盖度、相似度均达到90%以上, 表明本研究所分析序列大部分为新发现的WRKY转录因子, 可将其进一步在小麦中进行克隆分析验证。同时, 本研究通过对TRIUR3_16697等4条非生物胁迫相关WRKY转录因子的荧光定量分析发现, TRIUR3_16697和TRIUR3_35037在不同非生物胁迫下均能被诱导而上调表达, 表明其与非生物胁迫密切相关, 有进一步开展功能验证的价值。
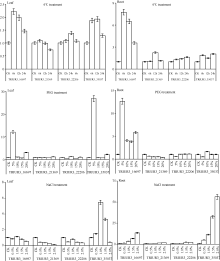 | 图4 4条非生物胁迫相关TuWRKY转录因子的荧光定量表达分析Fig. 4 qRT-PCR analysis of four abiotic-related TuWRKY transcription factors |
系统分析了小麦A染色体组供体乌拉尔图小麦基因组数据中的WRKY转录因子, 获得62条全长TuWRKY转录因子, 其中55条为新发现序列, 筛选到2条与非生物胁迫密切相关的TuWRKY转录因子。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|