第一作者联系方式: E-mail: huliqin_2012@163.com, Tel: 13051859528
非特异性磷脂酶C (NPC, nonspecific phospholipase C)水解磷脂酰胆碱或磷脂酰乙醇胺产生第二信使二酰甘油(DAG), NPC类基因在植物处于干旱、高盐、低磷等非生物胁迫条件下时发挥重要的作用, 并参与脱落酸(ABA)、油菜素内酯(BL)等激素信号传导途径。本研究以谷子品种龙谷25为试验材料, 通过序列比对, 克隆到一个新的NPC类基因, 命名为 SiNPC4。该基因被定位在谷子第8条染色体上, 编码区全长2877 bp, 由3个外显子和2个内含子组成, 编码512个氨基酸, 蛋白分子量为56.77 kD。系统进化树分析表明该基因位于NPC基因家族第3亚族, 保守结构域分析表明该蛋白含有保守的磷脂酶结构域和4个基序。亚细胞定位结果显示, SiNPC4蛋白被定位在细胞质、细胞膜、细胞核中。基因表达谱分析结果表明, 该基因主要在谷子根部表达, 并受干旱、盐、低温、黑暗、ABA、BL、赤霉素(GA)和茉莉酸甲酯(MeJA)等的诱导表达。将 SiNPC4基因转入拟南芥中, 转基因拟南芥与野生型拟南芥相比对ABA和BL激素的敏感性降低, 推测该基因可能作为负调控因子参与植物对ABA和BL激素的响应过程。在干旱和高盐处理下, 过表达植株与野生型植株长势没有明显差异。
Nonspecific phospholipase C (NPC) catalyzes the hydrolysis of phosphatidylcholine or phosphatidylethanolamine to generate diacylglycerol (DAG), which is an important second messenger in cells. NPC family plays a key role in response to some stresses such as drought, high salinity and phosphorus deficiency, and is involved in some hormone signaling pathways such as abscisic acid (ABA) and brassinolide (BL). Taking foxtail millet as material, we cloned a novel NPC gene named SiNPC4 by sequence alignment. This gene was detected to be located on chromosome 8. The full length of SiNPC4 was 2877 bp with three exons and two introns encoding 512 amino acid residues and the protein molecular weight was 56.77 kD. Phylogenetic analysis of NPC protein sequences indicated that SiNPC4 distributed to the third subfamily. The predicted protein structure of the gene contained conserved phosphoesterase domain and four motifs. The protein subcellular localization analysis revealed that SiNPC4 was localized in cytomembrane, cytoplasm, and nucleus. The gene expression profile results indicated SiNPC4 mainly expressed in root and was induced by drought, salt, cold, dark, ABA, BL, methyl jasmonate (MeJA), and gibberellic acid (GA) treatments. Arabidopsis carrying SiNPC4decreased the sensitivity to ABA and BL compared with WT. Thus, we deduced that SiNPC4 may act as a negative regulator in ABA and BL signaling pathways. Besides, there were no significant difference in growth between transgenic and wild type plants under drought and high salinity treatments.
磷脂酶是一类磷脂水解酶, 根据水解磷脂位点的不同主要分为3个家族(磷脂酶A、磷脂酶C和磷脂酶D)[1], 在响应植物生物和非生物胁迫信号转导途径中发挥重要作用[2, 3, 4]。其中, 磷脂酶C (PLC)依水解底物不同分为特异性水解磷脂酰肌醇磷脂酶C (PI-PLC)和非特异性磷脂酶C (NPC)[5]。NPC类磷脂酶是在植物中发现的与细菌PC-PLC同源的新型磷脂酶[6, 7], 拟南芥和水稻中分别有6个和5个NPC类磷脂酶基因[6, 8]。研究表明, NPC类基因在不同处理条件下的表达模式不同, 分别参与不同生长发育和逆境响应过程。在拟南芥中, AtNPC1在各个器官的表达量相当, AtNPC2和AtNPC6在角果中表达最高, AtNPC3主要在根部表达, AtNPC4主要在成熟的叶片和根部表达, AtNPC5主要在花序中表达。AtNPC4对ABA信号途径起正向调控作用[7], 在拟南芥中过表达AtNPC4植物对干旱和高盐胁迫的抗性增强[7, 9]。AtNPC3和AtNPC4受低磷胁迫诱导表达, 并且参与生长素(IAA)和油菜素内酯(BL)介导的根系生长发育过程[6, 10]。 AtNPC5在低盐胁迫下表达显著上调, 促进侧根生长[11]。在水稻中, OsNPC1在盐胁迫条件下表达上升, 低温条件下表达下降; OsNPC2和OsNPC4均受盐和干旱诱导表达; OsNPC5受低温和干旱诱导表达下调; OsNPC3表达不受干旱、低温和高盐胁迫的影响; OsNPC2、OsNPC4和OsNPC5在生殖发育阶段表达上调; 在种子发育的晚期, 只有OsNPC4表达持续增加; 圆锥花序阶段, 只有OsNPC1表达下调。此外, NPC类蛋白的亚细胞定位也不相同, AtNPC5定位在胞质[12], AtNPC4定位在质膜上[6], OsNPC1主要定位在细胞质, OsNPC3定位在叶绿体中[8]。
谷子具有抗旱、耐贫瘠、适应性广等特点[13], 是单子叶作物抗逆研究的理想材料之一。并且谷子基因组序列的公布为我们进一步解析谷子逆境应答分子机制创造了条件[14, 15]。目前, 关于谷子NPC家族的研究尚未见报道。本研究利用生物信息学方法从谷子基因组中获得5个NPC类基因, 对其基因结构、染色体分布、蛋白序列特征以及系统进化关系进行了系统分析, 并克隆到一个与拟南芥AtNPC4同源的基因SiNPC4。鉴于该基因受不同逆境胁迫和激素处理的诱导表达, 将其构建到过表达载体上转化拟南芥验证其功能。通过对过表达该基因的拟南芥植株对ABA、BL、PEG和NaCl等的响应, 探讨了SiNPC4在ABA等激素信号传导过程和逆境胁迫抗性中的作用。本研究为进一步阐明谷子NPC家族在其抗逆响应过程中发挥的作用提供了依据。
谷子品种龙谷25种子由中国农业科学院作物科学研究所刁现民研究员课题组提供, 拟南芥Columbia野生型(WT)种子、农杆菌GV3101菌株、163hGFP载体和带有CaMV35S启动子无GUS的pBI121载体由本实验保存。植物总RNA提取试剂盒、FastQuant RT Super Mix反转录试剂盒和Real Master Mix (SYBR Green)试剂盒购自天根生化公司。脱落酸(ABA)、油菜素内酯(BL)、赤霉素(GA)、茉莉酸甲酯(MeJA)、利福平和卡那霉素购自拜耳迪公司, DNA聚合酶PrimeSTAR HS DNA Polymerase、In-Fusion HD Cloning System试剂盒购于TaKaRa公司; 试验中使用的各种引物由北京奥科生物技术公司合成并由其完成各种测序工作。
在温度22℃, 相对湿度65%, 光照周期16 h/8 h的温室环境中培养谷子和拟南芥幼苗。基因表达谱分析参考霍冬英等[16]和闵东红等[17]的处理方法, 取三叶一心期长势一致的谷子幼苗分别进行干旱(6% PEG-6000)、NaCl (100 mmol L-1)、低温(4℃)、黑暗、ABA (100 μ mol L-1)、BL (100 μ mol L-1)、GA (100 μ mol L-1)和MeJA (100 μ mol L-1)等水培处理。除了黑暗处理在0 h和48 h取样以外, 其他均在处理后的0、1、6和24 h取整个植株, 所有样品置液氮速冻, -80℃保存备用。
参考已报道的拟南芥NPC4 (AT3G03530)的蛋白质序列(http://www.uniprot.org/), 在谷子基因组数据库(Phytozome v10.0.4)中进行BlastP比对, 搜索可能的谷子NPC家族成员; 用Pfam (http://pfam. xfam. org/)和InterPro (http://www.ebi.ac.uk/interpro/)数据库分析蛋白质的保守结构域, 用在线工具MEME (Multiple Em for Motif Elicitation)和ScanProsite tool (http://prosite.expasy.org/scanprosite/)分析蛋白质的motif序列, 用SignalP 4.1 Server (http:// www.cbs.dtu.dk/services/SignalP/)预测蛋白质的信号肽, 用Compute pI/Mw tool (http://expasy.org/ tools/)计算蛋白质的分子量和等电点, 用GSDS在线工具(http://gsds.cbi.pku.edu.cn/)分析基因结构, 利用ClustalX 2.0进行多序列比对, 并用软件MEGA 5.1以相邻法(Bootstrap设为1000)构建系统进化树。
采用植物总RNA提取试剂盒提取1.2中各样品以及谷子幼苗的根、茎和叶的总RNA, 按照FastQuant RT Super Mix反转录试剂盒说明书的要求合成cDNA。使用Real Master Mix (SYBR Green)试剂盒, 荧光定量PCR仪(ABI7500)进行Real-time PCR扩增。以SiActin (Si001873m.g)为内参(表1)。25 μ L反应体系含2× SuperReal PreMix Plus1 2.5 μ L, 正向引物和反向引物(10 μ mol L-1)各0.75 μ L, cDNA模板(约120 ng μ L-1) 1 μ L, 50× ROX Reference Dye 0.5 μ L, ddH2O 9.5 μ L。采用3步法反应程序, 95℃ 5 min; 95℃ 10 s, 60℃ 20 s, 72℃ 32 s, 40个循环。根据各样品特定荧光阈值下的Ct值, 采用2-Δ Δ Ct法计算基因在不同样品中的相对表达量
1.5SiNPC4基因克隆和表达载体构建
设计SiNPC4基因引物SiNPC4-F和SiNPC4-R (表1), 以谷子品种龙谷25的cDNA为模板扩增SiNPC4基因的完整编码框。采用In-Fusion重组酶技术将测序正确的目的基因分别构建到163hGFP和带有CaMV35S启动子的pBI121载体上(表1)。
以生长2周的谷子叶片为材料, 参考Yoo等[18]的方法进行原生质体制备和转化, 25℃培养16 h以后, 在激光共聚焦荧光显微镜(Zeiss LSM700)下观察蛋白亚细胞定位情况。
参考Clough等[19]的方法进行SiNPC4基因的拟南芥遗传转化, 将收获的T0代拟南芥种子种于含卡那霉素(50 mg L-1)的MS培养基上进行筛选, 获得2个转基因株系OE3和OE4, 经筛选和扩繁后, 选择纯合的T3代种子进行功能分析。
1.8SiNPC4转基因拟南芥的处理条件
将拟南芥野生型及转基因株系OE3和OE4种子经70%的无水乙醇处理3 min、1%的NaClO溶液浸泡15 min、ddH2O漂洗3次后, 点播于MS培养基上, 4℃春化3 d后于1.2中的环境下培养。参考Peters等[7]和Wimalasekera等[10]的处理方法, 将生长4 d长势一致的WT、OE3和OE4的幼苗移至分别含有25和35 μ mol L-1ABA、0.01和1.00 μ mol L-1BL、4%和6% PEG、100和150 mmol L-1 NaCl的MS培养基方皿上, 对照为MS培养基, 每个处理每个株系各放4棵幼苗。生长7 d后在EPSON Flatbed Scanner EPSON Expression 10000XL扫描仪上用WINRHIZO proLA2400软件对各种处理下的幼苗进行根系扫描, 测量根长和根系表面积, 采用单因素方差分析法统计分析数据, 用GraphPad Prism 5软件作图。
利用拟南芥AtNPC4保守区氨基酸序列对谷子基因组数据库进行BlastP比对, 获得5个谷子NPC类基因, 分别命名为SiNPC1-5 (表2), 5个NPC类基因之间核酸序列和基因结构变化较大, 其中, SiNPC2最长, 为4959 bp, 包含4个外显子; SiNPC3最短, 为1629 bp, 只有1个外显子(图1-A)。谷子NPC家族各成员间的氨基酸序列长度变化范围较小, 在512~546个氨基酸之间, 预测的平均分子量为58.39 kD, 最大的SiNPC1为61 kD, 最小的SiNPC4为56.77 kD。预测NPC家族各成员蛋白质的等电点在5.56~7.22之间。对这5个谷子NPC的氨基酸序列比对分析发现(图1-B), NPC3和NPC4相似度最高, 达到71.29%, 而其他蛋白序列间的相似度在47.09%~56.86%之间。蛋白结构域预测发现这5个NPC蛋白均含具有磷酯酶活性的结构域(Pfam PF04185), 并且结构域长度变化不大, 在359~370个氨基酸之间。蛋白基序分析表明SiNPC1-5没有如C2、XY、EF、PH、PX、ENTH、FYVE等已知的磷脂酶基序, 但含有4个与拟南芥NPC家族相同的保守基序, 即ENRSFD× × × G、T× PNR、DE× × G× × DHV和G× RVP× × × × × P。信号肽预测发现, SiNPC1、SiNPC2和 SiNPC5的N端含有信号肽。5个NPC类基因位于3条不同的染色体, SiNPC1和SiNPC3位于第9染色体, SiNPC2和SiNPC5位于第5染色体, SiNPC4单独位于第8染色体。
| 表1 SiNPC4基因克隆和Real-time PCR分析所用引物以及引物退火温度 Table 1 Primers used for SiNPC4 gene cloning and Real-time PCR analysis and annealing temperature of the primers |
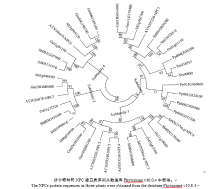
为了分析谷子NPC家族基因的进化关系, 选择单子叶植物水稻和高粱, 双子叶植物拟南芥、大豆和苜蓿以及苔藓类植物小立碗藓(Physcomitrella patens)和石松门类植物江南卷柏(Selaginella moellendorffii), 将其NPC类蛋白序列与谷子的NPC类蛋白序列进行多重序列比对, 构建系统发育树(图2)。结果表明植物NPC类蛋白家族被分为4个亚族, 分别被命名为Subfamily1、Subfamily2、Subfamily3和Subfamily4。在4个亚族中均有单子叶植物和双子叶植物的NPC类蛋白, 并趋向于形成独立的分支, 每个亚族至少含每个物种的一个NPC基因, 说明NPC家族基因在单子叶和双子叶植物分化之前就已经存在。苔藓和江南卷柏的基因只存在于亚族1中, 并且更趋向于彼此独立, 说明NPC基因家族的祖先可能来自亚族1, 而且苔藓和江南卷柏的NPC蛋白来源于不同的祖先。谷子NPC基因家族的5个成员在4个亚族中都有分布。SiNPC4与AtNPC4蛋白序列的相似度最高, 达57.83%。
| 表2 谷子NPC家族基因的基本信息 Table 2 Genes information of NPC family in foxtail millet |
构建了35S::SiNPC4-GFP载体, 将其转化到谷子叶片的原生质体中, 在共聚焦显微镜下观察SiNPC4-GFP融合蛋白亚细胞定位情况表明, 对照GFP蛋白在整个细胞中都可以表达(图3-A), SiNPC4-GFP融合蛋白定位在细胞质、细胞膜和细胞核中(图3-B)。qRT-PCR分析结果显示SiNPC4主要在根部表达, 而在茎和叶中表达量很低(图3-C), 表明SiNPC4可能主要在根部发挥功能。
利用qRT-PCR分别检测SiNPC4在各处理下的表达情况(图4), 在PEG和MeJA处理条件下, SiNPC4的表达量随着处理时间的延长而持续增加, 均在24 h达到最大, 分别为0 h的8.8倍和13.3倍; 与此相反, 经低温处理后SiNPC4的表达量随着处理时间的延长逐渐降低, 24 h的表达量是处理开始时的0.28倍; 在NaCl、ABA、BL和GA处理条件下, SiNPC4的表达呈现相似的变化趋势, 即在处理1 h时表达量最高, 随后下降, 在NaCl处理24 h时, 表达量又有所上升, ABA处理24 h的表达量低于0 h; 黑暗处理48 h后, SiNPC4的表达量明显上升, 达到处理开始时的30倍。
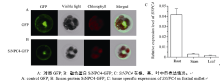 | 图3 SiNPC4蛋白亚细胞定位与在不同组织的表达情况Fig. 3 Subcellular localization and the tissue specific expression analysis of SiNPC4 |
为鉴定SiNPC4基因的功能, 构建pBI121- 35S::SiNPC4过表达载体, 遗传转化拟南芥获得2个纯合株系。正常生长条件下, 转基因株系苗期根系长度较WT短, 根系表面积较小, 而地上部生长与WT一致(图6-A), 表明该基因对根系的生长具有抑制作用。
2.5.1 转基因拟南芥植株在ABA处理下的表型
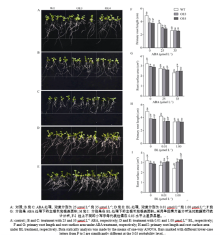
分析转SiNPC4基因拟南芥在种子萌发期和幼苗期对ABA的反应(图5)表明, 在不含ABA的培养基中, 转基因株系OE3、OE4和WT的萌发率基本一致, 在3 d内均达到了100% (图5-A, 5-D)。在2 μ mol L-1和4 μ mol L-1 ABA处理下, OE3和OE4的萌发速率显著高于WT, 到处理第6天时, OE3、OE4和WT的萌发率都达到100% (图5-B, C, E, F)。此外, 将在正常培养基上生长3 d、长势一致的OE3、OE4和WT幼苗分别移至MS、含25 μ mol L-1和35 μ mol L-1ABA的MS培养基中培养7 d (图6-A, B, C), 结果发现, 在MS条件下, OE3和OE4幼苗的主根长分别是WT的77.4%和76.1%, 显著短于WT (图6-A, F); 在25 μ mol L-1ABA处理下, 与MS相比, WT根长明显缩短, 减少了24.8%, OE3和OE4的根长分别缩短了20.0%和22.1% (图6-B, F); 在35 μ mol L-1ABA处理下, WT与OE3和OE4的根长差异不显著, 与MS相比, WT根长缩短了47.2%, 而OE3和OE4的根长分别缩短31.3%、31.7%, 说明OE3、OE4根长缩短比例较WT小(图6-C, F)。WT、OE3和OE4根系表面积与根长变化趋势一致, 随着ABA浓度升高表面积逐渐减少, 但是在35 μ mol L-1ABA处理下, OE3、OE4的根长与WT相当, 而根系表面积却大于WT (图6-G)。上述结果显示, 随着ABA处理浓度的增加, 转SiNPC4基因拟南芥萌发速率和苗期根系伸长生长速率受抑制程度较WT弱, 表明其对ABA的敏感性降低。
2.5.2 转基因拟南芥植株在BL处理后表型分析
BL在植物根系生长发育过程中具有重要的调控作用。用0.01 μ mol L-1和1.00 μ mol L-1BL处理WT、OE3和OE4后, 如图6-D、6-E所示, WT的主根随着BL含量的增加, 生长明显受到抑制, 在0.01 μ mol L-1和1.00 μ mol L-1 BL处理下, 主根长分别比正常生长条件下缩短了14.8%和32.6%; 而OE3和OE4的主根长略有降低, 但是不明显, 且在1.00 μ mol L-1BL处理下, OE3和OE4的主根长仅比正常生长时分别缩短了11.7%和11.6%, 缩短量显著低于WT (图6-H)。WT的根系表面积随着BL浓度的增加而减少, 相反, OE3和OE4的根系表面积随着BL浓度的增加而增加, 在1.00 μ mol L-1BL处理下, OE3和OE4主根长与WT的相当, 而根系表面积分别比WT高21.0%、26.7%, 说明处理条件下过表达株系的根系比WT发达(图6-I)。上述结果表明转SiNPC4基因株系对BL的敏感性相对低于WT。
2.5.3 转基因拟南芥植株的抗逆性分析 基于SiNPC4基因表达受PEG和NaCl诱导上调的特性, 为了验证SiNPC4能否提高植株的抗逆性, 对WT、OE3和OE4分别在PEG和NaCl胁迫条件下的表型进行分析, 结果表明过表达植株与WT在此两种胁迫下长势差异不明显(数据未显示), 即过表达株系未增强植株对PEG和NaCl胁迫的抗性。
 | 图5 35S::SiNPC4过表达拟南芥种子在不同ABA浓度处理下的萌发率Fig. 5 Seed germination rates of 35S::SiNPC4 transgenic lines under treatments with different concentrations of ABA |
非特异性磷脂酶C是一类在植物中新发现的磷脂酶, 其水解产物之一二酰甘油(DAG)是胞内重要的第二信使[1, 6]。DAG激酶(DGK)可磷酸化DAG产生磷脂酸(PA)信号[20, 21], 在植物响应生物和非生物胁迫及激素信号转导途径时具有重要的作用。谷子
具有耐旱、耐瘠薄的特性, 该作物中抗逆相关基因的研究还很少, 克隆该家族基因并解析其功能对谷子抗逆分子机制的研究具有重要意义。目前, 仅在拟南芥和水稻中分别鉴定出6个[6]和5个[8]该类基因成员, 本研究利用生物信息学方法在谷子基因组中筛选出5个NPC类基因, 系统进化分析表明该基因家族起源于单、双子叶植物分化之前, 且在单、双子叶植物分化后独立进化。从基因结构方面看, 这5个基因含有0~3个内含子(图1-A), 水稻的NPC类基因同样也含有0~3个内含子[8], 然而, 拟南芥的NPC基因家族根据内含子数目被分为两组, 分别含有2个和4个内含子[10], 说明谷子与水稻NPC基因家族具有类似的基因结构, 而不同于拟南芥, 这一现象可能与单、双子叶植物的分化相关。在蛋白质序列和结构方面, 谷子(图1-B)、水稻[8]、拟南芥[22]氨基酸序列长度无明显差别, 且均含有保守的磷脂酶结构域以及4个保守的基序, 表明NPC类蛋白结构在不同物种间是保守的。
本研究发现SiNPC4基因的启动子含有DRE、LTRE、MYB、MYC、WRKY等非生物胁迫相关的顺式作用元件, 以及ABRE、ARF等响应ABA、BL等植物激素的顺式作用元件, 其中, ARF经Nemhauser等[23]研究是应答BL的元件。表达谱分析表明在PEG、高盐、低温和黑暗等胁迫条件下, SiNPC4有不同程度的响应, 说明该基因在逆境胁迫应答中起重要作用; 在外源激素ABA、BL、GA和MeJA处理下, 该基因也表现出不同的表达模式(图4), 表明该基因在各种激素信号传导途径中也发挥不同的作用。
表达谱分析结果证明SiNPC4主要在根部表达。同时, 在正常条件下, SiNPC4过表达植株的根系与WT相比生长受到抑制, 表明SiNPC4基因可能参与植物根系生长发育的调控。BL对主根生长的促进或抑制作用依赖于其含量的变化[24]。本研究在外施0.01~1.00 μ mol L-1 BL时, WT主根长表现出对BL敏感的特性, 该结果与Mussig等[24]、Bao等[25]和Wimalasekera等[10]的研究结果一致, 而SiNPC4过表达植株主根长对BL的敏感性相对于WT降低, 这一表型与SiNPC4同源的拟南芥基因Atnpc4突变体对BL处理的表型一致[10], 说明SiNPC4和AtNPC4在BL信号途径中发挥的作用一致, 即负调控BL信号途径。另一方面, Belkhadir等[26]研究证明AtNPC4在BL受体和受BL调控的转录因子之间建立了桥梁, Nakamura等[27]研究发现BL可以诱导IAA5、IAA19和DR5的表达, Bao等[25]研究证明BL通过增加生长素向根尖的运输促进侧根生长发育。本研究中过表达植株的根系表面积比相应BL浓度处理下WT的根系表面积大, 推测SiNPC4在BL诱导下促进了侧根的生长, 而SiNPC4在根生长发育方面的作用机制还有待进一步研究。Peters等[7]的研究结果显示过表达拟南芥AtNPC4基因对ABA的敏感性增强, 该基因水解膜磷脂的产物DAG经磷酸化产生PA信号并正向调控ABA信号。而本研究中SiNPC4转基因拟南芥对外源ABA的敏感性降低, 推测SiNPC4在谷子中是ABA信号途径的负调控因子。这一结果与BL处理结果不同, SiNPC4与拟南芥中的同源基因AtNPC4在ABA信号途径中的作用方式完全不同, SiNPC4可能作为负调控因子参与植物对ABA的响应。这一差异可能与单子叶植物和双子叶植物的分化相关, 或者与谷子和拟南芥对于植物逆境反应的差异相关, SiNPC4在谷子中的具体调控机制还需要多方面的实验加以分析。SiNPC4受PGE和NaCl胁迫诱导表达上调, 但本研究发现过表达拟南芥植株在PGE和高盐胁迫条件下与WT长势表现一致, 并没有提高转基因植株的抗逆性, 推测该基因在PEG和NaCl胁迫响应中的功能可能需要其他调控因子的协同作用, 或者在不同的生长发育阶段对胁迫的响应程度不同。
从谷子中克隆了一个属于NPC家族的基因SiNPC4。SiNPC4蛋白被定位在细胞质、细胞膜和细胞核。该基因主要在根部表达, 并受多种处理如PEG、NaCl、低温、黑暗、ABA、BL、GA、MeJA等的诱导表达。过表达该基因拟南芥植株对ABA和BL的敏感性降低, 推测该基因可能作为负调控因子参与植物对ABA和BL激素的响应过程。在PEG和NaCl处理下, 过表达植株与野生型植株长势没有明显差异。该基因在谷子中的作用机制与其拟南芥同源基因AtNPC4的作用方式显著不同。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|