第一作者联系方式: E-mail: zhouyh@tricaas.com
低温胁迫是影响茶树产量、生长发育和地域分布的重要环境因子之一, 需要通过冷驯化的诱导来提高其抗寒性。DNA甲基化是植物表观遗传的重要方式, 环境胁迫会引起植物DNA甲基化状态的改变。为研究DNA甲基化是否参与茶树的低温响应, 本研究利用甲基化敏感扩增多态性技术(MSAP)和高效液相色谱法(HPLC), 分析了不同冷驯化阶段茶树基因组DNA甲基化水平及状态变化。MSAP分析结果表明, 50对选择性扩增引物在对照、驯化后和脱驯化样品中分别扩增出905、968和970个甲基化条带, 总甲基化位点比例分别为50.6%、54.1%和54.2%。与未驯化的样品相比, 冷驯化后和脱驯化的样品基因组DNA甲基化水平升高。HPLC的检测结果与MSAP结果类似。进一步分析甲基化模式发现, 与对照相比, 茶树冷驯化过程中同时发生了甲基化和去甲基化现象, 但总体变化趋势表现为甲基化水平的增加。表明茶树在抗寒响应中出现DNA甲基化现象。
Low temperature is one of the most critical environmental factors that limit tea plant growth, survival and geographical distribution. Tea plant can enhance its cold tolerance after undergoing a period of cold acclimation. DNA methylation is one of the epigenetic phenomena in plant, and can be altered by environmental stress. In order to explore the relationship between DNA methylation and low temperature stress response in tea plant, methylation sensitive amplified polymorphism (MSAP) and high performance liquid chromatography (HPLC) were used to analyze the changes of DNA methylation level and pattern in this study. The MSAP results showed that 905, 968, and 970 methylated bands were amplified with 50 selected primers in non-acclimated sample (CK), fully acclimated and de-acclimated samples, with the methylation levels of 50.6%, 54.1%, and 54.2%, respectively. DNA methylation levels in fully acclimated and de-acclimated samples were increased compared with CK. HPLC results were similar with MSAP results. In addition, DNA demethylation and methylation were both occurred during cold acclimation, but the DNA methylation level was increased more than the DNA demethylation level. These results indicated that DNA methylation may participate in the response of cold acclimation in tea plant.
茶树原产于热带及亚热带, 是一种喜温暖气候条件的叶用植物[1]。作为一种多年生植物, 茶树在其一生中要经历多次的冬季低温胁迫。而能否安全越冬是茶树能否存活以及茶叶种植北限的决定因素。另一方面, 春季的“ 倒春寒” 对茶芽的危害是影响茶叶生产效益的一种非常重要的气象因子, 轻者影响当年茶叶产量和品质, 重者导致茶树死亡。近年来, 随着全球气温的不稳定性, 预防气温特别是低温给茶树生长可能造成的伤害, 也愈来愈显示出其重要性。怎样提高茶树的抗寒性, 已成为关系茶叶能否正常生产的关键问题之一。因此研究茶树抗寒性的机制具有重要的意义。现有研究结果表明, 茶树的抗寒性需要通过一定时间和程度的低温驯化才能体现出来, 在经过冷驯化之后, 茶树在形态结构、生理生化以及基因表达等方面都发生一定的适应性变化, 从而能够忍受一定程度低温胁迫的影响[2, 3, 4, 5]。一些与茶树抗寒有关的基因也被分离出来[6, 7, 8], 然而对茶树表观修饰与抗寒之间关系的研究尚未见报道。
表观遗传主要是研究没有DNA序列变化的、可遗传的基因表达的改变[9]。许多研究结果表明, 表观遗传在植物生长发育和逆境胁迫响应过程中起着极其重要的作用, 其中DNA甲基化是表观遗传学的重要修饰形式之一[10]。DNA甲基化(DNA methylation)是指在DNA甲基转移酶的作用下, 将甲基基团添加到胞嘧啶的5位碳原子上, 而形成5-甲基胞嘧啶(5mC)的过程。DNA甲基化现象在高等植物中普遍存在, 但在不同植物种类中甲基化水平存在差异, 一般基因组DNA有30%~50%的胞嘧啶处于甲基化状态[11]。作为表观遗传的重要修饰途径之一, DNA甲基化与基因表达调控[12]、基因组印记[13]和转基因沉默[14]等生物学过程密切相关。
非生物胁迫或生物胁迫均会引起植物基因组DNA甲基化水平的改变。如潘雅姣等[15]发现水分胁迫导致水稻基因组约20%的CCGG位点发生胞嘧啶甲基化, 其中根部增加幅度尤为明显。Steward等[16]发现玉米幼苗在遭受低温胁迫时, 根组织基因组出现广泛的去甲基化。高桂珍等[17]发现油菜种子在热胁迫处理后, 基因组DNA甲基化水平表现出下降趋势, 同时有甲基化和去甲基化现象发生, 并以去甲基化为主。Robert等[18]发现将拟南芥暴露在致病或无害的丁香假单胞菌菌株下或用水杨酸处理后, 拟南芥基因组出现差异甲基化, 在防御基因附近表现出甲基化水平降低。
甲基化敏感扩增多态性(MSAP)技术是目前用于检测植物DNA甲基化的主要方法之一[19], 该技术是在扩增片段长度多态性(AFLP)技术的基础上建立的。采用对甲基化敏感的同裂酶HpaII和MspI分别与EcoR I组合切割基因组DNA。由于HpaII和MspI能够识别相同的序列(5° -CCGG-3° ), 但对甲基化的敏感性不同, 因此能够通过切割产生的片段反映出DNA序列中胞嘧啶的甲基化水平和状态, 能够在全基因组范围内进行甲基化位点的扫描, 是当前研究DNA甲基化水平和模式的重要方法之一[20, 21, 22, 23, 24]。高效液相色谱(HPLC)方法主要是通过核酸酶将DNA降解为单个核苷酸后进行定量分析, 该方法能够获得基因组胞嘧啶总体甲基化的程度[25]。本研究利用MSAP结合HPLC技术, 通过分析茶树在自然冷驯化过程中不同阶段叶片的DNA甲基化水平及模式变化, 研究冷驯化诱导茶树抗寒性过程中DNA甲基化与茶树抗寒性之间的关系, 探讨DNA甲基化参与茶树低温胁迫响应的可能分子机制, 以期为茶树性状改良以及抗逆性提高等方面提供一定理论依据。
试验材料为种植于中国农业科学院茶叶研究所国家茶树改良中心的20年生特早生国家级茶树品种龙井43。采用TIANGEN植物基因组快速提取试剂盒提取茶树基因组DNA。MSAP酶切所用到的EcoR I、Hpa II和Msp I以及HPLC酶解所用到的碱性磷酸酶均购自TaKaRa公司。HPLC分析所用的C18色谱柱(Supelcosil LC-18S)、标准品以及酶解所用的核酸酶购自Sigma公司。聚丙烯酰胺凝胶配制所用的尿素、N, N, N’ , N’ -四甲基乙二胺(TEMED)、过硫酸铵(APS)、丙烯酰胺以及N, N’ -亚双甲基丙烯酰胺均购于生工生物工程(上海)股份有限公司。
参照杨亚军等[3]的方法, 从2013年11月2日开始, 根据外界气温变化情况, 从10株茶树上采摘成熟度一致的成熟、完整、无病虫害的茶树叶片, 混合后分成2份, 一份用于电导率测定, 一份液氮速冻后于-80℃保存备用。根据电导率测定结果, 选取11月2日(驯化前)、12月19日(驯化后)和2014年2月20日(脱驯化) 3个时期的样品作为研究对象。
采用植物基因组快速提取试剂盒(TIANGEN)提取茶树基因组DNA, 采用NanoDrop ND-2000微量核酸蛋白测定仪(Pittsburgh, PA, USA)测定DNA浓度和纯度, 并用1%琼脂糖凝胶电泳检测其完整性, 于-20℃冰箱中保存备用。
参照Zhao等[26]的方法稍作修改进行MSAP分析。采用双酶切组合EcoR I/ Hpa II和EcoR I/Msp I进行酶切, 接头和扩增引物序列见表1。取700 ng DNA用2 U EcoR I在37℃下酶切3 h后65℃灭活30 min; 分别用0.5 U Hpa II和Msp I在37℃下酶切4 h后80℃灭活30 min; 将酶切产物用5 pmol EcoR I接头和50 pmol Hpa II-Msp I接头, 1 U T4连接酶在22℃连接3 h后于65℃灭活10 min。取连接产物4 μ L进行预扩增反应, 预扩增反应体系20 μ L, 包括连接产物4 μ L, 10× PCR buffer 2 μ L, 10 mmol L-1 dNTPs 1.5 μ L, MgCl2 1.2 μ L, 预扩增引物E+A和HM+T各1 μ L, 2.5 U rTaq酶。以94℃ 20 s, 56℃ 30 s, 72℃ 3 min, 循环35次。将预扩增产物稀释10倍后进行选择性扩增, 除引物为选择性扩增引物外, 反应体系同预扩增体系。PCR程序为94℃ 30 s, 65℃ (每个循环下降0.7℃) 30 s, 72℃ 1 min, 共13个循环; 接着94℃ 30 s, 56℃ 30 s, 72℃ 1 min, 共30个循环。将选择性扩增产物变性后进行变性聚丙烯酰胺凝胶电泳, 分离后经银染显色, 拍照保存。统计扩增条带, 分别用1和0代表扩增条带的有无。
参考潘雅姣等[15]的方法分析DNA甲基化水平和模式变化。根据Hpa II和Msp I能够识别并切割没有甲基化的5° -CCGG-3° 序列, 但却对胞嘧啶的不同甲基化状态敏感性不同的特性, 分析酶切片段能够反映酶切位点的甲基化状态以及基因组的甲基化程度。Hpa II能够切割单链内部或外部甲基化的胞嘧啶, 对全甲基化不敏感; 而Msp I能够切割内部全甲基化的胞嘧啶, 而对外部全甲基化的胞嘧啶不敏感。Hpa II和Msp I酶切产物的扩增条带有4种类型, 当两条泳道均有带时记为类型I, 表示没有发生甲基化; 当Hpa II有带而Msp I无带时记为类型II, 表示单链DNA发生外部甲基化; 当Hpa II无带而Msp I有带时记为类型III, 表示双链DNA发生内部甲基化; 当2条泳道均无带时记为类型IV, 表示双链DNA发生外部甲基化。
| 表1 MSAP所用的接头和引物序列 Table 1 Adapter and primer sequences for MSAP |
参照Chakrabarty等[27]报道的方法稍作修改进行HPLC分析。取约20 μ g DNA, 用ddH2O稀释后, 加入5 U核酸酶P1 (Sigma)和0.595 U碱性磷酸酶(TaKaRa), 最后用酶解消化液(30 mmol L-1 NaCH3CO2, 0.1 mmol L-1 ZnCl2, pH 5.3)补足到200 μ L。水解反应于37℃进行3 h后, 加入490 μ L无水乙醇终止反应。11 000 × g离心15 min后, 将上清液转移至新的离心管中, 冷冻干燥后用1 mL ddH2O溶解并过0.45 μ m滤膜后进行HPLC分析。HPLC在C18色谱柱上进行; 缓冲液为50 mmol L-1 KH2PO4, pH 3.5; 流速为0.8 mL min-1; 检测波长为285 nm; 时间为30 min。5mdC(%) = 100× [5mdC]/([5mdC]+[dC]), 式中[dC]和[5mdC]分别是2种形式dC的含量, 数值由标准曲线计算得出。
2.1.1 不同冷驯化阶段茶树DNA甲基化水平变化共筛选出50对能够稳定扩增的选择性扩增引物对冷驯化过程中3个时期的茶树叶片进行MSAP分析, 共扩增出1789条带, 每对引物组合扩增出的条带数为19~43条, 平均为36条。3个时期样品各扩增的甲基化条带数分别为 905、968和970, 甲基化比率为50.6%、54.1%和54.2%, 全甲基化条带数为478、471和475, 全甲基化比率为26.7%、26.3%和26.6% (表2)。
| 表2 不同时期茶树DNA甲基化水平变化 Table 2 Changes of DNA methylation level during different periods in tea plant |
由表2可知, 茶树叶片冷驯化后的样品(12.19) DNA甲基化水平(MSAP%)与对照相比升高, 由50.6%增加到54.1%。而在脱驯化(2.20)后, 甲基化水平与冷驯化样品大致相当, 并没有明显变化。上述结果表明低温胁迫能够引起茶树DNA甲基化水平的改变。全甲基化百分比在整个冷驯化过程中表现出先减后增的趋势, 在低温胁迫时期(冷驯化后)全甲基化水平最低, 说明甲基化水平升高的同时发生了一定程度的去甲基化现象, 但是相对于MSAP%来说, 变化不明显。
2.1.2 低温胁迫过程中甲基化状态变化情况 甲基化状态的变化主要是指甲基化带型的多态性变化, 即低温胁迫前后的甲基化模式发生改变。将统计的多态性带型分为2种类型, A类为去甲基化带型, 表明冷驯化后, 低温胁迫诱导茶树基因组DNA甲基化水平降低, 其中A1、A2、A3为完全去甲基化, 即所有被甲基化的胞嘧啶均发生去甲基化, A4、A5为部分去甲基化, 即部分被甲基化的胞嘧啶发生去甲基化; B类为甲基化带型, 表明低温胁迫导致茶树基因组DNA甲基化水平升高, 其中B1、B2为重新甲基化, 即胁迫前没有甲基化, 胁迫后出现甲基化位点, B3为超甲基化, 即在胁迫前甲基化位点的基础上, 胁迫后又有新的位点被甲基化(图1)。
由表3可以看出, 以冷驯化前样品作为对照, 冷驯化后样品(低温胁迫时)和脱驯化样品所扩增出的多态性条带数占扩增条带总数的百分比分别为5.4%和5.6%, 在扩增的多态性条带中A型条带所占百分比为33%和32%, B型条带占多态性条带总数百分比为67%和68%。上述结果表明, 冷驯化以后, 茶树基因组DNA同时发生甲基化和去甲基化, 但甲基化水平明显高于去甲基化, 因此, 冷驯化提高了茶树基因组DNA的甲基化水平。
根据5种标准品的含量及峰面积(图2), 获得各标准品所对应的标准曲线(表4)。各标准曲线的决定系数(R2)均大于0.999, 且标准品的出峰时间都有间隔, 保证各标准品均能有效分开。
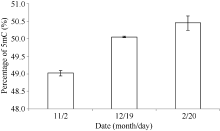
根据样品中胞嘧啶C和5-甲基胞嘧啶5mC的峰面积(图3), 通过所对应的标准曲线求得各自的含量, 得到不同时期的5-甲基胞嘧啶百分含量。冷驯化前后不同时期的样品5-甲基胞嘧啶的百分含量在49%~51%之间, 其中在低温胁迫前(未驯化样品)甲基化胞嘧啶含量最低, 而驯化后及脱驯化样品的甲基化程度较对照升高, 与MSAP的结果一致(图4)。HPLC的结果证实了MSAP结果的可靠性。
| 表3 低温胁迫过程中DNA甲基化状态变化情况 Table 3 Variation of DNA mathylation pattern during cold acclimation |
| 表4 各标准品的保留时间和标准曲线 Table 4 The retention time and standard curve of each standard nucleoside |
高等植物的DNA甲基化比例因植物种类和组织而异, 不同植物之间存在较大差异, 以低于50%为多[15, 28, 29]。本试验中利用MSAP技术和HPLC技术相结合, 首次检测了茶树的DNA甲基化水平, 超过49%, 高于水稻[15]、拟南芥[30](35%~43%)、棉花[29](41%)等所报道的DNA甲基化水平, 而与牡丹[31]、黑麦[32]{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #487}{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #487}{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #486}{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #486}{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #486}{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #464}{张勇, 邓科君, 张韬, 彭金华, 周建平, 任正隆, 2009 #464}等基因组甲基化水平接近, 反映了不同物种基因组间DNA甲基化水平存在差异。低温是影响植物生长和分布范围的重要环境因素, 研究表明, 植物的抗寒性是在一段时间的低温驯化基础上获得的, 这种抗寒性与植物本身的遗传性相关, 在低温驯化过程中某些与抗寒相关的基因发生变化从而对低温做出响应[33], 目前已在拟南芥[34]、小麦[35]等多个物种中得到证实, 茶树上也通过转录组测序等技术分离出一大批与冷驯化相关的基因, 这些基因在经过冷驯化后上调或下调表达[5]。基因表达的改变, 涉及许多调控机制, 其中DNA甲基化是一个调控基因功能的重要手段[29]。近年来已有许多关于低温胁迫与DNA甲基化的研究报道, 但不同物种的研究结果有一定差异, 表现出物种(品种)、组织等的特异性。Burn等[36]发现, 低温处理后, 拟南芥的甲基化程度降低, 而Zhang等[37]则发现, 草莓在低温诱导休眠过程中, 甲基化程度表现出明显的上升趋势。Pan等[38]对低温胁迫下水稻基因组DNA甲基化研究发现, 处于苗期的耐寒品种LTH只在根部检测到甲基化的明显升高, 而在圆锥花序则发生去甲基化; 处于孕穗期的低温敏感品种IR64, 低温胁迫后在叶片中表现出甲基化水平升高, 而圆锥花序则出现去甲基化。Mayer等[39]发现4个品种大麻在经过低温驯化后, 甲基化水平都表现出不同程度的提高, 但2个品种恢复到未驯化的水平, 1个品种仍然保持较高的甲基化水平, 另外1个品种的甲基化程度较对照降低, 表现出明显的品种特异性。本研究利用MSAP技术分析发现, 与对照(未驯化样品)相比, 冷驯化样品和脱驯化样品其基因组DNA同时发生甲基化和去甲基化, 但总体呈现甲基化水平增加趋势, 与Mayer等[39]的研究结果类似。这些甲基化或去甲基化位点可能参与冷驯化过程相关表达基因的调控, 从而参与冷驯化诱导茶树抗寒性的提高[5]。HPLC检测到的结果也证实了MSAP结果的可靠性。这些结果说明DNA甲基化与茶树的抗寒性密切相关。
DNA甲基化作为一种可遗传的遗传变异, 对基因的表达具有调控作用, 这种调控作用又可以在世代间稳定遗传。通过分析甲基化的作用位点序列信息, 可以鉴定出相关基因参与某种生命活动的可能功能, 从而为植物育种服务[40]。本研究发现了茶树在冷驯化过程中, 其DNA甲基化水平和模式都发生了变化, 证明DNA甲基化与茶树冷驯化诱导抗寒性有一定的关系。但是这种关系还需要结合具体基因的表达情况以及特定基因位点的甲基化变化情况, 才能得出更准确的结论。因此, 后续还需要对分离到的差异片段进行测序与功能验证, 为从表观遗传学方面揭示茶树冷驯化诱导抗寒性的机制提供参考, 也可以利用这些基因的信息, 开发抗寒相关的分子标记, 用于茶树抗寒育种的早期鉴定。
茶树在冷驯化的不同时期, 其基因组DNA同时发生甲基化或去甲基化, 并以甲基化为主。与冷驯化前相比, 茶树在驯化后DNA甲基化水平明显增加, 脱驯化阶段则维持较高的甲基化水平, 表明DNA甲基化与茶树冷驯化诱导抗寒性之间的确存在密切关系。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|