第一作者联系方式: E-mail: 819848239@qq.com
适当的株高不仅是抗倒性和机械化收获的需要, 而且是建立高产理想株型的需要。本研究测定来自世界油菜主产国的371份种质资源材料构成的自然群体的株高, 利用60K SNP芯片对该群体进行基因型检测, 获得了21 856个SNP, 利用这些SNP和株高数据进行全基因组关联分析, 结果表明: (1)检测到4个与株高显著关联的SNP, 即Bn-A07- p3583161、Bn-scaff_22790_1-p1271170、Bn-scaff_20735_1-p42779和Bn-scaff_18702_1-p589589, 分别分布于A07、C01和C02染色体; 它们对表型变异的解释率分别为11.33%、11.75%、12.31%和10.97%; (2)参考基因组信息, 在上述前3个SNP附近的492.0、9.5和69.5 kb处分别找到一个与株高性状相关的候选基因; (3) 4个显著SNP各自的等位基因间表型均值差异分别为11.09、15.12、10.48和8.93 cm, 双尾 t测验结果表明各等位基因组间差异显著。
Appropriate plant height is a trait related to lodging resistance and mechanical harvest, and ideal plant height is a basis of ideotype for high yield. The plant height of 371 rapeseed accessions collected from different growing regions in the world was measured in field experiments, all accessions were genotyped using a 60K SNP chip and 21856 informative SNPs were acquired, then the data of SNPs and plant height were used for genome-wide association analysis. Major results were as follows: 1) 4 significant SNPs related to plant height were detected, including Bn-A07-p3583161, Bn-scaff_22790_1-p1271170, Bn-scaff_ 20735_1-p42779, and Bn-scaff_18702_1-p589589, which located in A07, C01 and C02 chromosomes, and explained 11.33%, 11.75%, 12.31%, and 10.97% of phenotypic variation, respectively; 2) According to the reference genome annotation information, three candidate genes corresponding to the first three significant SNPs were predicted, each gene was located in 492.0, 9.5, and 69.5 kb away from the three SNPs, respectively ; 3) The significant (two-tailed t-test) difference of plant height between means of two allelic groups of accessions corresponding to each significant SNP was 11.09, 15.12, 10.48, and 8.93 cm, respectively.
甘蓝型油菜(Brassica napusL.)是3种油用油菜(甘蓝型油菜、白菜型油菜、芥菜型油菜)中籽粒产量最高的, 是我国重要的油料作物之一[1]。当前国际市场油菜籽供需矛盾尖锐, 加拿大和法国等主要油菜出口国都实现了从播种到收获的全程机械化, 生产成本低, 菜籽含油量也普遍比我国长江流域及黄淮冬油菜区高2~3个百分点, 低投入的生产和高含油量抵消了长途运输的成本, 严重冲击我国油菜产业的发展[2]。因此加快我国油菜育种进程, 提高我国油菜生产水平, 提升国际竞争力已迫在眉睫。
株高是油菜重要农艺性状之一, 与植株抗倒伏性有关[3], 据报道因倒伏造成的减产可高达10%~30% (严重时可达50%以上), 含油量亦比正常油菜低10%~30%[4]。随着甘蓝型油菜育种方向朝着杂交品种发展, 油菜株高都有所增高, 导致倒伏问题越来越严重。倒伏一方面会导致产量的严重损失, 另一方面也使机械化收获无法进行, 因此培育半矮秆品种在一定程度上既可以稳产保质, 又有利于实现机械化收获。理想株型育种正成为油菜育种重点之一, 其中确定适当的株高是理想株型的一个关键要素。
研究发现甘蓝型油菜株高的遗传比较复杂, Foisset等[5]用EMS诱变处理种子获得一个矮秆突变体, 遗传分析表明该突变体的矮秆性状受一对隐性基因(bzh/bzh)控制; 浦惠明等[6]通过对1个从澳大利亚引进的甘蓝型矮秆油菜矮源1号研究发现矮秆性状受1对显性基因控制, 显性度为0.66~0.80; 黄天带[7]对矮秆突变体DS-1遗传分析发现, DS-1的矮秆性状受一对不完全显性基因控制; 王宇[1]采用综合混合分布分析和模型适合性检验方法对2个群体的株高进行遗传分析发现株高遗传都受1对主基因控制; 张倩[8]的研究结果发现株高符合2对主基因控制的等显性模型, 这2个主基因的显性效应相等并且相应的加性效应也相等, 不存在上位性效应。这些结果的不同可能是材料及其遗传背景不同所致。目前对于甘蓝型油菜株高性状相关的QTL的定位研究发现株高QTL在除C08外的其他18条连锁群上都有分布, 但是分布频率不同, 其中在连锁群A03、A01、C06上定位到的次数最多[9, 10, 11, 12, 13, 14, 15, 16]。
关联分析(Association analysis)是在植物数量性状研究和植物育种中应用的一种较新的分析方法。它以连锁不平衡为基础鉴定某一群体内性状与遗传标记或候选基因间的关系。与重组群体比较, 它的显著优点是高通量, 即能在全基因组范围有效地检测大量的具有不同遗传背景的种质资源材料的性状控制基因位点或区域; 除高通量外, 关联分析还有花时间少的优点, 一般以现有的自然群体为材料, 无需构建专门的作图群体。同时, 精度高, 可达到单基因的水平[17]。目前全基因组关联分析已经成功应用于玉米[18]、水稻[19]、拟南芥[20]、油菜[21, 24]、大麦[22]等重要作物。本研究检测与株高性状显著关联的SNP, 旨在为油菜株高的分子育种提供有效的分子标记及克隆控制油菜株高相关基因提供有价值的信息和依据。
关联群体由来自不同育成年代、国内外遗传差异较大的371份甘蓝型油菜构成, 其中国内材料共296份, 主要来自湖北、湖南、陕西、江苏等地, 国外材料75份, 主要来自法国、澳大利亚、日本和瑞典等国家。
自然群体的SNP数据信息使用DNA与芯片杂交方法获得。将幼嫩叶片置10%乙醇中漂洗; 然后剪取0.1~0.2 g叶片放入碾钵中, 利用液氮快速碾磨至粉末状, 装入2 mL离心管中; 加入预热DNA提取液700 μ L; 混匀后置65℃水浴锅中1 h, 每10~15 min混匀1次; 加入700 μ L混合液(苯酚∶ 氯仿∶ 异戊醇=25∶ 24∶ 1), 轻轻颠倒混匀10 min; 室温下, 10 000× g离心15 min; 吸取上清液至新的2 mL离心管中; 加入等体积混合液(氯仿∶ 异戊醇=24∶ 1), 颠倒混匀, 静置5 min, 10 000× g, 离心15 min, 用枪吸取上清液到新的离心管中; 加入2倍体积无水乙醇, 混匀后在-20℃静置1 h, 10 000× g, 离心10 min, 弃上清液; 再加入500 μ L预冷的75%乙醇洗涤沉淀, 去上清液; 经过连续2次洗涤沉淀后, 晾干; 加入含有2% RNase A溶液100 μ L, 在37℃静置1 h后4℃过夜; 用等体积混合液(氯仿∶ 异戊醇=24∶ 1)再次抽提DNA溶液, 颠倒混匀, 静置10 min, 10 000× g, 离心15 min或20 min, 去除RNase A, 吸取上清液(约60 μ L), 再次离心, 1 min; 利用琼脂糖凝胶电泳(0.8%)和紫外分光光度计检测DNA浓度、质量及完整性; 确定所有DNA样品的吸光度260/280比值在1.8~2.0之间。群体SNP分型检测由北京怡美通德有限公司完成(http://bjemtd.cn.china.cn/)。
2013年在中国农业科学院油料作物研究所武汉阳逻试验基地进行试验, 试验点位于东经113° 41′ ~115° 05′ , 北纬29° 58′ ~31° 22′ , 属亚热带季风气候。田间试验按3个重复随机区组设计, 每个小区种植4行, 行距40 cm, 株距20 cm, 条播, 于五至七叶期定苗。其他同大田管理。待成熟收获时, 从各小区随机选取10株, 测定株高, 即子叶节至全株最高部位的长度, 以厘米(cm)为单位。用DPS处理系统[23]分析表型数据。
利用第一张甘蓝型油菜60K SNP芯片(IlluminaInc, San Diego, CA)对群体进行SNP分型[24]。使用Genomestudio软件进行SNP分析, 参考Fan等[25]和Yan等[26]方法对SNP数据进行评估, 最终挑选出高质量并且在基因组上位置唯一的21 856个SNP; 从21 856个SNP中挑取7286个均匀分布于染色体上的SNP用于群体结构分析(Structure 2.3.4[27]), 设参数为: length of burning period为10 000, MCMC reps为10 000, K=1~10, 重复5次。群体结构参考Evanno的方法[28]; 将SNPs数据调整为HapMap格式并导入Tassel 5.0 (http://www.maizegenetics. net/tassel)软件中计算亲缘关系, 将参试的371份材料分为3个类群, 亲缘关系处于中等偏下水平。在Tassel 5.0中用混合线性模型(MLM)方法以群体结构(Q)和亲缘关系(K)作为协变量对SNP和株高表型数据进行关联分析(显著水平为P< 4.58× 10-7)。使用Clone Manager 5对显著的SNP位点进行限制性酶切位点的扫描。在Microsoft Excel中使用t-test检测显著关联标记的等位基因表型差异显著性。
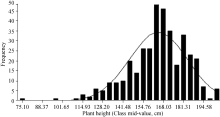
对由371份材料构成的关联群体的株高分析表明, 该群体株高性状表现出广泛的表型变异, 幅度为75.1~205.6 cm, 最大值为最小值的2.74倍; 均值为164.79 cm, 变异系数(CV)为11.58%; 正态性检验结果为W=0.9696, P=0.000 001, 表明该群体的株高表型符合正态分布; 3个重复之间的相关系数为0.91, 表明试验重复性被控制得较好(表1和图1)。
| 表1 株高表型的统计分析 Table 1 Statistical analysis of plant height (cm) |
使用混合线性模型(mixed liner model, MLM)中的Q+K模型来检测关联的SNP (Bonferroni校正的显著水平为P< 4.58× 10-07, 即P=0.01/21856; -lg (P) = 6.34)。共检测到4个与株高性状显著相关的SNP为Bn-A07-p3583161、Bn-scaff_22790_1-p1271170、Bn-scaff_20735_1-p42779和Bn-scaff_18702_1-p589589, 分别分布于A07、C01和C02染色体; 它们对表型变异的解释率分别为11.33%、11.75%、12.31%和10.97% (图2和表2)。
对这4个显著的SNP序列(序列为该SNP及其参考基因组的旁侧序列, 大小为200 bp左右)进行酶切位点 预测发现, 当Bn-scaff_22790_1-p1271170的SNP类型为CC时, 可以被限制性内切酶Apo I (R↓ AATTY, R: 任意嘌呤, Y: 任意嘧啶)识别并酶切(图3-A), SNP类型为AA时该位点则不能被识别、酶切; 当Bn-A07- p3583161的SNP类型为AA时也可以被Apo I识别、酶切(图3-B), 为GG时则不能。聚合这2个标记的高秆等位基因的材料有2个(辰二紫和78036), 它们的平均株高为178.93 cm, 而聚合矮秆等位基因的材料有339个(Ranger和华双5号等), 它们平均株高为163.89 cm。这些信息可以为株高的分子标记辅助选择育种提供有效的信息。
| 表2 与株高显著关联的SNP及候选基因 Table 2 Significantly associated SNP of plant height and candidate genes |
在全基因组关联分析结果中检测到4个与株高性状显著关联的SNP, 因此对这些显著关联的SNP进行等位基因表型差异分析(表2)结果表明, Bn-scaff_20735_1-p42779、Bn-scaff_22790_1-p1271170、Bn-A07-p3583161和Bn-scaff_ 18702_1-p589589这4个显著标记的等位基因间(每个显著标记在材料间有2种基因型)表型均值差为10.48、15.12、11.09和8.93 cm, 且双尾t测验结果表明每个标记对应的组间差异是显著的, 说明这4个显著标记的等位基因的差异与株高有关。
基于油菜参考基因组注释信息[29], 在Bn-A07-p3583161附近的492 kb处找到一个与株高相关的基因(GSBRNA2T 00038726001), 该基因在拟南芥中的同源基因(AT2G04240.2)的表达受盐和渗透胁迫诱导, 转录水平受转录因子DELLA蛋白(DELLA蛋白位于细胞核中, 在赤霉素信号转导中起阻遏作用)的诱导和赤霉素抑制, 并且参与脱落酸的代谢[30]; 在Bn-scaff_22790_1-p1271170附近的9.5 kb处找到一个与株高相关基因(GSBRNA2T0009 0741001, AT4G25000.1), 该基因参与碳水化合物代谢过程, 能在赤霉素和脱落酸的刺激下产生应答反应[31]; 距离Bn-scaff_20735_1-p42779标记69.5 kb处也找到一个与株高性状有关的基因(GSBRNA2T00137198001, AT1G72560.3), 该基因的突变会导致发芽后顶端分生组织和叶原基生长的延迟, 及植株从营养生长向生殖发育的延迟[32]。这些基因是否确实控制着株高还有待进一步进行功能验证。
株高是影响油菜产量和品质的重要农艺性状之一, 自20世纪90年代以来, 已有众多研究人员努力寻找油菜矮秆基因和开展相应的遗传学研究, 试图由此解决油菜生长后期的倒伏问题, 但目前在生产上大面积应用的推广品种中携带这些矮秆基因的很少, 一是因为可能存在不利基因的连锁累赘, 二是单基因控制体系较难将植株调整到一个最合适的高度, 因为株高往往和花序长度成正比[33], 因此培育高产、半矮秆油菜新品种是当前最重要的育种目标之一。本研究在A07、C01、C02这3条染色体上共检测到4个与株高表型显著关联的SNP (表2), 并成功在A07、C01和C02上的显著SNP附近找到与株高表型相关的候选基因。前人发表的株高QTL研究结果显示, 已定位到株高QTL分布在除C08外的其他18条连锁群上[9, 10, 11, 12, 13, 14, 15, 16], 定位到最多的QTL为A03 (25个次)、A01 (24个次)、C06 (24个次), 最少的为C05 (1个次)、A04 (2个次)、C01 (2个次)。根据已报到的QTL峰值附近的标记的序列与油菜基因组序列[29]比对, 直接获得每个QTL在基因组上的物理位置, 进而将本研究中的QTL与前人报道的比较, 发现A07 (10个次)和C01 (2个次)上的QTL都与本研究的A07和C01上显著性标记距离较远, 因此认为Bn-A07-p3583161和Bn-scaff_22790_1-p1271170为株高性状相关的新标记; 而Joshua等[14]在C02上发现的ph12.3的区间包含了本研究的显著性标记Bn-scaff_18702_ 1-p589589和Bn-scaff_20735_1-p42779, 且ph12.3的峰值附近标记与它们的距离分别为580 kb和1264 kb, 因此认为它们可能属于相同的QTL。并且其中的2个SNP (Bn-scaff_22790_1-p1271170和Bn-A07-p3583161)的不同等位基因类型存在限制性酶切位点的差异, 这些QTL信息和标记信息可以为油菜株高的分子遗传育种提供有效信息。
分子标记辅助选择(molecular-assisted selection, MAS)是指利用与特定性状相关联的分子标记作为辅助手段进行的育种。在基因的聚合和基因转移上的应用可以使得作物遗传改良的准确性及效率大大提高。比如国际水稻研究所Huang等[34]利用4个含有不同水稻白叶枯病抗性基因的近等基因系进行抗性基因聚合, 所产生的抗性基因累积的品系比含有单个抗性基因的品系材料具有更高的抗性水平和对病原菌更广的抗谱。在本研究中检测到的4个显著SNP的等位基因表型差异均为10.48、15.12、11.09和8.93 cm, 且组间双尾t测验结果都表明各自组间差异显著, 我们在群体中还发现了包含这4个显著SNP的全部高值等位基因的材料2个(辰二紫和78036), 他们的平均株高为178.93 cm, 含全部低值等位基因的材料8个(扬2008、青油4号、慈油1号、川油花叶、0011-1、21006、P11和Das-chosen), 他们的平均株高仅为154.31 cm。因此分子标记辅助选择和优异等位基因的聚集可以有效地进行作物的特定性状改良。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|