第一作者联系方式: 张天泉, E-mail: zhangt_q@163.com; 郭爽, E-mail: guoshuang16@163.com
叶色突变体是研究高等植物光合作用、叶绿素代谢途径、叶绿体结构与功能分子机制的理想材料。本研究从EMS (ethyl methane sulfonate)处理的缙恢10号( Oryza sativa L. ssp. indica)诱变群体中发现了一个苗期呈现黄绿色、抽穗期渐变为淡绿色的叶色突变体, 命名为 yellow green leaf 9( ygl9)。与野生型相比, ygl9苗期和分蘖期光合色素极显著降低, 抽穗期光合色素显著降低, 气孔长度、气孔导度和蒸腾速率极显著增加, 净光合速率无明显变化。透射电镜观察表明, ygl9的嗜锇小体增多、基粒模糊、基质片层减少且疏松, 但叶绿体结构基本完整。遗传分析显示该突变性状受1对隐性核基因调控。利用西农1A/ ygl9 F2群体中的759株隐性单株, 最终将 YGL9定位在第3染色体短臂SSR标记S03-1和InDel标记Ind03-19之间, 遗传距离分别为0.13 cM和0.07 cM, 物理距离为63 kb。本研究为 YGL9基因的克隆和功能分析奠定了基础。
Leaf color mutants are ideal materials in illuminating molecular mechanism of photosynthesis, chlorophyll metabolic pathway and chloroplast development. A novel mutant named yellow green leaf 9( ygl9) was isolated from the progeny of ethyl methane sulfonate (EMS) treated Jinhui10 ( Oryza sativa L. ssp. indica) and displayed yellow-green leaves at the seedling stage while light green at the heading stage. Compared with those of the wild type, the photosynthetic pigments of the ygl9 mutant reduced very significantly before the tillering stages and significantly in the heading stage. However, there was no obvious changing for net photosynthetic rate between the wild type and the mutant. The characteristics of stomata length, stomatal conductance and transpiration rate increased significantly in the ygl9. The observation by transmission electron microscope showed that the ygl9 mutant contained comparable chloroplasts with more osmiophilic granules, fuzzy grana and fewer/looser stroma lamella to the wild type. Genetic analysis suggested that the mutational trait was controlled by a single recessive gene. Using 759 mutational individuals from the F2 generation of Xinong 1A/ ygl9, the YGL9locus was finally mapped on the short arm of chromosome 3 between SSR marker S03-1 and InDel marker Ind03-19 with genetic distances of 0.13 cM and 0.07 cM respectively, and the physical distance was only 63 kb. These results provided a foundation for map-based cloning and functional analysis of YGL9gene.
叶色突变是自然界中比较常见的一种突变性状, 其主要特点是叶色变异, 表现不正常的绿色。叶色突变往往直接或间接影响叶绿素的生物合成或降解途径, 最终改变叶绿素含量, 从而引起叶色的变化。到目前为止, 在拟南芥[1]、水稻[2]、玉米[3]、油菜[4]、小麦[5]、大豆[6]等多种植物中均有叶色突变体的报道。叶色突变体在研究高等植物光合作用机制、叶绿素生物合成途径、叶绿体的结构功能和遗传发育调控机理等方面发挥着重要作用[7]。此外, 叶色标记在良种繁育中也具有重要的应用价值。
导致叶色变化的因素很多, 最主要的是基因突变。叶绿体发育相关途径、光合色素、血红素和光敏色素等代谢途径基因变异均可能引起叶色变化, 此外, 核糖核酸还原酶等基因的突变也可能导致叶色发育异常[8]。因此, 叶色变异是一个复杂的遗传性状。水稻是单子叶模式植物, 也是重要的粮食作物, 然而, 目前克隆的叶色调控基因还相对较少, 不利于禾本科作物的分子改良。在水稻中, OsHAP3A、OsHAP3B和OsHAP3C为叶绿体发育的调控基因, 控制细胞核编码的相关基因在叶绿体中的表达, 以此调节叶绿体的发育[9]; OsCAO1、OsCAO2和CBL均编码叶绿素a加氧酶, 是催化叶绿素a合成叶绿素b的关键酶[10]; OsCHLH、OsCHLD/CHL1和OsCHLI/CHL9编码Mg-原卟啉IX螯合酶 (Mg- chelatase)的3个亚基ChlH、ChlD和ChlI, 能促进Mg2+插入原卟啉IX, 形成叶绿素和亚铁血红素的前体[11, 12]; OsDVR编码联乙烯还原酶, 催化联乙烯叶绿素(酸酯) a转化为单乙烯叶绿素(酸酯) a[13]; VYL编码锌指结构的精氨酸甲基转移酶互作蛋白功能域AIRl, 在调节核基因的表达和信号传导中起重要作用[14]; YGL1编码叶绿素合酶, 是叶绿素a生物合成最后一步反应的催化酶, 催化叶绿素酸酯a转化为叶绿素a[15]; YGL2编码一个heme oxygenase (HO1)血红素加氧酶, 是催化血红素降解的起始酶和限速酶[16]。
我们利用EMS诱变优良恢复系缙恢10号, 获得了一个从苗期开始叶片即为黄绿色, 抽穗期叶色转为淡绿的突变体ygl9, 该突变性状受1对隐性核基因控制。本研究将其定位在第3染色体短臂端63 kb的区间内, 该区间内并未发现已报道的与叶色相关的基因, 表明它是一个新的黄绿叶突变体, 该结果为YGL9基因的图位克隆和功能分析奠定了基础。
ygl9来源于经化学诱变剂EMS处理的恢复系缙恢10号, 经过多代自交观察, 其黄绿叶表型遗传稳定。2013年, 将叶色表型正常不育系西农1A与ygl9杂交, 收获F1种子。同年, 于海南种植F1并收获F2种子。2014年, 在西南大学水稻研究所研究基地种植亲本、F2群体, 从苗期开始观察叶色表型。在同一块实验田相邻种植野生型缙恢10号和突变体ygl9各40株, 成熟后分别调查小区中间10株的株高、穗长、有效穗数、每穗粒数、每穗实粒数、结实率、千粒重等农艺性状。
苗期、分蘖期和抽穗期, 分别随机选取5株长势一致的野生型和突变体单株, 参照Lichtenthaler[17]描述的方法测定叶片的光合色素含量。即将新鲜叶片中部去除中脉后, 称取0.1 g, 剪碎浸泡在丙酮∶ 无水乙醇(1∶ 1, v/v)混合液中, 定容至25 mL, 密封黑暗处理24~48 h, 期间轻轻震荡数次, 使光合色素浸提充分。用Beckman22S型分光光度计测量提取液在663 nm、645 nm、470 nm 3个波长下的吸光值, 重复3次, 计算光合色素的含量。
水稻孕穗期, 在晴天上午9:00— 11:00, 选择长势一致的野生型和突变体单株各5株, 利用LI-6400型便携式光合作用测定仪测定净光合速率、气孔导度、胞间CO2浓度和蒸腾速率等光合特性, 每个单株重复测定3次, 取平均值。采用冷冻法观察野生型和突变体气孔特征, 以SU3500型扫描电镜观察并照相。
参照何瑞峰等[18]的方法, 于抽穗期取野生型缙恢10号和突变体ygl9倒二叶中部叶片, 以戊二醛和锇酸双重固定后, 利用不同梯度的乙醇逐级脱水, 再置换和包埋, 制超薄切片, 以醋酸双氧铀和柠檬酸铅液双重染色, H600型透射电镜观察并照相, 分析叶肉细胞和叶绿体结构。
采用BSA[19]法定位目标基因, 即从F2分离群体中选取10株正常单株和10株突变单株, 剪取等量叶片构成正常基因池和突变基因池。采用改良的CTAB法提取亲本和基因池DNA[20], 按碱煮法提取F2群体DNA[21]。参照http://www.gramene.org/microsat/公布的SSR引物, 并运用Vector NTI Advance 10自行设计InDel引物。SSR和InDel引物由上海英骏技术公司合成。PCR总体系为12.6 μ L, 含1.25 μ L 10× PCR buffer、1 μ L 50 ng μ L-1 DNA模板、0.75 μ L 25 mmol L-1 MgCl2、0.5 μ L 2.5 mmol L-1 dNTPs、8.0 μ L ddH2O、1.0 μ L 10 μ mol L-1引物、0.1 μ L 5 U μ L-1TaqDNA聚合酶。PCR程序为94℃预变性5 min; 94℃变性30 s, 55℃退火30 s, 72℃复性1 min, 35个循环; 72℃延伸10 min。PCR产物经10%非变性聚丙烯酰胺凝胶电泳, 快速银染后观察[22]。
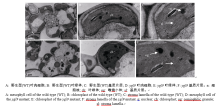
突变体ygl9叶片苗期呈现黄绿色(图1-A, B), 一直保持到拔节期(图1-C~E), 抽穗期叶色渐变为淡绿色(图1-F), 直至成熟; 野生型缙恢10号(WT)的叶片全生育期均呈绿色(图1)。与野生型相比, ygl9的株高、穗长、每穗粒数、每穗实粒数、结实率和千粒重等主要农艺性状均极显著降低, 分别降低了23.48%、11.34%、38.75%、53.95%、24.81%和30.46%, 仅有效穗数没有明显差异(表1)。
| 表1 野生型(WT)与ygl9突变体主要农艺性状分析 Table 1 Main agronomic traits of the wild type (WT) and the ygl9 mutant |
苗期与野生型相比, ygl9突变体的叶绿素a、叶绿素b、总叶绿素和类胡萝卜素含量均极显著降低, 分别只有野生型的63.28%、47.90%、60.80%和66.95% (图2-A); 分蘖期, ygl9突变体的各光合色素含量仍然极显著低于野生型, 分别下降了33.72%、55.82%、36.91%和33.63% (图2-B); 抽穗后突变体叶色由黄绿色渐变为淡绿色, 与野生型相比, ygl9突变体的光合色素含量仅表现为显著降低(图2-C), 降低幅度明显小于苗期和分蘖期。
为了探究叶色变异对突变体光合作用的影响, 在孕穗期对野生型和突变体的净光合速率、气孔导度、胞间CO2浓度、蒸腾速率4个光合特性指数进行测量。ygl9净光合速率与野生型相比并没有明显差异(图3-A), 而气孔导度比野生型提高了101.97% (图3-B), 差异达到了极显著水平。可能因为气孔导度的增加, 突变体细胞更容易摄取空气中的CO2, 蒸腾作用也更加强烈, 所以突变体的胞间CO2浓度和蒸腾速率也极显著提高, 分别比野生型增加了11.53%和73.21% (图3-C, D)。
透射电镜观察表明, 野生型和ygl9突变体叶肉细胞发育完全, 叶绿体规则地贴壁分布, 完整地被膜包裹, 大小相似, 叶绿体内部均有基粒、基质和片层结构。但野生型叶绿体基粒丰富, 基质浓厚, 基质片层排列整齐紧密(图5-A~C); 而ygl9突变体叶绿体嗜锇小体增多, 基粒模糊, 基质片层明显少于野生型且较为疏松(图5-D~F)。
以表型正常的不育系西农1A与突变体ygl9杂交, 所有F1植株均表型正常。F2群体出现明显的双亲性状分离现象, 在3156株群体中正常单株2397株、黄绿叶突变单株759株, 经卡平方测验, 符合3∶ 1分离比(χ 2=1.14 < χ 20.05=3.84), 表明ygl9黄绿叶突变性状受1对隐性基因控制。
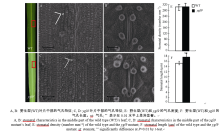 | 图4 野生型(WT)和ygl9突变体的气孔扫描电镜观察Fig. 4 Scanning electron microscopy observation of stomatal characteristics in the wild type (WT) and the ygl9 mutant |
以西农1A与ygl9杂交的F2代群体作为定位群体, 用均匀分布于水稻12条染色体上的400对SSR引物和60对InDel引物扩增亲本和基因池DNA。结果显示, 位于第3染色体短臂上的RM14281、Ind03-2、Ind03-7和Ind03-8可能与YGL9连锁。为验证连锁关系, 利用这4对引物进一步分析了210株F2突变单株, 发现RM14281、Ind03-2、Ind03-7和Ind03-8的重组子个数分别为36、3、20和40个, 其中RM14281的重组子包含Ind03-2的重组子, Ind03-8的重组子包含Ind03-7的重组子, 且Ind03-2和Ind03-7的重组子不同, 因此初步将YGL9基因定位于Ind03-2和Ind03-7之间。
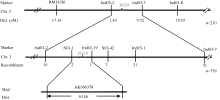
在初步定位区间内设计了12对InDel引物和44对SSR引物, 其中S03-1、S03-42、Ind03-13和Ind03-19在亲本间表现多态性, 利用多态性引物进一步分析759株F2定位群体。发现, SSR标记S03-1有2个重组子, InDel标记Ind03-19有1个重组子, 且二者的重组子不同。因此, 最终将YGL9精细定位在标记S03-1和Ind03-19之间, 遗传距离分别为0.13 cM和0.07 cM, 物理距离为63 kb (图5)。根据Gramene网站上提供的基因注释信息, 该区间内共有11个开放阅读框(open reading frame, ORF), 分别编码过氧化氢酶、泛素类蛋白、核苷酸转移酶、电子载体/蛋白质二硫键氧化还原酶、卷曲螺旋域结合蛋白124、还原转座子、叶绿体信号识别颗粒、AMP结合域蛋白、假定蛋白、棒曲霉素蛋白和HV22蛋白。
本文报道的ygl9突变体, 从苗期开始叶片便表现为黄绿色, 直到抽穗期叶片逐渐转变为淡绿色, 基因被定位于第3染色体短臂前端的63 kb区间内。这与目前已鉴定的ygl80[25](ygl1[15]等位突变体)、ygl2[16]、ygl3[26]/ygl7[27](chl1[12]等位突变体)、ygl4[28]、vyl[14]和ylc1[29]等黄绿叶突变体的定位区间均不相同, 表型也有差异。其中, 突变体ygl80、ygl3/ygl7和ygl4在整个生育期叶片均表现为黄绿色, 分别位于第5、第3和第10染色体; 定位在第9染色体的突变体vyl, 其未展开叶表现淡黄色, 展开后从叶尖向叶基部转绿; 另一个位于第9染色体上的突变基因YLC1, 其苗期便表现出黄叶性状, 但从分蘖早期开始, 只有老叶会逐渐转绿而新叶仍然保持黄叶性状。本研究的ygl9突变体与ygl2表型较为相似, 但ygl2从分蘖期到抽穗期开始转绿并几乎与野生型叶色相同, 此外, ygl2突变体被定位于第6染色体上, 与ygl9突变体定位区间不同。因此, 从叶色表型和基因定位均表明ygl9是一个新的叶色突变体。
| 表2 用于基因定位的新开发多态性标记 Table 2 Newly designed polymorphic markers used for gene mapping |
叶片中光合色素含量不仅影响植物光合作用的有序进行, 往往还决定植物叶片的颜色[30]。在苗期和分蘖期, ygl9突变体各种光合色素含量都极显著低于野生型; 而在抽穗期, ygl9突变体各种光合色素均略微升高, 仅显著低于野生型, 叶片颜色由黄绿色渐变为淡绿色。由此, 我们推测ygl9突变性状与光合色素含量降低及变化有关。细胞结构观察发现ygl9的叶绿体结构基本完整但嗜锇小体增多、基质片层减少、排列疏松, 表明突变体的叶绿体退化, 叶绿体光合膜完整性不如野生型。由于抽穗后突变体光合色素含量上升且叶绿体结构基本完整, 故该时期突变体的净光合速率略有下降但未达到显著水平。吕典华等[31]对叶色突变体ygl5和pygl1孕穗期的光合特性研究发现了类似现象: 两个突变体叶绿体内基粒数量明显少于对照, 叶绿素含量也大幅下降, 而ygl5和pygl1的光合速率都高于对照。此外, 前期营养生长阶段突变体光合色素的极显著降低及叶绿体发育的变化最终导致ygl9突变体植株矮化, 穗长、每穗粒数、每穗实粒数、结实率和千粒重等农艺性状均极显著降低。通常情况下, 随着叶片色级的降低, 叶片的气孔导度会相应减少[32]; 而ygl9的气孔长度明显增长, 气孔导度极显著增加。欧立军[33]鉴定了一个淡叶突变体, 其在强光下具有比野生型更高的气孔导度, 但其气孔大小与野生型并没有差异。因此我们猜测YGL9基因不仅在调控水稻叶色方面有重要作用, 同时还可能参与气孔的发育。
在YGL9定位区间内有11个ORF, 其中有4个ORF可能参与叶色变化。ORF1编码一个过氧化氢酶, 其功能是降解细胞内的一种活性氧H2O2, 它可作为信号分子调节叶绿体及细胞核基因的表达, 从而参与调控叶绿体发育、细胞周期、细胞程序化死亡等生理过程[34, 35]。ORF7编码叶绿体信号识别颗粒(cpSPR), cpSPR是将参与光合作用的叶绿体蛋白运输到类囊体膜的介导物质[36], 在拟南芥中已经克隆了一个编码cpSRP43的基因CHAO, 其突变体表现为黄绿叶, 各种光合色素含量明显降低[37]; 在水稻中也发现了编码cpSRP54的基因YGL138(t), 其突变体同样表现为黄绿叶[38]。ORF8编码一个AMP结合结构域蛋白, 通过影响ATP水解或与ATP互作而影响ABC转运蛋白的功能[39], 水稻SGRA编码一个ABC转运蛋白, 该基因的突变导致了叶色的变化, 突变体在6~8叶期新生白叶, 随后转绿, 呈现阶段性白化现象[40]。ORF11编码一个HVA22蛋白, 最先发现于大麦中, 是ABA应激感应蛋白[41], ABA作为类胡萝卜素的前体, 在类胡萝卜素生物合成途径中有十分重要的作用, 同时内源ABA含量降低可导致植物叶绿素含量降低, 叶片颜色介于灰绿和绿色之间[42]。因此, 我们暂将这4个基因作为候选基因, 进行下一步的分析, 当然, 其他基因是否与叶色有关, 我们也将继续关注。
ygl9突变体叶片从苗期到拔节期一直保持黄绿色, 光合色素含量极显著降低, 抽穗期叶色渐变为淡绿色, 光合色素含量显著降低。株高、穗长、每穗粒数、每穗实粒数、结实率和千粒重均极显著下降。气孔长度、气孔导度及蒸腾速率极显著增加, 净光合速率没有明显变化, 叶绿体结构基本完整但嗜锇小体增多、基粒模糊、基质片层减少且较疏松。该突变性状受一对隐性核基因控制, 定位于第3染色体短臂的SSR标记S03-1和InDel标记Ind03-19之间, 遗传距离分别为0.13 cM和0.07 cM, 物理距离为63 kb, 包含11个注释基因, 受一个功能尚未鉴定的新基因调控。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|