大麦麦芽作为啤酒酿造的主要原料之一, 其纯度决定了麦芽原料的均一性, 进而影响加工工艺和啤酒品质。为高效准确地鉴定麦芽纯度, 在啤酒企业进行麦芽原料采购和质量监测时提供参考依据。本研究分别利用EST-SSR和SNP标记定性检测了按比例预混的麦芽样品纯度, 并利用SNP标记定量检测了4份送检的麦芽盲样纯度。结果表明, EST-SSR标记能定性检测混杂度高于10%的麦芽样品, 而SNP标记能够有效鉴定混杂度低至5%的麦芽样品。SNP标记对纯度定量检测的单次抽样的测定值与真实值之间的误差在3%以内。比较发现, 本研究所用的两类分子标记均可用于麦芽样品的纯度检测, 但基于KASP技术的SNP标记可以满足麦芽纯度的快速定量检测需要。
Barley malt is one of the main raw materials used in beer production. Malt purity determines the level of its homogeneity, which affecting processing techniques and beer quality. For the efficient and accurate identification of malt purity, providing evidence for the malt raw materials procurement and quality monitoring in beer production, the purities of six proportionally premixed malt samples and another four blind samples were detected by using EST-SSR and SNP markers in this study. The results showed that the samples with mixing ratio higher than 10% were distinguished with EST-SSR markers in qualitative detection, whereas those with SNP markers were more effective even the sample impurity as low as 5%. In the quantitative detection, the standard error between the measured value and the true value was lower than 3% in one sampling. Obviously, both of the two types of molecular markers are all can be used for malt purity test, but KASP assay based-SNP detection is more suitable for rapid and quantitative malt purity test.
自2002年起, 我国啤酒产量连续12年居世界首位, 目前年产销量达到5000万吨, 占全球总量的四分之一[1]。啤酒消费量稳定增长的同时, 消费者对啤酒的风味和质量要求也进一步提高。大麦麦芽作为啤酒工业的主要原料, 其质量的优劣直接关系到啤酒的品质和口感。由于不同大麦麦芽品种之间的淀粉、蛋白质、β -葡聚糖以及酶系活性等均存在一定差异, 其酿造工艺也有一定差异, 啤酒企业对大麦麦芽纯度要求在97%以上, 因此麦芽纯度检测是啤酒加工企业尤为重视的问题[2, 3, 4]。然而, 由于我国农业生产的不确定性和进口大麦原料价格低廉等原因, 国内多数啤酒和麦芽企业没有建立自有的原料大麦供应基地, 而不同的啤酒大麦品种从种源控制、田间收获、销售收购、制麦进料, 直至麦芽出厂的产业链中, 多个环节都存在导致麦芽纯度变化的风险。
常用的品种纯度检测方法有蛋白质电泳法、同工酶电泳法和DNA指纹图谱法等。蛋白质电泳法是依据蛋白质电泳产生的特征蛋白质分子标记将不同品种区分开来, 具有重复性好, 稳定性强的优点。早在1986年, 国际种子检验协会就正式颁布了应用蛋白质酸性聚丙烯酰胺凝胶电泳(pH 3.2)鉴定小麦和大麦品种的标准程序[5], 但该方法不能有效区分亲缘关系较近的品种, 限制了它的使用范围[6]。同工酶电泳法可以实现大量样品的低成本检测, 具有稳定性好、重复率高的特点, 是目前啤酒生产企业应用最为广泛的麦芽纯度检测方法, 但可利用的同工酶数量少、多态性低, 而且酶提取要求高, 导致检测效率偏低[7]。随着DNA分子标记的开发和利用, DNA指纹图谱逐渐成为鉴定种子纯度的另一种重要方法。DNA指纹图谱法主要依靠RFLP、AFLP、RAPD和SSR等类型的DNA分子标记来构建不同品种的指纹图谱, 然后利用具有多态性的标记检测样品的纯度。谷方红等[8]从20对引物中筛选出3对多态性高的AFLP标记完成对11个啤酒大麦品种的区分。Tinker等[9]从70个RAPD标记中筛选出9个标记可区别27个大麦品系和20个DH系。黄祥斌等[10]采用PCR-RAPD技术, 鉴定了来自北美洲的5个二棱和11个六棱啤酒大麦品种。Russell等[11]利用4对SSR标记的不同组合可以区分24个大麦品种, 也可以有效区分来自相同亲本的不同基因型。
与其他类型的分子标记相比, SNP标记在植物基因组中的分布最为普遍, 不断发展的SNP检测技术为其应用提供了便利。目前, 利用SNP标记进行农作物品种鉴定的研究已有相关报道, 兰青阔等[12]应用高分辨率熔解曲线(high resolution melting, HRM)技术筛选出用于黄瓜杂交种纯度鉴定的SNP位点CLA6 (A/G), 结合焦磷酸测序技术, 建立了相应的黄瓜杂交种纯度鉴定方法。喻辉辉等[13]开发了基于水稻全基因组SNP信息的芯片RICE6K, 该芯片包含籼稻和粳稻亚种的多态性信息, 可用于水稻品种的基因型以及纯度鉴定。
DNA提取是保证研究结果真实可靠的前提, 叶片、根系等植物新鲜组织是最常用的提取材料, 而麦芽很少用于DNA提取, 并且麦芽DNA提取方法也鲜有报道, 可能是由于麦芽DNA在制麦过程的高温阶段容易被破坏, 致使完整的DNA量少且易降解。谷方红等[14]利用植物DNA提取试剂盒(DNeasy Plant Mini Kit, QIAGEN)及CTAB法, 获得了质量较好的麦芽DNA, 而采用酚-氯仿法的提取效果不理想。为确定最适合的麦芽DNA提取方法, 本研究比较了4种DNA提取方法的提取效果。
目前, 针对SNP标记的检测手段也从常规测序、CAPS (cleaved amplified polymorphic sequences)、dCAPS (derived cleaved amplified polymorphic sequences)等发展到用Taqman探针[15]、HRM [16]、AS-PCR (allele-specific PCR) [17]和KASP (Kompetitive allele-specific PCR) [18]等多种快速精准检测方法。同时由于当前基于同工酶和SSR分子标记等常规检测技术已不能满足企业开展规模化、快速高效的麦芽纯度检测的需求, 本研究提出利用最新发展的SNP标记检测技术鉴定麦芽纯度。通过比较EST-SSR和SNP标记对大麦麦芽预混样品和盲样的定性和定量纯度检测, 确定基于KASP技术的SNP标记检测方法可以实现麦芽纯度的快速定量检测, 能够满足啤酒原料质量监测的产业化需求, 为保障啤酒的商品品质提供技术支撑。
麦芽预混样品和盲样均由中国食品发酵工业研究院制备。其中, 预混样由样品A (红08-746, 国家大麦北方区试的参试品系)和样品B (甘啤6号, 国家大麦北方区试对照品种)按不同粒数比混合而成, 混杂比例为2%、5%、10%、20%、40%和50%, 依次命名为T1~T6; 4个麦芽盲样由参加区试的其他品种(系)按照质量比混合制备, 命名为C1~C4。
将上述制备的麦芽预混样和盲样分别混合均匀, 然后各取100粒混合磨粉、DNA提取, 并分别用EST-SSR标记和SNP标记进行纯度定性检测。
从混合均匀的4份麦芽盲样中2次抽样(每份样品每次48粒), 以单粒提取DNA, 在麦芽盲样定性检测筛选出多态性SNP标记的基础上, 开展纯度定量检测。
分别采用植物种子DNA快速提取法[19]、改良CTAB法[20]、植物DNA提取试剂盒(DNeasy Plant Mini Kit, QIAGEN)和新型植物基因组DNA快速提取试剂盒(北京强欣博瑞生物技术有限公司) 4种方法提取DNA, 并取1.5 μ L DNA在0.8%琼脂糖凝胶上电泳检测纯度。
1.3.1 EST-SSR引物 根据已报道的遗传图谱信息, 挑选出均匀分布于大麦7条染色体的40对大麦EST-SSR引物。引物序列从Graingenes2.0 (http:// wheat.pw.usda.gov/GG2/index.shtml)数据库获得, 引物由北京赛百盛公司合成(表1)。PCR体系为10 μ L, 含1 μ L DNA (50 ng μ L-1), 5 μ L无染料的2× pfu PCR Mix (北京强欣博瑞生物技术有限公司), 正向和反向引物各1 μ L (2 μ mol L-1)。在T100型热梯度循环仪(Bio-Rad)上进行PCR, 反应程序为94℃预变性5 min; 94℃变性30 s, 56℃退火30 s, 72℃延伸45 s, 37个循环; 72℃延伸7 min。利用QIAxcel Advanced全自动DNA/RNA分析系统(QIAGEN), 通过高分辨率毛细管电泳分离扩增产物。
1.3.2 SNP引物 根据本实验室前期研究结果(未发表), 挑选出均匀分布于7条染色体上的30个SNP标记, 并将其侧翼序列信息提交LGC公司(Laboratory of the Government Chemist, Hoddeston, UK)进行KASP引物的设计和合成(表2); PCR体系为5 μ L, 包括1.5 μ L DNA (10 ng μ L-1), KASP Mix 2.5 μ L (LGC Genomics, Hoddeston, UK), 0.07 μ L KASP Assay Mix和1 μ L ddH2O。PCR和荧光信号检测在ABI 7900荧光定量仪上完成。PCR程序为94℃热激活15 min; 94℃变性20 s, 61~55℃退火和延伸60 s, 10个循环(touch-down, 每循环降低0.6℃); 94℃变性20 s, 55℃退火和延伸60 s, 30个循环。
利用QIAxcel Screen Gel软件分析EST-SSR标记的毛细管凝胶电泳分型结果, 按照峰图及带型特点, 统计同一对SSR引物扩增条带在各个样品中的有无或片段大小差异。
利用ABI 7900荧光定量PCR仪配套分析软件的基因分型模块, 读取各样品的两种不同荧光信号值并分析基因型, 在SNPViwer2软件(LGC Genomics, Hoddeston, UK)中生成基因分型图。
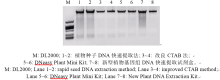
用4种方法分别提取麦芽样品B的基因组DNA, 用琼脂糖凝胶电泳检测提取纯度, 2次重复结果一致(图1)。用植物种子DNA快速提取法获得的DNA有少量杂质, 而其他3种方法均能得到质量较好的DNA。鉴于用改良CTAB法提取的DNA质量可以满足本实验需要, 且该方法成本低、批量提取所耗时间与试剂盒差别不大, 故后续实验中均采用改良CTAB法提取麦芽样品的基因组DNA。
2.2.1 EST-SSR对纯度的定性检测 高分辨率毛细管电泳结果显示, 40对EST-SSR标记中有5对(HvHVA1、HVM70、GBM1008、HVPLASC1B和HVMLOH1A)在麦芽样品A和B间的扩增产物片段具有长度多态性。利用这5对多态性标记对麦芽预混样品T1~T6进行纯度定性检测, 结果HVM70和GBM1008可有效区分混杂度在10%以上的样品, HVPLASC1B能够区分混杂度在20%以上的样品, 而HvHVA1和HVMLOH1A仅能区分混杂度在40%以上的样品(图2-a)。
2.2.2 SNP对纯度的定性检测 在所有30个SNP标记中, D01、D03、D22、D26、D27和D30呈现多态性高且稳定性好。利用这6个标记对T1~T6麦芽预混样进行检测, 对混杂比例为2%的样品进行分型时, 除标记D01可以区分样品外, 其他标记由于荧光信号值非常接近样品B而不能被确定为杂合样品, 但5个多态性标记均可以对混杂度在5%以上的麦芽样品进行有效区分。从SNP荧光信号二维图中可以看出, 样品A和样品B分别靠近两坐标轴, 即分别对应不同的SNP类型; 多数麦芽预混样落在2种荧光信号值接近的对角线附近, 说明对应2种SNP的荧光信号均被有效检出, 从而被判定为含杂样品。
| 表1 本研究所用EST-SSR标记 Table 1 EST-SSR markers used in this study |
| 表2 本研究所用SNP标记 Table 2 SNP markers used in this study |
检测结果还显示, 按照预混比例从小到大逐渐偏离样品B (图2-b)。
麦芽盲样的定性检测发现, 在30个SNP位点中13个呈杂合类型, 分别是D06、D15~D17、D20~D25和D28~D30, 其中D20和D21在样品T1~T6中呈多态性, 因此选择这2个SNP标记对4份麦芽盲样进行纯度定量检测, 结果显示各盲样均有不同程度的混杂, 且2次抽样检测结果一致(图3)。
对SNP检测为纯合的样品进行计数, 计算麦芽盲样的样品纯度(数量百分比)。与盲样制备方提供的真实值(质量百分比)比较, 结果2次抽样检测的均值与真实值的相对误差介于-0.29%和1.79%之间。C1和C4为麦芽纯度为93% (质量百分比)的同一样品, 经多次检测, 实测纯度(数量百分比)为92.71%~ 94.79% (表3), 符合工业化检测的误差要求。
 | 图2 分子标记对不同混杂比例麦芽预混样的定性检测Fig. 2 Qualitative test of premixed malt samples with different mixing ratios using molecular markers |
 | 图3 SNP标记D20和D21对麦芽盲样C1~C4的定量检测结果Fig. 3 Quantitative detection of blind malt samples C1 to C4 using SNP markers D20 and D21 |
| 表3 SNP标记D20和D21定量检测盲样的麦芽纯度 Table 3 Quantitative test of blind malt samples using SNP markers D20 and D21 (%) |
当前, 由于啤酒产业链中麦芽生产环节众多, 各种主客观因素导致的大麦麦芽纯度下降已成为影响啤酒企业生产效率和啤酒品质的亟待解决问题之一。尽管基于同工酶和SSR分子标记等技术的检测手段已经广泛应用于麦芽纯度检测, 但前人基于分子标记的麦芽纯度检测, 仅限于对发芽前的种子和培养后的叶片DNA进行检测, 并非对制备好的麦芽样品直接检测, 难以满足企业对于麦芽纯度结果的时效性需求。因此, 本研究首先比较了4种DNA提取方法在麦芽DNA提取上的适用性、DNA质量、单个样品提取的时间及费用成本, 发现4种常用方法虽均能够满足麦芽DNA提取的需要, 但综合考虑DNA质量、操作时间和实验成本等因素, 确定改良CTAB法是最适合的麦芽DNA提取方法。其次, 比较了当前常用的两种分子标记麦芽纯度检测的效果, 发现两种标记均能用于一定混杂度麦芽样品的纯度检测, 但EST-SSR检测需要电泳才能显示结果, 而SNP分型结果仅需在可检测荧光标记的酶标仪或荧光定量PCR仪上读取信号, 可以大大节省结果产出时间; 而且SNP标记在定性检测中的灵敏度和准确性更高, 大于5%的麦芽混杂均可以被定性检出。因此, 将多态性高的SNP标记结合KASP技术, 可以实现麦芽纯度快速定性和定量鉴定, 这为满足啤酒原料的源头质量监测和啤酒品质控制提供了保障。
SNP标记针对麦芽盲样定量检测结果显示, 测量值与真实值间误差小于3%, 且以质量比计算得出的麦芽样品纯度与以数量比得出的纯度测量值, 在无法确定混合前各自样本千粒重的前提下, 本身应存在着一定的差异。因此, 本研究的误差水平完全可以满足工业化检测的需求。同时, 建议在样本量允许的情况下, 可以通过增加抽样检测的次数和增加单次检测的样本量以进一步提高检测的精度。
此外, 通过逐步建立啤酒工业常用的大麦品种的SNP指纹数据库, 一方面可以利用最少的标记实现麦芽纯度检测的需要, 大大降低针对盲样开展多态性标记筛选的工作量和鉴定成本, 另一方面利用最少数量的多个SNP标记联合鉴定, 可以实现真正意义上的混杂麦芽来源品种追溯, 实现麦芽纯度的全流程控制, 为啤酒产业的良性发展提供保障。
推荐改良CTAB法作为大麦麦芽DNA提取的最适方法。EST-SSR和SNP两种分子标记对麦芽纯度的检测结果具有有效性和可靠性, 但综合考虑检测灵敏度、检测效率和实验成本等因素, SNP标记较EST-SSR标记更具优势。建立了基于SNP标记的麦芽纯度高效定性、定量检测技术体系, 为大麦麦芽生产和质量控制的全流程动态监测提供了解决方案。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|