在水稻品种Dongjin的T-DNA插入突变体库中筛选到一份黄绿叶突变体 T113, 该突变体在生长的整个时期叶片都呈现黄绿色, 且越到后期表型越明显。 T113与野生型亲本Dongjin相比, 叶片光合色素含量明显降低, 株高变矮, 结实率降低, 每穗着粒数、穗长和千粒重均明显减少, 抽穗期延迟, 且黄绿叶性状不受温度影响, 叶绿体中的类囊体排列较为疏松, 出现更多的嗜锇体, 叶绿素合成和质体发育相关基因表达量发生改变。遗传分析表明, T113的突变性状由1对隐性核基因控制。利用 T113/N22的F2群体, 将突变基因定位在第2染色体长臂Indel标记CX2和JX18之间,物理距离约为79 kb, 此区间内包含12个预测基因。
The yellow-green leaf mutant T113, which was isolated from a T-DNA mutant pool with Dongjin variety as the background material, showed a yellow-green leaf phenotype in whole developing stage. Compared with wild type, the contents of chlorophyll and carotenoid decreased, the yellow-green leaf became more and more obvious along with developing in T113. At maturity, the number of productive panicles per plant, panicle length, seed setting rate, 1000-grain weight and plant height reduced. The date of heading of T113 also delayed. The phenotype of mutant was not affected by temperature. Ultrastructural analysis showed that the chloroplast of mutant was brighter than that of wild type, the mutant developed loosed thylakoid lamellar structures. The expression of genes associated with chlorophyll biosynthetic and chloroplast development of T113changed a lot. Genetic analysis showed that the yellow-green leaf trait of the T113 mutant was controlled by one pair of recessive nuclear genes. Genetic mapping of the mutant gene was conducted using a F2 mapping population of T113/N22. Finally, the mutant gene was mapped between Indel markers CX2 and JX18 on the long arm of chromosome 2 with physical distance of 79 kb, in which 12 predicted genes had been annotated.
叶色变异发生频率高, 且容易鉴定, 在20世纪初就有关于叶色突变体的报道[1], 但由于多数叶色突变同时伴随产量的降低, 甚至植株的死亡, 叶色突变体在过去常被认为是无意义的突变, 其价值没有得到重视。随着功能基因组学的发展, 众多的研究结果都显示叶色突变体是研究植物光形态建成、光合作用、激素生理和核-质基因信号传导途径等光合系统功能、结构及其调控机制的理想材料。目前已在多种植物中均发现了叶色突变体[2, 3, 4, 5]。
导致叶色变异的原因众多, 主要由叶绿素合成及叶绿体分化和发育受阻引起。叶绿素的生物合成从谷氨酰-tRNA开始, 先是一步步转变为原卟啉IX, 接下来一部分原卟啉IX螯合镁离子, 通过一系列的反应, 最终形成叶绿素a和叶绿素b, 高等植物参与叶绿素合成的所有酶都已鉴定[6], 目前已报道的水稻中编码这些酶的基因有Chl1、Chl9[7]、OsCHLH[8]、OsDVR[9]、OsPORA/FGL、OsPORB[10]、YGL[11]、OsCAO1和OsCAO2[12]。另一部分原卟啉IX螯合铁离子生成亚铁血红素和光敏色素, 已有研究证明血红素可反馈调节叶绿素的合成[13]。叶绿体的分化和发育受到核基因组、叶绿体基因组、光照及器官特异性的共同调节[14, 15]。例如参与光系统I和光系统II构成的PsaA、PsaB、PsbA[16, 17], 参与光合作用的RbcL[18], 参与叶绿体基因表达的RpoA (RNA聚合酶α 亚基)及在类囊体膜上参与电子传递及光呼吸的NDH2和NDH4[19]。叶色突变主要由核基因突变引起, 水稻的叶色突变一般由单隐性核基因控制, 较少由显性基因或细胞质基因控制。
目前已分离和鉴定众多水稻叶色突变体, 其中已克隆的基因达50多个。但人们对水稻叶色突变的分子机制仍没有十分完整的认识, 需要对更多水稻叶色突变体进行研究。本研究中对T113黄绿叶突变体进行了表型和生理等方面的研究, 并利用T113/N22的F2分离群体, 将黄绿叶突变基因精细定位到第2染色体长臂下端Indel标记CX2和JX18之间79 kb的区间, 区间内包含12个基因。
水稻黄绿叶突变体T113筛选自粳稻品种Dongjin的T-DNA插入突变体库。从苗期开始观察叶色表型, 成熟期时调查突变体T113和野生型亲本Dongjin的株高、剑叶长、剑叶宽、分蘖数、穗长、穗粒数、结实率、千粒重等主要农艺性状, 每个样本3次重复, 调查样本数为20株。
将野生型和突变体T113种植于大田。在幼苗长至二叶一心期和抽穗时分别取相同部位的叶片, 每个材料重复3次, 每次重复从相同处理的5个植株称取0.2~0.5 g, 剪成2~3 mm碎片, 在避光条件下用5 mL的95%酒精处理48 h, 之后吸取200 mL上清液, 用酶标仪在黑暗条件下测量上清液在665 nm、649 nm和470 nm 3个波长下溶液光密度。参照Lichtenthaler的方法[20], 测定和计算叶片单位鲜重或单位面积的叶绿素a (Chl a)、叶绿素b (Chl b)和类胡萝卜素的含量。Chl a浓度Ca= 12.21 A665-2.81 A645; Chl b浓度Cb= 20.13 A645-5.03 A665; 类胡萝卜素浓度Cx = (1000D470-2.05Ca-114Cb)/245。
将野生型和突变体T113分别培育在20℃、23℃、26℃和30℃的光照恒温培养箱中培养相同的时间, 观察叶色表型并测定相同部位叶片的光合色素含量, 测定方法同1.2。
将二叶一心时期的野生型和T113突变体相同部位叶片材料固定于3%戊二醛和0.1 mol L-1磷酸缓冲液(pH 7.2)中4 h; 冲洗后再固定于1%锇酸(pH 7.2)中4 h, 用30%、50%、70%、80%、90%、100%系列乙醇脱水后用环氧树脂SPURR包埋、聚合。随后用LKB-V型切片机将样品切成约l μ m厚的切片, 经醋酸铀染色, 最后在日立JEM-1230透射电子显微镜下观察、照相。
使用天根生化科技(北京)有限公司植物总RNA提取纯化试剂盒, 提取生长至二叶一心期时的野生型和突变体相同部位叶片组织总RNA, 提取步骤见其说明书。使用TaKaRa生物公司的反转录试剂盒(PrimeScript 1st Strand cDNA Synthesis Kit)对总RNA进行反转。内参基因为ACTIN基因, 在Applied Biosystems公司7500 Realtime PCR仪上扩增, 按2-∆ ∆ CT方法分析基因的相对表达量[21], 每个样本3次重复。
将T113与叶色正常的野生型亲本Dongjin及籼稻品种N22杂交, 在海南陵水及江苏南京分别播种F1群体及F2群体。将T113与N22的F2群体作为定位群体, 选取叶色变异的F2单株作为极端个体, 分单株取叶片提取DNA。参照McCouch等方法提取水稻叶片总DNA [22]。
参照http://www.gramene.org/microsat的SSR引物标记, 并运用Primer5.0软件设计Indel标记。SSR和Indel引物由南京金斯瑞生物科技有限公司和上海英俊生物技术有限公司合成。利用引物对供试亲本进行多态性检测, 然后利用多态性引物对T113/ N22 F2极端个体进行连锁分析。按照Shi等[23]的PCR扩增体系, 用8%聚丙烯酰胺凝胶电泳检测PCR产物, 230 V电压下电泳1~2 h, 电泳后银染显色, 最后在灯箱上进行DNA条带观察记录, 与隐性亲本一致的带型记为1, 与显性亲本带型一致的记为2, 同时具有两亲本带型的记为3。最后计算遗传距离, 构建分子标记连锁图谱。
利用水稻基因组注释数据库http://rice.plantbiology.msu.edu/和http://rapdb.dna.affrc.go.jp/及水稻表达数据库http://ricexpro.dna.affrc.go.jp/, 根据T113突变体的突变性状、分子标记间物理距离, 在突变基因精细定位的染色体区段上遴选T113突变体的候选基因。
水稻黄绿叶突变体T113在人工光照培养箱及大田自然环境下均表现出黄化表型, 且大田条件下黄化表型更为明显。突变体在整个生育期均表现黄化, 非常稳定(图1)。与野生型相比, T113株高显著变矮, 仅为野生型的69%、抽穗期延迟一周左右、穗子变小、结实率和千粒重减少。其他性状如剑叶长、剑叶宽、分蘖数, 也存在不同程度的差异(表1)。因此, 突变体T113不仅叶色受到影响, 植株的生长也受到了影响。
 | 图1 野生型Dongjin (WT)和突变体T113在苗期和抽穗时期的表型Fig. 1 Phenotype of the wild type Dongjin (WT) and mutant T113 in seedling and heading stages |
| 表1 野生型Dongjin和突变体T113的农艺性状比较 Table 1 Agronomic traits comparison between Dongjin (WT) and mutant T113 |
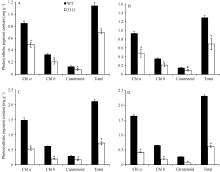
对比野生型和突变体在幼苗期和成熟期时的光合色素含量(图2), 发现T113比野生型显著降低, 幼苗期T113的叶绿素a、叶绿素b、类胡萝卜素含量分别为野生型的26.76%、33.34%和48.97%; 成熟期以后略有恢复, 可能是由于部分叶绿素降解导致, 但仍只有对照的68.43%、71.35%和77.93%, 表明T113的突变性状主要是由光合色素含量下降引起的。T113幼苗期及成熟期的叶绿素a/b分别为野生型相应时期的80.33%和95.86%, 说明突变体叶绿素a的减少比叶绿素b更为显著。
为了解T113叶色变异是否受温度影响, 我们在光照培养箱中进行了突变体的温敏性实验。结果显示, 在不同温度条件下, T113突变体均表现出黄绿叶表型(图3)。且光合色素含量均较野生型显著降低(图4)。说明T113突变体是一个不受温度影响的温钝型叶色突变体。
 | 图3 不同温度下野生型(WT)和突变体T113(MT)的表型对比Fig. 3 Phenotype of mutant (MT) T113 and wild type (WT) under different temperatures |
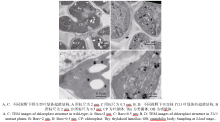
透射电镜观察显示, T113黄化叶片叶绿体中的类囊体相对于野生型排列较为疏松, 且多出了很多嗜锇体, 叶绿体比野生型整体颜色暗(图5)。
将T113分别与野生亲本Dongjin和籼稻品种N22杂交, F1植株均表现为正常绿叶, F2代群体中正常绿苗与黄绿苗分离明显, 分离比例经卡方(χ 2)测验符合3∶ 1 (表2), 表明T113的突变性状由1对隐性核基因控制。
| 表2 突变体T113与正常绿色品种杂交F2的叶色分离 Table 2 Segregation of leaf color in F2 population from the crosses betweenT113mutant and green leaf varieties |
进一步分析了叶绿素合成和叶绿体发育相关基因表达情况。荧光定量PCR的结果显示, T113突变体被检测的与叶绿素合成相关的基因表达量明显降低(图6), 而叶绿体发育相关的基因中, 参与叶绿体光合系统的PsaA、PsaB、PsbA、RbcL表达量显著增加, 但NDH2和NDH4及参与叶绿体基因表达的RpoA表达量则明显降低(图7)。表明T113突变体中叶绿体的发育和叶绿素的合成均受到了大的影响, 这个结果与其叶绿体的透射电镜结果一致。
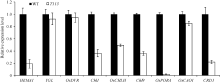 | 图6 野生型和突变体中叶绿素生物合成相关基因的荧光定量PCR分析Fig. 6 Quantitative RT-PCR analysis of genes associated with chlorophyll biosynthetic in wild type and T113 mutant |
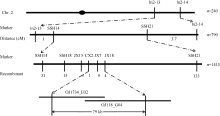
利用分布在水稻12条染色体上的160个多态性较好SSR及Indel标记, 对突变体T113和籼稻品种N22杂交组合的F2的10个极端隐性单株进行连锁分析, 将突变基因初步定位在第2染色体长臂Indel2-13和Indel2-14这2个标记之间(图8)。在初定位基础上, 根据发表在NCBI (http://www.ncbi.nlm. nih.gov/)和Gramene (http://www.gramene.org/Oryza_ sativa)上籼稻9311和粳稻日本晴的第2染色体的序列, 在Indel2-13和Indel2-14之间开发了一系列Indel标记, 用于进一步精细定位。最终利用1410个隐性单株将该基因定位在新开发的Indel标记CX2和JX18之间, 物理距离为79 kb。在该区间内有12个预测基因, 具体功能见表3。
| 表3 水稻第2染色体定位区间内基因及其预测功能 Table 3 Gene names and their putative functions in the target interval of chromosome 2 in rice |
高等植物产生叶色突变的机制十分复杂, 受遗传、环境及其互作的共同影响。例如温度对酶的活性产生影响, 因此有的叶色突变体在不同温度下突变表型不同[24]。叶绿素生物合成和降解途径的突变导致叶绿素含量降低或无法正常降解, 在光合色素总量中各色素的比例失调。水稻OsCHLH、Chl1和Chl9分别编码镁离子螯合酶的H、D和I亚基, chl1和chl9的突变体为黄绿叶, OsCHLH的突变体为白化表型, 是水稻中首个克隆的叶绿素合成的关键酶[7, 8]; 血红素合成途径的突变造成亚铁离子与卟啉分子中心内的四吡咯环上的氮原子无法正常配位结合, 导致四吡咯代谢紊乱, 叶绿素合成与降解的动态失衡。番茄aurea突变体即是一个编码血红素氧化酶的基因发生突变, 导致细胞内大量积累血红素, 四吡咯代谢紊乱, 影响叶绿素的合成[13]; OsTATC编码一个影响叶绿体分化的转运酶蛋白TATC, 其突变体叶片为苍白色, 叶绿素随着植株的生长逐渐丧失, 最终死亡[25]; 叶绿体的形成和代谢依靠细胞核-质基因组间的相互合作, 血红素被证实是核质信号转导途径中的信号分子[26]; 植物缺少某种必需元素会表现出缺素症, 病症多为缺绿和坏死斑点。YS1(yellow stripe 1-like gene)类蛋白能跨膜转运Fe-麦根酸复合物, OsYSL16蛋白通过维管束对脱氧麦根酸Fe3+的运输和分配, 维持植物体内Fe的稳态平衡[27, 28]。本研究中T113突变体在水稻的整个生长周期都表现出黄绿叶表型, 其光合色素含量比野生型明显降低, 且不受温度影响, 叶绿体类囊体排列较野生型更为疏松, 出现更多嗜锇体, 叶绿素合成和叶绿体发育相关基因表达量发生改变, 说明T113控制叶绿体发育的相关基因可能发生变异, 导致其无法形成正常的叶绿体。
| 表4 T113图位克隆的引物序列 Table 4 Primers used in map-based cloning of T113 |
目前在水稻第2染色体已经报道了一些与叶色有关的基因, 如Cga1[29]、OsGluRS[30]、Zebra524[31]、Abc1[32]等。本研究利用T113/N22的F2作为定位群体, 将突变基因定位在第2染色体长臂Indel标记CX2和JX18之间, 2个标记之间的物理距离约为79 kb, 此区间包含12个预测基因, T113突变体有可能是由一个新的叶色基因突变引起的。在该定位区间内, Os02g0744900含有FAD结构域, 编码香叶基香叶基还原酶, 在植物中, 香叶基香叶基焦磷酸是类胡萝卜素、赤霉素、维生素E和叶绿素的合成前体[33]。胡萝卜素与叶黄素组成类胡萝卜素, 在光合作用中有吸收和传递光能以及保护叶绿素的功能; Os02g0745000编码一个ATP酶, ATP酶催化ATP水解为ADP和磷酸根离子, 释放能量, 光合作用及叶绿素合成等过程都需要消耗ATP。ATP水解还与一些离子转运过程相关。例如, 钠钾ATP酶水解能够使钠离子运出细胞, 同时钾离子被运入[34]。而草莓白化植株的矿质元素含量测定发现, 白化植株具有较高的K+含量[35]; Os02g0745400编码糖基转移酶。植物内源激素不仅是植物生长发育及信号应答的重要参与者, 同时也是影响叶色的因素之一。在小麦花药和子房培养时添加吲哚乙酸和BA, 能够降低白化植株的出现率[36]。糖基转移酶能调节植物激素的含量。在赤豆(Vignaangularis)中克隆出一个能够特异催化反式脱落酸的糖基化的糖基转移酶[37]。Os02g0745700编码SUR2类似鞘脂类羟化酶, SUR2催化二氢鞘氨醇转化为植物鞘氨醇[38]。鞘氨醇属于鞘脂类, 为细胞膜组成成分之一。但哪一个基因是T113突变体的候选基因, 仍需进一步研究, 并探究最终候选基因的功能, 将该叶色基因应用到育种、光合系统功能、结构及其调控机制等更广泛的研究领域中。
黄绿叶突变体T113叶片在水稻生长的整个生育期均表现黄绿色。与野生型相比, T113光合色素含量降低, 且不受温度影响, 抽穗期推迟一周。株高降低, 仅为野生型的69%, 穗长变短, 每穗着粒数、结实率及千粒重均显著减少, 其他性状如剑叶长、剑叶宽、分蘖数也存在不同程度的差异。T113黄化叶片中的叶绿体类囊体排列较为疏松, 且出现更多嗜锇体, 其叶绿素合成和质体发育相关基因表达量发生改变。T113的突变性状由1对隐性核基因控制。利用T113/N22的F2作为定位群体, 将突变基因定位在第2染色体长臂Indel标记CX2和JX18之间79 kb的物理距离内, 此区间内包含12个预测基因。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|