第一作者联系方式: E-mail:kongshan08@163.com, Tel: 13720351933
以远杂9102为母本, 徐州68-4为父本杂交衍生的F5和F6共188个家系, 构建了一张包含365个标记, 总长度713.07 cM, 标记间平均距离1.96 cM的栽培种花生遗传图谱。图谱包含22个连锁群, 各连锁群平均长度12.37~ 81.39 cM, 连锁群上标记数量3~46个。结合2013和2014年采集的荚果表型数据, 采用WinQTLcart 2.5软件的复合区间作图法(composite interval mapping, CIM)进行QTL定位和效应估计。2个环境下共检测到41个QTL, 其中与荚果长、宽、厚和百果重相关的QTL分别为13、7、13和8个, 表型变异解释率为3.14%~18.27%。有6个QTL在2种环境下被重复检测到, 其中百果重相关的2个( qHPWLG13.1、 qHPWLG14.1), 分布在LG13和LG14连锁群, 遗传贡献率为6.95%~14.60%; 与荚果长相关的3个( qLPLG2.2、 qLPLG13.1、 qLPLG14.1), 分布在LG2、LG13和LG14连锁群, 遗传贡献率为3.14%~18.27%; 与荚果厚相关的1个( qTPLG3.4), 分布在LG3连锁群, 遗传贡献率为8.24%~9.24%。本研究涉及性状存在9个QTL热点区, 每个热点区涉及2~3个性状, 表型贡献率为3.57%~18.27%。
One hundred and eighty-eight recombinant inbred lines (RIL), derived from a cross between two Spanish type peanut cultivars (Yuanza 9102 × Xuzhou 68-4), were used as mapping population. Finally, a genetic linkage map consisting of 443 SSR loci in 22 linkage groups and covering 713.07 cM with an average distance of 1.96 cM was constructed. The length of linkage group was from 12.37 cM to 81.39 cM and the number of markers was 3-46. QTL mapping of the traits related to pod was conducted by using CIM model in WinQTLcart 2.5. A total of 41 QTLs were detected in the two environments, including thirteen for pod length, seven for pod width, thirteen for pod thickness and right for hundred pod weight, every single QTL explained 3.14%-18.27% of the phenotypic variation. A total of six QTLs were detected in both environments, including three for pod length with explained phenotypic variance of 3.14%-18.27% on the linkage group 2, linkage group 13 and linkage group 14. One for pod thickness with explained phenotypic variance of 8.24%-9.24% on the linkage group 3, and two for hundred pod weight with explained phenotypic variance of 6.95%-14.60% on the linkage group 13 and linkage group 14. The result showed that there were nine hotsports for QTL research, and each of them was associated with 2-3 traits, explaining 3.57%-18.27% of the phenotypic variation.
花生(Arachis hypogaea L.)是我国重要的油料作物和经济作物, 为居民提供重要的优质植物油脂和植物蛋白质。在我国油料作物中, 花生种植面积仅次于油菜和大豆, 但总产和单产一直位居全国油料作物第一位[1]。由于栽培种花生是异源多倍体, 其基因组较大, 遗传多样性不丰富[2], 有关花生遗传图谱构建、基因定位和重要性状QTL研究分析相对落后。随着分子标记技术的开发和所用材料的改变, 近年来在花生重要产量性状的QTL定位方面已取得一定的进展。Shirasawa等[3]通过2个F2群体的研究, 定位了一些与单株饱果重、荚果长、荚果厚、荚果宽、果嘴性状、粒重和单株粒数相关的QTL, 遗传贡献率为6.8%~28.2%。Selvaraj等[4]利用Tamrun OL01× BSS56构建的重组自交系群体, 结合分离群体分组分析方法和SSR标记, 研究了栽培种花生荚果和种仁相关性状的QTL, 认为存在主效QTL [5]。张新友等[6]和刘华等[7]通过对重组自交系群体的分析, 用2个不同的软件WinQTLCart 2.5和QTLNetwork 2.0对2个环境中的花生产量性状进行QTL检测, 发现2个软件在2个环境下均能检测到与产量性状相关的QTL, 但WinQTLCart 2.5检测到的QTL比QTLNetwork 2.0检测到的QTL多。从上面的分析可以看出, 与花生产量相关性状的QTL的研究报道较少, 而且用不同的材料可能会检测到不同的与花生产量性状相关的QTL。荚果大小是花生产量构成的重要成分。亲本品种徐州68-4 [8]是一个优良的中大果花生品种, 在我国育成品种中, 很多都含有徐州68-4亲缘; 远杂9102 [9]属珍珠豆型新品种, 经栽培种与野生种有性杂交得到, 性状优良。本研究鉴定与荚果大小相关的QTL, 将为花生高产育种中优良亲本的选择和分子标记辅助育种提供基本信息和材料基础。
以远杂9102为母本, 徐州68-4为父本杂交构建的重组自交系群体F5和F6共188个家系, 远杂9102属于珍珠豆型中小果品种, 徐州68-4属普通型中大果品种。
2013年在武汉中国农业科学院油料作物研究所试验农场种植F5代材料及其亲本, 每份材料种植1行, 行长2.50 m, 行距0.33 m, 单粒播种, 出苗后确保每行12株, 株距0.20 m, 2次重复, 常规田间管理。收获时去掉每行的边株, 将其他10株晒干, 随机选取10个成熟饱满的荚果紧密排成直线, 中间不留空隙, 测量荚果长、荚果宽和荚果厚。重复3次, 计算平均值, 并换算成单个荚果的长度、宽度和厚度。2014年在相同试验农场种植F6材料, 2次重复, 种植方法、田间管理、收获考种同2013年。
取亲本和RIL群体代表性株系的幼嫩叶片, 采用改良CTAB法提取基因组DNA。采用国内外文献已发表的[10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27]和本实验室开发的基因组SSR引物和EST-SSR引物。PCR体系为10 μ L, 含10 mmol L-1 Tris-HCl (pH 8.3)、50 mmol L-1 KCl、300~400 μ mol L-1 dNTPs、10~40 pmol L-1引物对、10~20 ng模板DNA。反应程序为Touchdown, PCR产物经6%变性聚丙烯酰胺凝胶电泳检测, 硝酸银染色, 显影, 扫描保存。
SSR数据按母本带型记为“ a” , 父本带型记为“ b” , 杂合带型记为“ h” 统计。对于显性标记, 若母本等位基因为显性, 则父本带型记为“ d” , 杂种带型记为“ b” ; 若父本等位基因为显性, 则母本带型记为“ a” , 杂种带型记为“ c” 。无论在显性标记还是共显性标记中, 带型模糊不清或数据缺失均用“ -” 表示。
首先剔除缺失比例超过10%的标记。对剩余标记数据进行χ 2检验, 确定是否符合孟德尔分离比(显性标记为17:15, 共显性标记为15:2:15), 统计发生偏分离的标记。采用JoinMap 3.0 [28]软件进行连锁分析, 设置LOD≥ 2, 步长为0.5, 在2.0~20.0的LOD值范围内将所有标记分组, 并利用Kosambi函数将重组率转换为图谱距离(cM), 构建连锁图谱。采用WinQTLcart 2.5软件[29]的复合区间作图法(composite interval mapping, CIM) [30]进行QTL定位和效应估计。QTL的命名以q加性状的英文名称首字母加所在连锁群命名, 若同一连锁群上出现2个或以上QTL时, 则QTL后面加“ .” 和数字区分。如qLPLG6.1表示位于第6连锁群上的一个控制荚果长的QTL。
亲本品种徐州68-4和远杂9102在荚果长、百果重等4个性状上均表现出明显的差异(表1)。徐州68-4 4个性状的表型值均大于远杂9102。RIL群体4个性状的表型值变异范围大, 在2个环境中最大值都超过了高值亲本, 最小值低于低值亲本, 表明这4个性状同时具有正向和负向超亲优势, 且呈连续分布, 其峰度和偏度绝对值都小于0.5, 基本符合正态曲线(图1), 表明花生荚果大小相关的性状为多基因控制的数量性状, 适合进行QTL的定位分析。
| 表1 亲本及RIL群体表型数据统计分析 Table 1 Statistics analysis of phenotypic data for two parents and its RIL population |
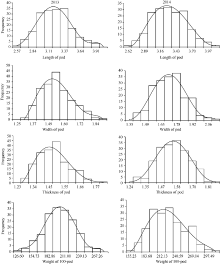 | 图1 2013和2014两个环境下荚果长、宽、厚和百果重表型数据频次分布图Fig. 1 Frequency distributions of length of pod, width of pod, thickness of pod, and weight of 100-pod in 2013 and 2014 |
花生的荚果长、荚果宽、荚果厚和百果重4个性状在2个环境中均两两呈极显著正相关(表2), 表明这4个与荚果大小相关的性状之间是互相影响的, 而且荚果长、宽、厚都对提高花生果重具有贡献。
| 表2 花生荚果性状的相关分析 Table 2 Correlation coefficients among the pod traits in peanut |
通过亲本徐州68-4和远杂9102对3765对SSR引物(1452对引物为本实验室自主开发)进行多态性筛选, 得到560对多态性较好、条带清晰的SSR引物进行RIL群体基因型鉴定。在RIL群体中能重复检测到清晰条带的引物438对(204对为本实验室自主开发的), 其中434对引物检测到1个位点, 1对引物检测到3个位点, 3对引物检测到2个位点, 438对引物共产生443个位点, 多态性标记中共显性标记430个, 显性标记13个。经卡平方(χ 2)测验(P=0.01), 在443个标记中, 偏离孟德尔分离规律的标记有250个, 占56.43%。这250个偏分离标记有220个偏向父本徐州68-4的基因型, 30个偏向母本远杂9102的基因型。
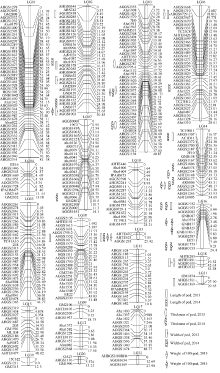
利用JoinMap 3.0软件对上述多态性分子标记进行遗传连锁分析, 在2.0~20.0的LOD值范围内, 构建遗传图谱, 得到包含22个连锁群、365个标记位点的遗传图谱(图2)。连锁群总长度为713.07 cM, 标记间平均距离1.96 cM, 连锁群长度在12.37~ 81.39 cM范围内, 标记个数介于3~46之间(表3)。长度最大的是LG1连锁群(81.39 cM), 同时也是标记最多的连锁群(46个), 标记密度最大的LG7连锁群, 其标记间平均距离只有0.56 cM。
| 表3 标记在连锁群上的分布 Table 3 Distribution of markers in the linkage groups |
在上述遗传连锁图的基础上, 结合2013年和2014年的荚果表型数据, 采用Windows QTL Cartgrapher 2.5软件进行QTL定位分析, 采用复合区间作图法(composite interval mapping, CIM), LOD值选为2.5。分析结果表明, 2013年和2014年2种环境下共检测到41个QTL, 分布在10个连锁群上(表4), 其中2013年检测到17个QTL, 2014年检测到24个QTL (表4)。
| 表4 复合区间作图方法检测到的 QTL及同一性状重复性QTL分析结果 Table 4 QTLs associated with peanut pod traits by CIM method and repeated QTL analysis with the same trait |
在2013年检测到的17个QTL中, 7个与荚果长相关, 贡献率为3.57%~9.83%, LOD值为3.27~6.91, 其中6个QTL的加性效应为正值, 1个QTL的加性效应为负值; 2个与荚果宽相关, 贡献率为4.56%~5.26%, 其中1个QTL的加性效应为正值, 另1个QTL的加性效应为负值; 5个与荚果厚相关, 贡献率为4.70%~8.92%, 其中4个QTL的加性效应为负值, 1个QTL的加性效应为正值; 3个与百果重相关, 贡献率为8.44%~14.60%, LOD值为5.86~6.52, 其中2个QTL的加性效应为正值, 1个QTL的加性效应为负值。在2014年检测到的24个QTL中, 6个与荚果长相关, 贡献率为3.23%~18.27%, LOD值为3.40~15.01, 其中5个QTL的加性效应为正值, 1个QTL的加性效应为负值; 5个与荚果宽相关, 贡献率为5.32%~8.84%, 其中3个QTL的加性效应为正值, 2个QTL的加性效应为负值; 8个与荚果厚相关, 贡献率为3.90%~9.24%, 其中6个QTL的加性效应为负值, 2个QTL的加性效应为正值; 5个与百果重相关, 贡献率为4.74%~12.36%, LOD值为3.64~7.92, 其中3个QTL的加性效应为正值, 2个QTL的加性效应为负值。
同一连锁群上控制不同性状的QTL存在置信区间重叠现象, 本研究共检测到9个区域与2个或3个性状相关(图1和表5), 区间内不同性状QTL具有相同的效应方向。如连锁群LG6上的区间AHGS1710~ AHGS0729和连锁群LG18上的区间AHTE0381~ AGGS0100内均存在与荚果宽和荚果厚相关的QTL。位于LG6连锁群的qWPLG6.1、qTPLG6.1加性效应值分别为0.027、0.024; 位于LG18连锁群的qWPLG18.1、qTPLG18.1加性效应值分别为-0.026、-0.025。连锁群LG2上的区间AGGS1925~AGGS1003内存在与荚果宽和百果重相关的2个QTL, 连锁群LG3上的区间AGGS1442~AHGS2191内存在与荚果厚和百果重相关的2个QTL, 连锁群LG13上的区间AHTE523~AGGS1233内存在与荚果长和百果重相关的2个QTL, 连锁群LG14上的区间ARS806~ AHGS2159内存在与荚果长、荚果宽和百果重相关的3个QTL, 连锁群LG16上的区间AGGS1932~ AGGS1953内存在与荚果长、荚果宽和荚果厚相关的3个QTL, 区间Ahz0574~GNB1040内存在与荚果厚和百果重相关的2个QTL, 区间AGGS1773~ AHGS0153内存在与荚果宽、荚果厚和百果重相关的3个QTL, 这些QTL的表型贡献率在3.57%~ 18.27%范围内。
| 表5 不同性状间重复性QTL分析 Table 5 Repeated QTL analysis between different traits |
本研究选用的2个亲本材料均是育种中的优良品种, 其结实性能和对环境的适应性较好, 在群体构建过程中, 每一个F2发育来的家系都完整保留, 没有丢失。该群体的双亲在荚果性状方面的差异显著, 徐州68-4的4个与荚果大小相关性状的表型值均大于远杂9102的对应值。通过表型性状的统计分析, 本研究所用的RIL群体在4个与荚果大小相关性状方面的变异符合正态分布, 其峰度和偏度绝对值都小于0.5 (图1)。而且, RIL群体在4个相关性状方面的差异均较大, 最大值和最小值均超过了双亲间的差异, 这些结果为遗传图谱构建和QTL分析奠定了基础。
近年来, 随着更多的SSR标记被开发出来, 以SSR标记为主的花生遗传连锁图谱构建工作取得了一定的进展。目前花生遗传连锁图的构建工作主要围绕栽培种花生展开, 所用作图群体多为F2或RIL群体, 单个遗传连锁图上的标记数量一般都在200个左右, 利用单个遗传群体构建的包含300个以上SSR标记的遗传连锁图还较少。Qin等[31]构建了一张包含324个SSR标记、总长度1352.1 cM的栽培种花生遗传图谱; 洪彦彬等[2]利用RIL群体为材料构建了一张包含108个SSR标记位点的遗传连锁图, 该图谱总长568 cM, 标记间平均距离为6.45 cM; Wang等[32]构建了一张包含318个SSR标记位点的栽培种花生遗传连锁图, 总长1674.4 cM, 标记间平均距离5.3 cM。将本研究构建的遗传连锁图与Wang (2012)构建的连锁图比较发现, 虽然本研究构建的连锁图覆盖的总图距没有Wang (2012)构建的连锁图长, 但本研究构建的连锁图的标记密度高, 而且有很多标记(204个)是本实验室新开发的, 2个连锁图上的共同标记只有23个。因此, 本研究构建的连锁图为栽培种花生高密度遗传图谱的整合奠定了基础。Shirasawa等[33]利用15个遗传连锁图整合了1张涉及20个连锁群、3692个标记的遗传连锁图, 这是目前花生中标记密度最高的遗传图谱。比较发现, 本研究构建的22个连锁群中的14个连锁连锁群能与Shirasawa (2013)整合图谱的11个连锁群对应(表3), 其中存在2个或3个连锁群对应Shirasawa (2013)整合图谱的某一个连锁群的现象(图3)。本研究有些标记在Shirasawa (2013)整合的连锁图中不存在, 可以作为新标记和今后图谱整合利用(包括217个新标记)。本研究构建的图谱覆盖率较低(长度仅713.07 cM), 可能是由于本研究所用的SSR标记集中分布在基因组的某些区域。从本研究构建的连锁群与Shirasawa (2013)整合的连锁群的比较中可以看出, 本研究所用的SSR标记在基因组的分布不均匀。下一步的研究中, 要加强Shirasawa (2013)整合的连锁群中标记的利用, 同时开发更多的SSR标记。
一般认为贡献率在10%以上的QTL为主效QTL; LOD值越大, QTL的准确率越高。本研究在2个环境下共检测到41个QTL, 贡献率在10%以上的QTL有5个, LOD值在5.0以上的QTL有18个。在2个环境下重复检测到的QTL有6个, 包括荚果长相关的QTL 3个, 其中qLPLG2.2的贡献率为7.85%~ 15.51%, LOD值6.60~16.15; qLPLG13.1贡献率为9.83%~18.27%, LOD值6.91~15.02; qLPLG14.1贡献率为3.14%~6.04%, LOD值3.40~4.81; 与荚果厚相关的QTL 1个, qTPLG3.4, 贡献率为8.24%~9.24%, LOD值4.32~6.48; 与百果重相关的QTL 2个, qWHPLG13.1贡献率为12.36%~14.60%, LOD值6.52~7.92; qWHPLG14.1贡献率为6.95%~9.45%, LOD值5.22~6.32。可见, 与荚果长相关的qLPLG13.3和与百果重相关的qWHPLG13.1为准确率较高的主效QTL。通过与Shirasawa (2012)以及Selvaraj (2009)等获得的荚果长、荚果厚、荚果宽和粒重相关的QTL的比较发现, 本研究获得的QTL与他们的不同, 是新的。
本研究发现9个区间与2个或3个性状相关, 这些位于同一区间内的QTL有可能是一个多效性QTL, 即一个QTL同时影响着多个性状的表型变异。LG13上的区间AHTE523~AGGS1233内存在与荚果长和百果重相关的QTL, 且荚果长和百果重相关的QTL在两个环境中可重复被检测到, QTL的表型贡献率为9.83%~18.27%, 是主效QTL; LG13上的区间ARS806~AHGS2159内存在与荚果长、荚果宽和百果重相关的QTL, 其中百果重的QTL在两个环境中都可被检测到, 单个QTL可解释的遗传变异在4.72%~9.45%范围内。这些与荚果大小相关的不同性状(荚果长、荚果宽、百果重)的QTL位于同一区间, 也表明了这些QTL的稳定性。因此, 这是2个有价值的QTL, 可以进行进一步加密的研究。
以往关于花生产量与主要农艺性状间的关联分析表明, 百果重是影响花生产量的主要因素之一, 是花生高产育种和品种推广中首先考虑的性状[34, 35, 36, 37]。本研究表明, 与荚果大小相关的4个性状两两极显著正相关。由此可以推测, 对这4个性状中任何1个性状的选择, 就可以预测另3个性状的变异。如选择荚果较长或较宽的材料, 可以预测该材料的百果重会比较大。因此该研究结果对于花生的田间选育和品种审查具有一定的指导意义。
构建了一张包含365个标记, 总长度713.07 cM, 标记间平均距离1.96 cM的栽培种花生遗传图谱。两年共检测到41个QTL。其中与荚果长、宽、厚和百果重相关的QTL分别有13、7、13和8个。单个QTL解释的遗传变异为3.14%~18.27%。在2个环境中重复检测到的QTL有6个, 即荚果长3个, 荚果厚1个, 百果重2个。根据不同性状的QTL是否具有重叠的置信区间, 共检测到9个QTL热点区, 表型贡献率在3.57%~18.27%范围内。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|