第一作者联系方式: E-mail:a09080003@163.com, Tel: 18045476676
为挖掘野生大豆( Glycine sojaL. G07256)耐碳酸盐关键功能基因, 利用前期高通量转录组测序数据, 从构建的碳酸盐胁迫基因表达谱中, 选取了一个碳酸盐胁迫下显著上调表达的肌醇-1-磷酸合酶类基因。采用同源克隆的方法, 获得该基因的全长cDNA, 命名为 GsMIPS2。实时荧光定量PCR结果显示该基因受碳酸盐胁迫诱导表达, 并且其表达量具有组织特异性。将 GsMIPS2基因转化拟南芥, 并结合拟南芥中T-DNA插入突变体 atmips2来验证其耐碳酸盐功能。结果表明, 碳酸盐胁迫条件下, GsMIPS2超量表达拟南芥种子萌发率显著高于野生型, 而拟南芥突变体 atmips2种子萌发率显著低于野生型。上述结果表明, GsMIPS2基因在植物应答碳酸盐胁迫过程中起重要作用。
To identify the key functional genes in response to bicarbonate stress in Glycine soja L. G07256, we constructed a gene expression profile under bicarbonate treatment using high throughput RNA-seq data, from which we selected a myo-inositol- 1-phosphate synthase gene whose expression was significantly induced by bicarbonate stress. By homology-based cloning, we acquired its full length cDNA and termed as GsMIPS2. The results of quantitative real-time PCR demonstrated the bicarbonate stress induced expression of GsMIPS2 and its tissue expression specificity. We also verified the function of GsMIPS2 in bicarbonate responses by using the GsMIPS2 transgenic Arabidopsis combined with the T-DNA insertion line atmips2. We revealed that the germination rate of GsMIPS2 overexpression lines was significantly higher, while that of atmips2 mutant line was much lower than that of wild type under bicarbonate stress. These results indicate the positive role of GsMIPS2 in plant bicarbonate stress responses.
由碳酸盐造成的盐碱逆境严重影响植物的生长和发育, 是限制作物产量和品质的重要因素[1]。土壤盐碱化问题在世界范围内广泛存在[2]。因此, 挖掘耐盐碱关键基因, 应用基因工程技术手段培育耐盐碱作物品种, 对开发利用盐碱地资源、增加耕地面积, 具有重要的理论和现实意义[3]。野生大豆(Glycine sojaL. G07256)是栽培大豆的祖先种, 自然环境下在遗传方面未受到人为选择的影响, 积累了丰富的耐逆基因资源, 因此成为克隆耐盐碱胁迫基因的理想材料[4]。
肌醇是生物体内一种重要的物质, 参与生长调节、激素调节及信号传导等过程[5], 并形成各种肌醇衍生物。有研究表明, 肌醇衍生物松醇能够通过保护细胞结构、减少活性氧积累等方面应答非生物胁迫[6]。肌醇-1-磷酸合成酶(MIPS: EC5.5.1.4)是肌醇合成的限速酶, 它通过将D-葡萄糖-6-磷酸转化为L-肌醇-1-磷酸合成肌醇。Das-Chatterjee等[7]和Patra等[8]分别克隆了野生旱稻(Porteresia coarctata)的肌醇-1-磷酸合酶基因(PcINO1)并将其在烟草中超量表达, 从而显著提高了转基因植物的耐盐性; 鹰嘴豆(Cicer arietinum)肌醇-1-磷酸合成酶基因(CaMIPS2)在拟南芥中异源表达可显著提高其耐盐、耐旱性[9]; 平滑网茅(Spartina alterniflora)肌醇-1-磷酸合成酶基因(SaINO1)在拟南芥中超量表达, 可以通过保护光合系统II提高植物耐盐性[10]。Hegman等[11]分离鉴定了大豆MIPS蛋白编码基因GmMIPS, 发现其参与种子发育过程。然而, Kido等[12]在干旱条件下分析大豆高通量串联基因表达发现, 大豆的MIPS基因并不受干旱诱导表达。
尽管目前对于MIPS基因耐盐方面的研究取得了一定进展, 但对其应答碳酸盐胁迫功能的研究仍然十分匮乏。本研究从实验室前期构建的野生大豆(Glycine soja L. G07256)碳酸盐胁迫下高通量转录组测序数据中, 选取了一个肌醇-1-磷酸合成酶类基因GsMIPS2, 结合超量表达拟南芥及拟南芥突变体, 研究该基因在植物应答碳酸盐胁迫中的功能, 旨在为作物耐碳酸盐转基因育种提供基因资源并奠定理论基础。
耐盐碱野生大豆(Glycine soja L. G07256)由吉林省农业科学院大豆研究所王跃强研究员提供; 野生型拟南芥(哥伦比亚生态型)及突变体(SALK_031685C)购自Nottingham Arabidopsis Stock Centre (NASC, Nottingham, UK)。大肠杆菌DH5α 、根瘤农杆菌GV3101和植物表达载体pCAMBIA3300 35Su均由东北农业大学生命科学学院植物生物工程研究室保存。
利用同源克隆法获得野生大豆GsMIPS2全长cDNA。将饱满的野生大豆种子浸没于98%的浓H2SO4中处理10 min以除去泥膜, 用无菌ddH2O冲洗4~5遍后用1/4 Hoagland培养液进行液体培养, 待幼苗长至3周龄取材, 并于-80℃保存。用RNA提取试剂盒RNAprep Plant Kit (TIANGEN, Beijing, China)提取材料总RNA, 取1.5 μ L总RNA为模板, 以Oligo dT为引物, 采用SuperScript III Reverse Transcriptase Kit (Invitrogen, USA) 合成cDNA第1条链。根据大豆同源基因(Glyma18g 02210.1), 设计GsMIPS2全长基因PCR扩增引物(5° -ATGTTCATC GAGAATTTTAAGGTAGAG-3° 和5° -TCACTTGTAC TCGAGAATCATGTTATTC-3° ), 以野生大豆cDNA为模板, 采用PrimeSTAR HS DNA Polymerase进行PCR扩增。将大小符合要求的电泳条带回收、克隆至pGEM-T载体, 转化大肠杆菌DH5α 后送交测序, 每段序列至少测3个转化子, 分析测序结果以确定GsMIPS2的全长序列。
利用Mega 5.0进行多重序列比对, 并用Neighbor- Joining法构建系统发育树; 利用MEME在线软件(http://meme.nbcr.net/meme/)分析目的蛋白所具有的保守结构域。
将3周龄的野生大豆幼苗, 置含有50 mmol L-1 NaHCO3的1/4 Hoagland培养液中, 分别处理0、1、3、6、12和24 h后迅速剪取根尖部分, 于-80℃保存; 按1.2所述方法提取RNA及合成cDNA第1条链。根据SYBR Green Real-time PCR Master Mix (TOYOBO, Japan)试剂盒操作说明进行实时荧光定量PCR。采用比较CT法(2-Δ Δ Ct)分析结果, 以GAPDH作为内参基因。本试验进行3次独立的生物学重复和3次技术重复, 取3次重复的平均值。原始数据标准化处理的方法参见文献[13]。
取1/4 Hoagland培养液培养的野生大豆各个时期的组织, 包括种皮、根、幼茎、老茎、上胚轴、下胚轴、叶和花, 参见1.3方法提取RNA, 并进行实时荧光定量PCR分析。
利用不依赖酶切位点的植物超量表达载体pCAMBIA330035Su[14], 根据GsMIPS2基因序列设计带U碱基的引物(5° -GGCTTAAUATGTTCATCGA GAATTTAAGG-3° 和5° -GGTTTAAUTCACTTGTAC TCGAGAATCATG-3° ), 转化根瘤农杆菌GV3101, 利用农杆菌介导的Floral dip法[15]对野生型拟南芥进行遗传转化; 通过连续3代的草胺膦筛选(25 μ g L-1)、PCR检测及半定量RT-PCR检测, 确定超量表达目的基因的转基因纯合体株系。
采用PCR技术鉴定拟南芥T-DNA插入突变体atmips2(SALK_031685C)。使用基因特异性引物FP: 5° -TGATCCCACTTCCTGGTATCTATG-3° 及T-DNA特异性引物LB: 5° -GAGATTAACCTATGGCTCCC CTT-3° 验证T-DNA是否插入到基因中; 使用基因特异性引物FP: 5° -TGATCCCACTTCCTGGTATCTAT G-3° 和RP: 5° -CCGGGTGGAAAGAGTGAAAC-3° , 验证突变体是否为T-DNA插入纯合突变体。
将同一批获得的野生型、转基因纯合体及突变体拟南芥种子用5% NaClO消毒6 min, 其间反复振荡, 用灭菌的ddH2O清洗6~8次, 放于4℃避光保存3 d用于萌发期表型分析。将春化后的种子同时播种在添加10 mmol L-1 NaHCO3(pH 8.5)的1/2 MS固体培养基上, 以播种在不含NaHCO3的1/2 MS固体培养基的种子作为对照。由于前3 d种子萌发率变化较快, 因此每0.5 d统计一次萌发率, 3 d后每天统计一次萌发率, 共统计5 d, 于播种第3天拍照。试验保证3次技术重复及3次生物学重复。
通过对高通量转录组测序数据的分析, 从肌醇-1-磷酸合酶家族中选取碳酸盐胁迫下显著上调表达的基因GsMIPS2 (图1)。根据与GsMIPS2同源的大豆基因(Glyma18g02210.1)序列, 设计基因特异性引物, 使用高保真酶进行PCR扩增并测序。结果表明3个转化子的测序结果完全一致, 将测序得到的序列进行ORF finder预测, 最终确定GsMIPS2的全长CDS序列, 大小为1533 bp, 编码510个氨基酸, 经ExPASy预测蛋白质的分子量为56 444.52 Da。在NCBI网站上以GsMIPS2蛋白序列为靶序列进行BlastP比对, 下载比对结果中与GsMIPS2相似度较高的蛋白序列, 使用MEGA 5.0进行多重序列比对, 并构建系统发育树。如图2, 来自多个物种的MIPS蛋白都与GsMIPS2蛋白具有极高的相似性, 与双子叶植物和单子叶植物的相似度分别为88%~100%和83%~86%。系统发育树结果表明, 单子叶与双子叶植物的MIPS蛋白位于不同的分支, GsMIPS2蛋白与大豆GmMIPS2蛋白序列完全一致, 相似度为100%。同时, 通过MEME在线软件结构域预测结合相关文献[16, 17]发现, 在所选取的这几个物种的蛋白序列中都含有“ GWGGNNG” , “ LWTANTER” , “ NGSPQNTFVPGL” 和“ SYNHLGNNDG” 这4个保守的结构域(图3), 并含有由1个Rossman折叠(GXGGXXG)和1个酶催化活性区组成的NAD结合位点。在GsMIPS2蛋白中该催化活性区位于第118~510个氨基酸的区域, 其中对MIPS酶结合底物起到关键作用的氨基酸残基已用红色箭头标出。
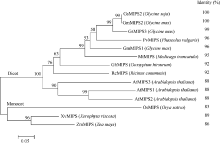 | 图2 GsMIPS2蛋白与单子叶和双子叶植物MIPS蛋白序列相似度及进化树分析Fig. 2 Similarity and phylogenetic tree analysis of GsMIPS2 with MIPS proteins from monocotyledon and dicotyledon |
为研究GsMIPS2在碳酸盐胁迫条件下的表达模式, 采用实时荧光定量PCR分析了GsMIPS2基因在50 mmol L-1 NaHCO3胁迫下, 野生大豆根中的表达特性。结果显示(图4), GsMIPS2基因能被碳酸盐胁迫诱导表达, 并呈现先上升后下降的表达趋势。在50 mmol L-1 NaHCO3处理后3 h达到最高值, 比0 h增加了约18.8倍, 该结果表明GsMIPS2基因参与碳酸盐胁迫过程, 并且在短期内显著上调表达。该结果与高通量转录组测序数据相一致(图1), 同样能够说明GsMIPS2基因受碳酸盐诱导表达。
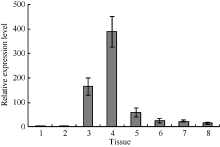
同时, 我们研究了GsMIPS2基因在野生大豆组织中的表达特异性, 采用实时荧光定量PCR分析了GsMIPS2基因在野生大豆种皮、根、幼茎、叶、上胚轴、下胚轴、老茎、花等组织的表达特性。如图5所示, GsMIPS2基因的表达量在野生大豆不同组织中存在着明显的差异, 在野生大豆叶片中的表达量最高, 幼茎次之, 而在其他组织中则相对较低, 说明该基因在野生大豆组织中的分布具有特异性。综上所述, 我们推测野生大豆GsMIPS2基因在植物应答碳酸盐胁迫反应中起着重要的作用, 其组织特异性分布表明该基因行使功能的主要部位可能是在植物叶片中。
为进一步研究GsMIPS2基因的功能, 采用不依赖酶切位点的USER技术构建GsMIPS2基因植物超量表达载体, 载体图谱如图6-A, 并用Flora dip法对野生型拟南芥进行侵染, 经草铵膦(25 μ g L-1)筛选得到T0代抗性植株, 随后进行了连续两代草胺膦筛选及PCR跟踪检测, 对T2代不发生分离的抗性植株视为超量表达纯合体, 共获得3株纯合体植株(#4、#11和#20), 通过进一步半定量RT-PCR检测, 证明这3株植株均为GsMIPS2基因超量表达纯合体(图6-B)。
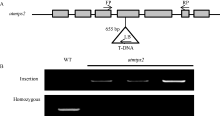
同时对拟南芥中肌醇-1-磷酸合酶2基因(AtMIPS2)的突变体atmips2(SALK_031685C)进行了分子鉴定。如图7-A所示, 该突变体插入位点为第4个外显子655 bp处。通过PCR技术, 利用基因特异性引物FP及T-DNA特异性引物LB进行PCR检测, 野生型拟南芥无条带而突变体有条带, 说明T-DNA已经插入到该基因中; 利用基因特异性引物FP和RP进行PCR检测, 野生型拟南芥有条带而突变体无条带, 则说明该突变体为纯合突变体(图7-B), 可应用于下一步表型试验。
将野生型、突变体及2个GsMIPS2基因超表达量相近的拟南芥株系(#11和#20)的种子经表面消毒后, 播于1/2 MS固体培养基或添加10 mmol L-1 NaHCO3(pH 8.5)的胁迫培养基上, 每12 h统计一次种子萌发率。如图8和图9-A, 在正常生长条件下转基因、突变体与野生型拟南芥种子的萌发状态及萌发率大致相同, 表明导入的GsMIPS2基因并未对拟南芥的生长、发育造成影响。在10 mmol L-1 NaHCO3(pH 8.5)处理下, 转基因、突变体与野生型拟南芥的萌发都受到抑制, 但是与野生型拟南芥相比, 突变体的萌发率受抑制更明显, 而转基因拟南芥2个株系种子的萌发率受NaHCO3胁迫影响的程度明显低于野生型(图9-B), 以上结果初步说明GsMIPS2基因的超量表达增加了拟南芥的碳酸盐耐性, 而atmips2突变体则使拟南芥对碳酸盐胁迫更加敏感。
 | 图8 碳酸盐胁迫对野生型、突变体和GsMIPS2超量表达拟南芥萌发期的影响Fig. 8 Effect of bicarbonate stress on the germination phenotype of wild type, atmips2, and GsMIPS2 overexpression lines |
全世界近932 hm2的土地都正在遭受盐碱胁迫的危害[18], 而通常意义上的碱胁迫主要是由NaHCO3和Na2CO3这两种碳酸盐引起的[19]。碳酸盐胁迫不仅能引起离子胁迫、渗透胁迫、氧化胁迫, 还能产生高pH值, 因此比盐胁迫更具有危害性[20]。然而迄今为止, 对于碳酸盐胁迫的研究相比盐胁迫要少得多。对于肌醇-1-磷酸合成酶的研究表明, 只有少数自身具有胁迫耐受性的植物所编码的MIPS蛋白, 才能够在高温或高盐浓度的环境下维持自身MIPS酶蛋白的催化活性, 从而继续发挥催化作用, 促进肌醇的合成[21, 22], 而这种特性可能与它们在胁迫生长环境下蛋白质的结构特性及其对胁迫环境耐受程度的不同密切相关。针对上述研究结果, 本研究所使用的试验材料野生大豆在长期的自然选择过程中积累了较强的盐碱胁迫耐受性, 其种子能够在pH 9.02的碱性土壤中萌发[23]; 同时, GsMIPS2基因是野生大豆碳酸盐胁迫RNA转录组测序数据中明显上调表达的基因, 因此对该基因的研究为阐明植物耐碳酸盐机制提供潜在的价值与意义。
进化树及结构域分析结果表明, GsMIPS2蛋白与其他物种的MIPS蛋白都有共同的保守结构域及极高的相似性, 这4个结构域可能会形成MIPS酶蛋白的核心结构, 因此在进化过程中高度保守[24, 25, 26]。然而, 蛋白质的功能与其结构密切相关。虽然GsMIPS2蛋白与大豆GmMIPS2蛋白氨基酸序列完全一致, 表明这2种蛋白在一级结构上并没有区别, 但是由于酶类蛋白质在发挥其功能时还与其二级结构、三级结构乃至四级结构有着紧密的联系, 因此, 这2种不同来源的蛋白在发挥催化作用时, 外界胁迫环境对蛋白构象影响程度是否相同还需要进一步研究。而这种构象的不同可能会使GsMIPS2蛋白与GmMIPS2蛋白在胁迫环境下酶的催化活性产生差异。由于目前尚无对非耐盐碱大豆品种在碳酸盐胁迫下MIPS基因功能的研究, 为了验证GsMIPS2基因耐碳酸盐功能是否与野生大豆自身的胁迫耐性相关, 下一步试验还可以将非耐盐碱大豆品种的MIPS基因超量表达拟南芥进行功能验证。
实时荧光定量PCR结果显示, GsMIPS2基因能够迅速受碳酸盐胁迫诱导表达, 并且具有组织特异性, 据此推测该基因在碳酸盐胁迫中发挥一定的功能, 并且其行使功能的主要部位可能是在植物的叶中, 该结论与Hegerman等[11]证明GmMIPS基因在大豆不同组织中的表达量具有明显差异的结果相一致。同时, 我们结合相关文献[27], 对野生型、突变体及超量表达株系在不同碳酸盐浓度下(9、10和11 mmol L-1 NaHCO3)萌发期的表型进行了浓度梯度预试验, 发现在10 mmol L-1 NaHCO3(pH 8.5)处理下萌发率差异最显著, 最终确定采用10 mmol L-1 NaHCO3(pH 8.5)进行试验。在碳酸盐胁迫下GsMIPS2基因超量表达拟南芥的萌发率明显好于野生型, 该现象从表型上初步说明GsMIPS2基因在植物碳酸盐耐性方面起着积极的作用。由于野生大豆中不易获得GsMIPS2基因敲除突变体, 我们采用了与野生大豆GsMIPS2基因相似度较高(88%)的拟南芥AtMIPS2基因突变体进行验证。结果表明, 在碳酸盐胁迫下拟南芥突变体atmips2的萌发率明显低于野生型, 该现象可以部分反映GsMIPS2基因在碳酸盐胁迫下的功能。然而, 虽然野生大豆GsMIPS2基因序列和拟南芥AtMIPS2基因序列相似度较高, 但是由于这2个基因来源不同, 后续试验还需要将GsMIPS2基因超量表达到拟南芥突变体中, 通过功能互补实验来进一步验证GsMIPS2基因的功能。
肌醇-1-磷酸合成酶是肌醇合成的关键酶, 肌醇在植物应答盐、干旱等非生物及病虫害等生物胁迫方面有着重要的功能[28, 29]。Raboy等[30]研究拟南芥中的MIPS基因家族(MIPS1, MIPS2, MIPS3)发现, MIPS1和MIPS2基因的突变体mips1和mips2能够降低植物种子中肌醇的含量; Saxena等[31]发现拟南芥超量表达鹰嘴豆(Cicer arietinum L.)的CaMIPS基因会引起肌醇含量的增加, 从而提高拟南芥种子萌发期对冷、盐、干旱等胁迫的耐性; Kaur等[9]同样发现, 受ABA诱导的鹰嘴豆CaMIPS2基因的超量表达, 增加拟南芥对盐及干旱胁迫的耐性, 与野生型相比, 该过程伴随着MIPS2酶活性的提高及肌醇含量的增加。针对上述研究结果, 后续试验还需要验证GsMIPS2基因的超量表达是否会增加肌醇的含量, 从而解释GsMIPS2基因提高植物碳酸盐耐性的生物学机制。
筛选出一个转录水平明显上调的肌醇-1-磷酸合酶类基因GsMIPS2, 该基因能被碳酸盐胁迫诱导表达, 并且具有组织特异性。将该基因超量表达到拟南芥中能够提高拟南芥萌发期的碳酸盐耐性。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|