第一作者联系方式: E-mail:lhzh66666@163.com, Tel: 0371-65751589
以栽培大豆晋豆23为母本, 以山西农家品种大豆灰布支黑豆为父本杂交衍生的447个RIL作为供试群体构建遗传图谱, 利用高效液相色谱法定性、定量测定样品中的异黄酮及其组分含量。采用主基因+多基因混合遗传分离分析法和WinQTLCart 2.5复合区间作图法, 对大豆异黄酮及其组分含量进行混合遗传分析和QTL定位。结果表明, 大豆苷、黄豆苷元、染料木素、染料木苷、大豆苷元和异黄酮总含量分别受4、4、2、3、2和2对主基因控制, 并有多基因修饰。检测到44个与大豆异黄酮及其组分含量相关的QTL, 与大豆苷、染料木素、黄豆苷元、大豆苷元、染料木苷和异黄酮总含量相关的QTL分别有10、9、4、7、8和6个。连续2年分别检测到与大豆苷、染料木苷、黄豆苷元和异黄酮关联, 分别位于标记区间satt430~satt359、satt038~satt570、satt197~sat_128和satt249~satt285的稳定表达QTL, 可尝试用于分子标记辅助育种。
A set of 447 recombinant inbred lines (RILs) derived from the cross between cultivars Jingdou 23 (female parent) and Huibuzhi (semi-wild, male parent) was used to construct a new map. Isoflavone content and its components were quantitatively and qualitatively evaluated by using high performance liquid chromatography (HPLC). We analyzed inheritance and detected QTLs for isoflavone content and its components in soybean seeds using major gene plus polygene mixed inheritance analysis and WinQTLCart 2.5 composite interval mapping. The results showed that daidzin, daidzein, genistein, genistin, glycitin, and total isoflavone contents were controlled by four, four, two, three, two and two main-genes, respectively. However, polygene effects were not detected in the study. Forty-four quantitative trait loci (QTLs) for isoflavone contents and its components were mapped, including ten for daidzin, nine for genistein, four for daidzein, seven for glycitin, eight for genistin, and six for total isoflavone content. The stable QTLs related to daidzin, genistin, glycitin, isoflavone content were respectively detected to be located in the intervals of satt430-satt359, satt038-satt570, satt197-sat_128, and satt249-satt285 during two years, which could be used in marker-assisted selection (MAS) for soybean breeding.
大豆异黄酮是大豆种子中积累的一类次生代谢产物。它的组成和存在形式主要包括染料木素、黄豆苷元、大豆苷元、染料木苷和大豆苷等。大豆异黄酮具有特殊的生物效能, 具有弱雌激素活性、抗氧化活性、抗溶血活性和抗真菌活性, 能有效预防和抑制白血病、骨质疏松、结肠癌、肺癌、骨癌、胃癌、乳腺癌、前列腺癌、妇女更年期综合症等多种疾病的发生, 尤其是对乳腺癌和前列腺癌有良好的预防和治疗作用[1, 2, 3, 4]。染料木素和黄豆苷元对抑制癌细胞的增殖具有特异作用[5, 6, 7, 8, 9]。自然界中异黄酮资源十分有限, 大豆是唯一含有异黄酮的食物资源, 但含量普遍较低[10], 提高大豆异黄酮含量是解决该问题的关键。研究大豆异黄酮及其组分含量的遗传与QTL分析, 对于推动采用分子标记辅助育种手段选育高异黄酮含量的大豆品种, 具有十分重要的的现实意义。国内外学者对大豆异黄酮及其组分含量进行了遗传分析和初步QTL定位研究。Li等[11]对大豆异黄酮与生态因子关系进行了研究, Liang等[12, 13, 14]对大豆异黄酮及其组分含量的配合力和杂种优势以及大豆籽粒异黄酮遗传效应和基因定位研究发现, 大豆鼓粒成熟期较低的均温和较大的昼夜温差有利于异黄酮的积累; 在出苗期较多的日照和较低的均温有利于异黄酮的形成。异黄酮含量和黄豆苷元既受加性效应又受非加性效应的控制, 染料木苷主要受非加性效应控制, 染料木素、大豆苷元和大豆苷这3个成分的遗传主要受加性效应控制。大豆异黄酮主要受制于母体遗传效应, 其次为胚基因效应, 细胞质效应影响较小。不同遗传体系的异黄酮基因主效应明显大于环境互作效应。利用Essex× Forrest、AC756× RCAT Angora、Essex× PI4375、Williams 82× Essex、Zhongdou27× Jiunong 20、Peking× Tamahomare、Hwangkeum× IT182932、Xiaoheidou× GR8836、LHD2 × NHZ等RIL群体, 已定位了一批QTL, 基本上覆盖了大豆所有的基因组[15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26]。其中, Meksem等[15, 16]利用Essex× Forrest 100个株系的重组自交系(Recombinant Inbred Lines, RIL)及结合150个多态性DNA标记, 定位了12个异黄酮及其组分含量的QTL, 分别位于第3、第5、第9、第11和第12染色体上; 11连锁群上与染料木苷含量紧密连锁的主效QTL可解释的遗传变异率高达50.2%; 3连锁群上与染料木苷和大豆苷含量紧密连锁的主效QTL可解释的遗传变异率分别为11.1%和10.3%; 5连锁群上与大豆苷含量紧密连锁的主效QTL可解释的遗传变异率为9.6%。Primomo等[17]利用AC756× RCAT Angora 207个株系F4:6的重组自交系群体检测到17个异黄酮及其组分含量的QTL, 分别定位在第1、第3、第5、第6、第7、第9、第12、第13、第16和第18染色体上; Zhang等[25]利用184个Xiaoheidou× GR8836株系的F2:9-11重组自交系群体(RIL), 检测到22个异黄酮及其组分含量的QTL, 分别定位在第3、第4、第7、第9、第10、第12、第13和第17染色体上, 遗传贡献率在4.48%~7.87%之间。检测到3对影响异黄酮含量上位性QTL, 其中, qEGp1和qEGw1影响黄豆黄苷含量, qETISOp1影响大豆异黄酮总量。张晶莹等[26]利用LHD2 × NHZ重组自交系群体(RIL), 检测到14个异黄酮及其组分含量的QTL, 分别定位在第3、第4、第5、第7、第9、第11、第12、第14、第15、第17和第20染色体上。目前, 国内外已检测到的大豆异黄酮及其组分含量QTL, 因遗传背景和环境条件的不同, 表现稳定的较少, 因此大豆异黄酮分子标记辅助育种受到限制。本研究以晋豆23× 灰布支黑豆衍生的447个RIL为试验材料, 利用WinQTLCart2.5[27] (http://statgen. ncsu.edu/qtlcart/WQTLCart-1.htm)混合线性模型分析方法, 对大豆异黄酮及其组分含量进行QTL定位分析; 采用主基因+多基因混合遗传分离分析方法[28], 分析大豆异黄酮及其组分含量遗传规律, 对其遗传基础全面剖析, 为高异黄酮大豆分子标记辅助育种提供参考依据。
作图亲本材料为晋豆23× 灰布支黑豆。母本晋豆23是山西省主栽大豆品种, 丰产性好; 父本灰布支黑豆是山西省农家品种, 具有较多的原始性。作图群体采用单粒混传法(single seed mutiple descent, SSD) RIL。1998年夏季在山西省杂交获得F0种子, 种植后收获一株种子产量732粒的F1单株, 1998年10月在海南岛单粒种成732个F2, 1999年2月单株收获每个F2形成F3株系。从F3代开始, 每个世代每个株系随机收获10株上的共20个豆荚, 每荚约2粒, 共约40粒种子, 随机选取其中的20粒种成20个株行, 形成下一个世代, 以此类推至高世代。
田间试验采取随机区组设计, 2次重复, 行长4.0 m, 行距0.4 m。2011年和2012年分别在河南省农业科学院现代农业科技试验示范基地(河南省原阳县)种植。田间管理按照大田模式进行, 周边设保护行, 一播全苗。
从每个家系选取20 g大豆籽粒, 用旋风磨(Retsch ZM100, Φ = 1.0 mm, Rheinische, Germany)磨粉; 准确称取100 mg大豆粉放入带有螺帽的有机玻璃试管, 加入5 mL含0.1% (v/v)乙酸的70% (v/v)乙醇水溶液; 室温下振荡提取12 h; 2795× g离心10 min, 上清液经0.2 μ m 滤膜过滤, 4℃冰箱保存备用。采用津岛高效液相色谱仪(LC-6A), 定性、定量测定样品异黄酮及其组分含量[27, 28]。手动进样, 进样量为10 μ L。色谱柱为YMC-Pack, ODS-AM-303, 250.0 mm × 4.6 mm I.D.; 柱温35℃; 流动相A为含0.1% (v/v)乙酸的超纯水, B为含0.1% (v/v)乙酸的乙腈水溶液; 梯度洗脱范围为13%~35%; 运行时间为70 min; 流速为1.0 mL min-1; 检测波长为260 nm。
根据标准样品的保留时间和最大吸收光谱定性, 以大豆苷(daidzin, D)在260 nm波长的紫外吸收值为基础, 参照Sun等[29]的方法计算样品中染料木素(genistein, G)、黄豆苷元(glycitein, GL)、大豆苷元(daidzein, DA)、染料木苷(genistin, GE)、大豆苷(daidzin, D)以及异黄酮总含量(isoflavone, IF), 利用SPSS 19.0 (SPSS, Chicago, USA)完成异黄酮及其组分含量的方差分析、相关分析和描述统计。
王珍[30]使用该RIL群体为材料, 构建了一个含有227个SSR标记的大豆遗传连锁图谱。以该图谱和大豆公共图谱中的标记为基础, 梁慧珍[31]重新整合, 整合后图谱全长2047.6 cM, 包括27个连锁群232个标记位点。该图谱在9(K)、13(F)和16(J) 3条染色体上, 均出现了2个间隙, 在12(H)染色体出现了1个间隙, 因而形成了27个连锁群。本研究采用梁慧珍[31]整合后的大豆分子遗传图谱对大豆异黄酮及其组分含量进行QTL定位分析。把不同年份作为环境因子处理, 根据2个环境的表型数据, 利用WinQTLCart 2.5软件中复合区间作图法(CIM) Zmapqtl方法的Model 6, 每2个cM对各性状进行全基因组扫描, 以确定各性状QTL数目及其在染色体上的位置, 通过逐步回归指定解释给定性状最大变异的5个标记作为余因子(co-factor), 选取临界阈值LOD=2.5, 检测每个环境下的QTL效应。当LOD≥ 2.5时, 认为QTL存在; 如果临近位点间图距小于5 cM, 就初步认定是同一个QTL。采取McCouch等[32]方式命名。
采用主基因+多基因混合遗传分离分析法[28], 分析晋豆23× 灰布支黑豆及其衍生的RIL群体, 通过极大似然法和IECM对混合分布的各世代、各成分分布的参数作出估计, 通过AIC准则和一组适合性测验, 从中选出最适遗传模型, 利用其各成分分布参数通过最小二乘法估计相应的遗传参数。主基因遗传率h2mg(%) = σ 2mg/σ 2p, 多基因遗传率h2pg(%) = σ 2pg/σ 2p; 环境遗传率h2e(%) = σ 2e/σ 2p。其中σ 2p为群体表型方差; σ 2mg为主基因遗传方差; σ 2pg为多基因遗传方差; σ 2e为多环境遗传方差; 各遗传方差间的关系为σ 2p = σ 2mg+σ 2pg+σ 2e。
观察大豆苷、染料木素、染料木苷、黄豆苷元、大豆苷元和异黄酮总含量6个性状在分离群体中的分离并进行方差和遗传力分析(表1)表明, 2个亲本6个性状在后代分离群体中表现出较大的分离程度, 在不同环境中均存在较大差异, RIL群体的最小值和最大值之间差异明显。各性状的广义遗传力(h2= Vg/VP)分别为35.28%、23.62%、37.15%、29.67%、21.01%和19.26%, 为QTL分析提供了较好的遗传背景。表1中两个环境下异黄酮及其组分含量共6个性状表型变异系数为0.339~0.568, 方差分析均呈现极显著差异, 表明异黄酮及其组分含量受遗传和环境因素的共同影响。同时, 从RIL群体的性状变异范围来看, 6个性状都有超越双亲的株系出现, 说明大豆异黄酮及其组分含量遗传基因分布在双亲中, 通过杂交可以得到超亲分离的株系。
| 表1 RIL群体亲本和群体家系性状描述统计与方差分析 Table 1 Descriptive statistics and analysis of variance for isoflavone contents in the RIL populations and their parents |
表2表明, 不同环境条件下的相关分析结果基本一致。除黄豆苷元外, 异黄酮总量与其余4个组分含量均表现出显著或极显著正相关, 且这4个组分含量间均表现出显著或极显著正相关。说明各性状之间存在着既相互影响又相互制约的关联关系。相关分析结果与异黄酮及其组分含量在RIL群体中的分离及遗传分析结果基本一致。
| 表2 6个性状之间的相关分析 Table 2 Phenotypic correlation coefficients among six traits in the RIL populations and their parents |
选用2011年和2012年试验数据的平均值进行主基因+多基因混合遗传分析, 依据AIC值小为最佳的准则结合Kolmogorov试验、均匀检验性检验和Smirnov检验选定最适遗传模型, 用最小二乘法估计异黄酮及其组分含量的遗传参数(表3)。
| 表3 6个性状最适模型及遗传参数估计结果 Table 3 Analysis of the best models and genetic parameters for six traits |
(1) 大豆苷最适遗传模型符合4MG-AI, 即4对主基因加性-加性× 加性上位性遗传模型。4对主基因间加性效应分别为-1.390、-0.564、0.042和-0.044; 主基因之间相互作用的上位性效应分别为2.176、2.207、0.403、2.210、1.272和-0.201。第3对主基因对第4对主基因(icd)上位性效应为负值, 与其他基因间上位性效应相反, 说明不同基因间上位性效应对大豆苷性状的影响并不完全一致。主基因遗传率为98.34%, 没有检测到多基因效应, 环境因素引起的变异为1.66%, 表明大豆苷主要受4对主基因影响, 受环境因素影响较小。
(2) 染料木素最适遗传模型符合MX2-DominanceI- A, 即2对显性上位主基因-加性多基因混合作用遗传模型。2对显性上位主基因间相互作用的加性效应分别为2.052和10.593。多基因间加性效应为-7.436, 其主基因遗传率为67.15%, 多基因遗传率为32.70%, 说明染料木素存在多基因效应, 其中主基因效应更大。
(3) 染料木苷最适遗传模型符合MX3-AI-AI, 即3对加性-上位性主基因× 加性-上位性多基因混合遗传模型。3对主基因间加性效应分别为-3.045、-1.072和-2.284。3对基因中每2对基因间加性效应之间的上位性互作效应分别为0.412、-0.206和0.719, 3对基因间上位性互作效应(iabc)为0.059, 表明3对基因之间存在着明显上位性互作, 并且互作后的上位性效应不同。正向的上位性效应表示两座位间互作基因型与具有正效应的加性基因型方向相同, 负向的上位性效应则表示两座位间互作基因型与具有正效应的加性基因型方向相反。3对主基因遗传率为97.75%, 多基因遗传率为2.16%, 说明染料木苷存在多基因效应, 其中以主基因效应为主。
(4) 黄豆苷元最适遗传模型符合4MG-AI, 即4对主基因加性-加性× 加性上位性遗传模型。4对主基因间加性效应分别为1.687、1.364、0.821和0.487。4对主基因加性效应值均为正值, 表明基因来源于母本晋豆23; 4对主基因间加性效应主基因之间相互作用的上位性效应分别为1.410、1.106、0.734、1.496、0.670和0.287, 说明不同基因间上位性效应对黄豆苷元性状的影响方向一致。主基因遗传率为99.53%。没有检测到多基因效应。环境因素引起的变异为0.47%。表明黄豆苷元主要受4对主基因影响, 受环境因素影响较小。
(5) 大豆苷元最适遗传模型符合MX2-Inhibiting- A, 即2对抑制作用主基因-加性多基因混合遗传模型。因为2对主基因间的抑制作用, 只检测到两对主基因间上位性效应(iab)为4.346, 多基因的加性效应为-0.755, 主基因遗传率为54.44%, 多基因遗传率为45.47%, 说明大豆苷元存在多基因效应, 其中主基因效应和多基因效应基本相当。
(6) 异黄酮总含量最适遗传模型符合MX2-AI-A, 即2对加性-上位性主基因× 加性多基因混合遗传模型。2对主基因间加性效应分别为-2.531和-9.147。2对基因加性效应值均为负值, 表明基因来源于父本灰布支黑豆。2对基因间加性效应之间的上位性互作效应为3.210, 存在着明显上位性互作。主基因遗传率为50.24%, 多基因遗传率为49.14%, 说明异黄酮总含量存在多基因效应, 其中主基因效应和多基因效应基本相等。
利用Windows QTL Cart 2.5软件, 共检测到42个控制大豆异黄酮及其组分含量的QTL, 分别位于5(A1)、8(A2)、11(B1)、14(B2)、17(D2)、13(F_1)、18(G)、20(I)、16(J_1)、9(K_2)、19(L)、3(N)和10(O)染色体上(表4和图1), 解释的表型变异范围为3.74%~55.20%。加性效应值正负表现不一, 加性效应值为正, 说明该QTL大豆异黄酮及其组分含量起正向作用, 等位基因来自母本晋豆23, 加性效应值为负, 说明该QTL大豆异黄酮及其组分含量起负向作用, 定位在该染色体上的等位基因来自父本灰布支黑豆。
| 表4 6个性状QTLs位置及其参数 Table 4 QTL positions and its parameters for six traits |
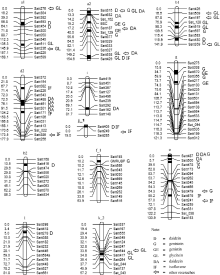 | 图1 检测到的QTL及加性效应QTL在连锁群上的分布以及与以往研究结果的对比Fig. 1 Distribution of main QTLs and additive QTLs on linkage groups and comparison of QTLs detected in this study with those in previous studies |
共检测到9个大豆苷QTL, 分别位于5(A1)、8(A2)、11(B1)、14(B2)、16(J_1)、9(K_2)、19(L)、03(N)和10(O)染色体上, 解释的表型变异范围为3.74%~17.05%。其中2011年和2012年均检测到qD-B1-1定位在11(B1)连锁群标记satt430~satt359间124.87 cM处, 解释的表型变异分别为14.54%和15.39%, 说明这是一个能够稳定遗传的QTL。qD-A2-1和qD-A2-2均被定位在8(A2)连锁群上, 但2个QTL位置相距较远, 说明8(A2)连锁群上可能存在多个与大豆苷相关的基因。位于16(J_1)染色体satt405~satt249间的qD-J_1-1, 表型贡献率为17.05%, 解释的表型变异最大。
共检测到9个染料木素QTL, 分别位于8(A2)、11(B1)、13(F_1)、18(G)和10(O)染色体上。解释的表型变异范围为7.26%~37.43%。位于08(A2)染色体satt187~sat_129间的qGE-A2-1, 表型贡献率为37.43%, 解释的表型变异最大。2011年检测到的qG-G-1和2012年检测到的qG-G-2均被定位在18(G)染色体上, 位置仅相距3.81 cM, 被定位在Satt038标记的两侧, 解释的表型变异均为35.16%, 很可能是同一个QTL。2011年检测到的qG-A2-1和2012年检测到的qG-A2-2均被定位在8(A2)染色体上, 解释的表型变异分别为37.43%和17.06%。2个QTL 位置相距较远, 很可能不是同一个QTL, 说明8(A2)连锁群上可能存在多个与染料木素相关的基因。
共检测到4个染料木苷QTL, 分别位于8(A2)和18(G)染色体上。解释的表型变异范围为25.42%~ 39.59%。2011年检测到的qGE-G-1和2012年检测到的qGE-G-2均被定位在18(G)染色体上, 且均被定位在Satt038~satt570标记区间, 解释的表型变异分别均为39.59%和38.92%, 尽管位置相距16 cM, 仍怀疑二者是同一QTL。qGE-G-1表型贡献率为39.59%, 解释的表型变异最大。
共检测到7个黄豆苷元QTL, 分别位于5(A1)、8(A2)、11(B1)和9(K_2)染色体上, 解释的表型变异范围为4.06%~41.42%。2011年检测到的qGL-B1-1和2012年检测到的qGL-B1-2均被定位在11(B1)染色体上, 且均被定位在satt197~sat_128标记区间, 解释的表型变异分别均为41.42%和41.12%, 但位置相距16 cM, 是否是同一QTL有待进一步验证。2011年检测到的qGL-A2-1和2012年检测到的qGL-A2-2均被定位在8(A2)染色体上, 位置仅相距5.77cM, 且被定位在Satt538标记的两边, 解释的表型变异分别为8.99%和7.32%, 有可能是同一个QTL。qGL-B1-1表型贡献率为41.42%, 解释的表型变异最大。
共检测到8个大豆苷元QTL, 分别位于8(A2)、17(D2)、20(I)和10(O)染色体上, 解释的表型变异范围为5.09%~32.02%。2011年检测到的qDA-D2-1和qDA-D2-2仅相距3.89 cM, 且被定位在satt082标记的两边, 解释的表型变异分别为32.02%和30.41%, 很可能是同一个QTL; qDA-D2-2和qDA-D2-3, 尽管位置仅相距2.85cM, 但被定位在不相邻的2个标记区间, 很可能不是同一个QTL。上面检测到的2011年3个QTL和2012年检测到的qDA-D2-4均被定位在17(D2)染色体上, 但相距相对较远, 很可能也不是同一QTL。说明Gm17(D2)连锁群上可能存在多个与大豆苷元相关的基因。qDA-D2-1表型贡献率为32.02%, 解释的表型变异最大。
共检测到5个异黄酮QTL, 分别位于8(A2)、17(D2)、16(J_1)和10(O)染色体上, 解释的表型变异范围为12.14%~55.20%。2011年和2012年均检测到qIF-J_1-1且定位在16(J_1)连锁群satt249~satt285标记区间0.01 cM处, 解释的表型变异分别为55.14%和55.20%, 说明这是一个能够稳定遗传的QTL。2011年检测到的qIF-D2-1和2012年检测到的qIF-D2-2均被定位在17(D2)染色体上, 但相距较远, 很可能不是同一QTL。说明17(D2)连锁群上可能存在多个与异黄酮相关的基因。qIF-J_1-2表型贡献率为55.20%, 解释的表型变异最大。
本研究通过对2个环境下大豆异黄酮及其组分含量方差分析, 均呈现出极显著差异, 表明异黄酮及其组分含量受遗传和环境因素的共同影响。两环境下, 虽然同一家系大豆异黄酮及其组分含量有所变化, 相关性分析中各环境下均表现出显著或极显著正相关的趋势, 说明遗传是主要决定因素, 控制这些性状的基因存在着紧密连锁, 异黄酮及其组分含量间的相关比较稳定, 对其中一种组分选择, 即可间接达到对其他组分的选择。该研究结果与Chiari等[33]和张晶莹等[26]相一致。同时, 从RIL群体的性状变异范围来看, 6个性状都有超越双亲的株系出现, 说明2个亲本都具有正效等位基因, 研究结果与Gutierrez-Gonzalez等[20]的报道一致。本研究中通过对异黄酮及其组分含量广义遗传力分析, 发现各个性状遗传力均不超过40%, 同一家系试验材料在同一地点, 不同年份间的表型有很大差异, 说明异黄酮及其组分含量是易受环境影响的性状, 从而影响到定位结果。在进行QTL定位分析时, 可以进一步采取BLUE (Best Liner Unbiased Estimator, 最佳线性无偏估计)或BLUP (Best Liner Unbiased Predictor, 最佳线性无偏预测)等统计分析方法, 尽量减少环境误差造成的影响。
基因多效性或者基因紧密连锁引起性状相关, 基因连锁或一因多效导致了不同性状间的相关性, 表现出同一个标记区间内检测出同时控制不同性状的QTL[34]。本研究所定位到的42个与大豆异黄酮及其组分含量相关的QTL中, 在8(A2)、17(D2)、18(G)、16(J)和10(O)染色体上有7个标记在多环境中均被检测到, 异黄酮及其组分含量QTL存在共位性(表5), 表明各性状QTL间存在着关联。如在大豆第16染色体上的标记satt249与D和IF性状相关, 其中与IF相关的QTL在2个环境中均被检测到。以往研究中, 该标记与大豆蛋白质7S相关[35], 而大豆异黄酮与蛋白质呈显著负相关[14], 说明该标记与大豆品质性状密切相关, 在大豆异黄酮与蛋白质形成与积累过程中, 二者存在相互制约的关系, 最后达到总体的平衡。另外, 在第11条染色体上, 2个环境下均在标记satt359定位到与大豆苷相关的QTL, 王英等[36]检测到该标记与大豆株高的光周期敏感度(PS)相连锁, 与短日处理比较, 长日照可增加株高, 降低蛋白质含量。以往研究未见报道该标记与异黄酮主要组分相关, 这可能是遗传背景不同所致。因此, 该标记可能是控制异黄酮主要组分大豆苷的新QTL, 其作用有待进一步研究证实。
| 表5 各年份重复检测到的与异黄酮及其组分含量相关的区间 Table 5 Stable marker interval for isoflavone contents and its components during two years |
本研究中检测出很多贡献率较高的QTL, 但其定位的区间是相邻的。如2011年检测到的qG-G-1和2012年检测到的qG-G-2均被定位在18(G)染色体上, 仅相距3.81 cM, 但被定位在相邻的标记区间, 解释的表型变异均为35.16%, 是否是同一QTL尚不能确定。下一步可以对两年群体数据进行联合分析, 进一步探讨产生这种现象的可能原因。同时, 对有一些两年试验同时检测的QTL, 特别效应值比较大的QTL, 如qIF-J_1-1在2个环境中同时被检测到, 且贡献率均超过55%, 希望进一步确认该基因位点, 并精细定位, 为分子标记辅助育种奠定基础。
Janse[37]发现, 同时对多年间不同环境下的数据进行分析, 能增大QTL的检测强度, 提高QTL的位置和效应的准确程度, 更有利于挖掘稳定的QTL。本研究所采用的RIL群体包含474个家系, 群体数量相对较大, QTL定位的精确性和稳定性相对较高。本次试验中, 连续2年检测到satt430~satt359标记区间与大豆苷相关, satt038~satt570标记区间与染料木苷相关, satt197~sat_128标记区间与黄豆苷元相关, satt249~satt285标记区间与异黄酮相关, 表明这些QTL有可能是稳定遗传的, 所定位的异黄酮及其组分含量相关区间是相对准确和可信的。同时, 也可能是因为标记区间范围相对较大, 在标记期间内存在较多的相关基因, 各组分之间存在相对稳定的相关, 反映了各性状之间相关的遗传特性。同时, 本研究与以往研究相比, 尽管试验地理环境条件不同, 研究群体不同, G、GL、DA和IF定位结果均部分吻合, 但不完全统一(表6和图1)。例如, 与Primomo等[17]相比, GL均被定位在9(K)染色体、DA均被定位在8(A2)染色体、IF均被定位在16(J)染色体satt249标记上; 与Yoshikawa等[23]相比, G均被定位在8(A2)染色体、GL均被定位在5(A1)、8(A2)和11(B1)染色体; DA均被定位在8(A2)和17(D2)染色体、IF均被定位在8(A2)和17(D2)染色体上; 与Kassem等[38]相比, GL均被定位在11(B1)染色体satt197标记上等。不同年份间试验均在同一条染色体上检测到QTL, 如IF在2011年和2012年均被定位在16(J_1)染色体上, 且均被定位在satt249~satt285区间的同一位点上; D在2011年和2012年均被定位在11(B1)连锁群标记satt430~satt359间, 且均被定位在124.87 cM处。与前人研究结果相比, IF均被定位在16(J)染色体satt249标记、GL均定位在11(B1)染色体satt197标记上, 说明这2个QTL能够稳定表达。
| 表6 QTL定位结果的比较 Table 6 Comparison of QTLs detected in this study with in previous studies |
本研究采用主基因+多基因混合遗传分离分析法对大豆异黄酮及其组分含量进行混合遗传分析表明, 大豆苷、黄豆苷元、染料木素、染料木苷、大豆苷元和异黄酮总含量分别受4、4、2、3、2和2对主基因控制, 并有多基因修饰。王春娥[39]以科丰1号与南农1138-2及其构建的184个重组自交系群体(NJRIKY)籽粒为材料, 对12种大豆异黄酮组分含量及其总含量进行了分离分析, 认为大豆籽粒异黄酮总含量和各组分含量的遗传均属2~3对主基因加多基因混合遗传模型, 主基因遗传率为16.42%~ 45.08%, 多基因遗传率为54.78%~83.44%。对比研究结果, 多数结论吻合, 少数结论不同, 有待下一步深入研究。但是, 本研究中也出现了QTL定位数目与遗传模型预测的数目不完全一致的现象。如大豆苷元最适遗传模型符合MX2-Inhibiting-A, 2次试验均定位出4个主效QTL, QTL定位分析结果与遗传分离分析结果预测值不一致。导致这种情况的可能原因有: (1)本研究选用的模型分离分析方法是4对主基因加多基因混合遗传模型[40], 该模型建立时只考虑了主基因间无连锁的情况, 对于主基因间有连锁的情况没有分析; (2)该遗传模型建立过程中, 成分分布数和待估参数过多, 分析过程受到限制, 需要进一步改进参数估算方法, 提高估算结果的准确性; (3)与QTL定位时选用的图谱质量有关。图谱本身存在不完善的地方。尽管研究结果还存在着不足, 仍然为大豆异黄酮及其组分含量的分子研究和遗传分析提供了有益参考。
大豆苷和黄豆苷元主要受4对主基因控制; 染料木素受2对显性上位主基因-加性多基因混合作用控制; 染料木苷主要受到3对主基因控制; 大豆苷元受2对抑制作用主基因-加性多基因控制; 异黄酮总含量受2对加性-上位性主基因× 加性多基因控制。检测到44个与大豆异黄酮及其组分含量相关的QTL, 与大豆苷、染料木素、黄豆苷元、大豆苷元、染料木苷和异黄酮总含量相关的QTL分别有10、9、4、7、8和6个。连续2年检测到satt430~satt359标记区间与大豆苷相关, satt038~satt570标记区间与染料木苷相关, satt197~sat_128标记区间与黄豆苷元相关, satt249~satt285标记区间与异黄酮相关。经比对文献, 认为位于Gm16(J)上的satt249和位于Gm11(B1)上的satt197附近存在稳定遗传的QTL, 分别控制异黄酮和黄豆苷元含量。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|


