第一作者联系方式: E-mail: sunziqi777@163.com, Tel: 0371-65718247
利用14个SSR标记构建了河南省2015年之前选育并审定的90个花生品种的DNA指纹图谱, 用14个SSR标记产生的95个多态性位点可将90个花生品种完全区分开, 其中84个品种间有≥2个位点的差异, 在剩余的3对品种中, 每对仅有1个差异位点。聚类分析结果表明, 在遗传相似系数0.98处, 90个花生品种被聚集成88类, 有2对品种分别聚集在一起, 是由于它们每一个品种分别以另一个品种作亲本选育而成, 仅有1个差异SSR位点, 表明所构建的指纹图谱是有效的。以遗传相似系数0.95为划分标准, 有74.4%的品种具有特异性, 与其他作物相比, 河南省育成花生品种总体上亲缘关系相对较近。根据60个SSR标记的群体结构分析, 90个花生品种可以分为3个亚群, 与根据分枝开花习性和荚果类型的分类相吻合, 亚群划分情况与聚类分析结果基本一致。
The DNA fingerprints of 90 peanut varieties released in Henan province before 2015 were generated using 95 polymorphic loci of 14 SSR markers. All varieties were distinguished by the DNA polymorphisms except for three pairs of varieties with only one SSR locus difference between each pair. SSR clustering analysis revealed that the 90 peanut varieties could be classified into 88 types at a 0.98 genetic similarity coefficient. The remaining two variety pairs had only one allele difference since one variety was the maternal parent of the other. About three fourth (74.4%) of the peanut varieties were distinctive from other varieties at a genetic similarity coefficient threshold of 0.95, indicating a very relatively narrow genetic background of the released peanut varieties in Henan province compared to other crops. Population structure analysis based on 60 SSR markers demonstrated that the 90 varieties could be divided into three subgroups in accordance with the classification according to botanical and pod characteristics, which was consistent with the results of SSR markers cluster analysis. This study provides some fundamental information for selection of parental lines, identification and evaluation of new varieties, as well as establishment of DNA fingerprinting standard in peanut breeding program.
花生(Arachis hypogaeaL.)是世界第四大油料作物, 具有重要的经济价值和营养价值。近年来, 随着国家对油料生产的日益重视以及人民生活水平的不断提高, 我国花生生产呈稳定上升趋势。河南省是我国第一花生生产大省, 面积和产量分别占全国的22%和27%。花生新品种选育和推广应用, 对河南省花生生产发展产生了重要的推动作用。从1982年我国实行品种审定制度至2014年, 河南省育成并审定的花生品种达90个, 这些品种的推广促进了河南省花生生产的快速发展。
随着作物遗传改良的进程, 新育成农作物品种的遗传多样性日益狭窄。了解河南省育成花生品种的遗传多样性, 对于明确花生种质资源的遗传变异, 指导花生育种中亲本组合选配有重要意义。同时, 随着新品种日益增多的推广应用, 品种识别与鉴定, 保证品种的真实性和纯度, 维护生产者与育种者的利益越发重要。传统的品种识别主要以鉴定形态性状为主, 鉴定周期长, 鉴定结果受环境影响大, 而且花生的遗传基础相对狭窄[1], 形态相似性高, 单纯依靠形态性状难以有效区分和鉴定不同品种。DNA指纹图谱的发展, 为品种鉴定提供了新的途径。目前构建DNA指纹图谱的常用分子标记包括RFLP、RAPD、AFLP、SSR、SRAP等[2]。
目前, 已有多家单位利用AFLP或SSR标记构建了特定类型或部分区域花生主栽品种的指纹图谱。翁跃进[3]利用两对AFLP标记构建了9个抗旱花生品种的指纹图谱。李双铃等[4]对10个山东花生主栽品种进行了AFLP分析, 研究结果表明, 至少2对标记可以将10个品种区分开。刘冠明等[5]用4对SSR引物构建了南方花生主产区20个品种的指纹图谱, 詹世雄等[6]用12对SSR引物构建了40个珍珠豆型花生品种的指纹图谱。本研究利用SSR标记构建河南省2015年之前选育并审定的全部花生品种指纹图谱, 将为新品种审定和评价提供技术支撑。
90个河南省选育和审定的花生品种由河南省农业科学院经济作物研究所保存、或收集自原育种单位, 其编号、名称、审定时间及育种单位见附表1。
将90个花生品种种植于育苗盘, 培养于人工培养箱, 取幼嫩叶片用CTAB法[7]提取基因组DNA。在2 mL离心管中加入2片幼嫩叶片和钢珠, 放在液氮中冷冻后使用球磨仪打磨30 s (30 Hz), 倒出钢珠加入600 µ L CTAB提取液, 65℃水浴30 min, 再加等体积氯仿在摇床上摇晃5 min, 12 000 × g离心后取上清液于1.5 mL离心管中, 加入冷乙醇沉淀DNA, 再用70%酒精纯化后晾干, 加适量ddH2O溶解备用。
按He等[8, 9]和Ferguson等[10]提供的序列, 根据已有的试验经验选取100对多态性好、带型清晰的SSR标记引物, 在上游引物5° 端加上19个碱基的通用引物(5° -CACGACGTTGTAAAACGAC-3° ), 以便和荧光标记引物M13引物(由ABI公司标记上4种荧光)配合使用, 使PCR产物能在ABI 3500XL遗传分析仪上分析。在Eppendorf 384 PCR仪上进行PCR扩增, 使用包括荧光引物的三引物10 µ L体系, 含10× PCR缓冲液1.0 µ L、10 mmol L-1各dNTPs 0.15 µ L、上游引物0.05 µ L、下游引物0.15 µ L、荧光引物M13 0.1 µ L、5 U Taq酶0.2 µ L、基因组DNA模板50 ng、ddH2O 8.5 µ L。其中荧光引物M13分为蓝色和绿色2种。PCR热循环参数为94℃预变性5 min; 94℃变性50 s, 58℃退火50 s, 72℃延伸1 min, 循环6次; 再以94℃变性50 s, 55℃退火50 s, 72℃延伸1 min, 循环25次; 最后72℃延伸10 min。PCR扩增后将产物用384针复制器蘸取并加入到每孔加有7 µ L甲酰胺(含有0.4% GeneScan 500LIZ橙色荧光内标)的384孔板中混匀, 短暂离心后在PCR仪上94℃变性5 min, 冷却后放在ABI 3500XL全自动测序仪上进行毛细管电泳分离检测, 使用Genographer v2.1软件分析电泳图。
先按基因型读取扩增出来的条带, 再转化成0、1格式, 在相同迁移率位置上, 有带记为1, 无带记为0, 并建立DNA指纹的数据库。数据经转化后, 用Powermarker v.3.25[11]计算引物的多态性信息量PIC值(Polymorphic Information Content), PIC值可代表引物的鉴定能力, PIC =1-Σ fi2, 其中fi为i位点的基因频率[12]。用NTSYS-pc 2.10软件[13]计算Dice遗传相似性系数[14], 用SAHN程序[15]和UPGMA方法[16]进行聚类分析。用Structure 2.2软件[17, 18]分析花生品种的遗传结构。
选取100对花生SSR标记引物在12个品种上进行多态性评价, 从中选取带型清晰、多态性好的60对引物对90个审定花生品种扩增, 共扩增出312条DNA条带, 扩增片段大小在99~426 bp之间, 每对引物扩增的SSR条带数在2~11之间, 平均5.2条, 其中多态性DNA条带267条, 多态性比率为85.6%, 部分引物的扩增带型见图1。SSR引物的PIC值变幅为0.11~0.77, ARS50的PIC值最高, 最低的是ARS1037, 平均值为0.503。
选取PIC值高, 扩增带型清晰的14对SSR引物(表1)用于构建90个审定花生品种的DNA指纹图谱。这14对引物扩增到101条DNA条带, 其中95条为多态性条带, 平均PIC值为0.60 (表2)。利用这95条多态性条带构建了90个河南省审定花生品种的指纹图谱(表3), 90个品种中没有指纹完全相同的品种, 表明所构建的DNA指纹图谱是有效的。90个花生品种中, 除漯花6号(7)和豫花9327 (18), 郑农花9号(10)和郑农花12 (72), 豫花11 (15)和濮科花4号(53) 3组品种间仅有一个差异位点, 其他品种间均有2个或2个以上差异位点。其中, 郑农花9号和郑农花12来自同一育种单位, 豫花9327是漯花6号的母本, 豫花11是濮科花4号的母本。因此, 这3个组合的品种间遗传相似性高, 差异位点相对较少。
 | 图1 花生SSR引物ARS202(上)和ARS1024 (下)的扩增带型Fig. 1 Amplification patterns of SSR markers ARS202 (up) and ARS1024 (down) |
| 表1 多态性SSR引物名称及其序列 Table 1 Polymorphic SSR primers |
| 表2 SSR引物扩增情况 Table 2 Amplification results of SSR primers |
| 表3 利用14个标记构建的审定花生品种DNA指纹图谱 Table 3 DNA finger prints of peanut varieties using 14 SSR markers |
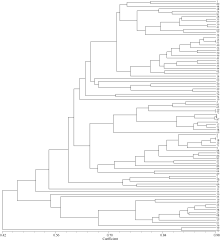
利用14对标记扩增得到的95条多态性条带用NTSYS-pc 2.10[13]进行聚类分析(图2), 结果表明, 在遗传相似系数为0.98处, 这些品种可被分为88类。豫花11 (15)和濮科花4号(53)及漯花6号(7)和豫花9327 (18)未被区分开, 是因为它们具有较高的遗传相似性, 豫花11是濮科花4号的母本, 豫花9327是漯花6号的母本, 且它们分别仅有一个差异位点(表3)。从图2还可以看出, 亲缘关系近即有共同祖先的品种优先聚集在一起, 不同育种单位育成的品种没有明显的聚集现象, 可能是因为各育种单位使用了较多的共同亲本。
90个育成品种间遗传相似系数的最小值、最大值和平均值见表4, 遗传相似系数最大值变幅为0.667~0.979, 最小值变幅为0.154~0.444, 平均值变幅为0.418~0.710。如果分别以遗传相似系数阈值T=0.80、0.85、0.90和0.95作为品种特异性标准[19, 20, 21], 那么具有特异性的品种比例分别为12.1%、25.6%、45.6%和74.4%, 即随着特异性标准的降低, 符合特异性标准的品种比例迅速增加。在本研究中, 若以T = 0.95为特异性品种划分标准, 有67个品种具有特异性, 22个品种不具备特异性。特异性较差的这些品种大多来源于同一育种单位, 且在图2中优先聚集在一起。
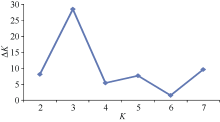
利用60对SSR引物得到的DNA条带, 用Structure 2.2软件分析了90个花生品种的群体结构。表明, 在K= 3时, Δ K值最大(图3), 90个花生品种可分为3个亚群(图4)。第1个亚群主要包括豫花9326、豫花9620、豫花21、商研9807、秋乐花177等33个品种, 主要是疏枝直立普通型大果品种; 第2个亚群包含45个品种, 可以分为3个类型, 疏枝直立普通型中小果品种如开农41、濮科花3号、濮科花4号、豫花9840等, 密枝直立普通型品种如豫花2号、豫花13、濮花16等, 疏枝直立中间型大果品种如豫花1号、豫花9331、豫花9634、远杂9614、濮科花5号等。第3个亚群包含远杂9102、远杂6号、豫花22、驻花1号、濮花17等12个品种, 为疏枝直立珍珠豆型品种。
| 表4 品种间的最大、最小及平均相似遗传系数 Table 4 Genetic similarity coefficient among varieties by pairwise comparison |
河南省是我国花生育种和种植大省, 近年来审定花生品种较多, 且多数直接作为亲本用于新品种培育, 在许多形态特征上表现相似, 单纯依靠表型很难将一些品种区分开, 因此构建花生品种分子指纹图谱具有重要意义。目前, 国内用DNA指纹图谱鉴定玉米品种的体系已经成熟, 并制定了农业行业标准《玉米品种鉴定— — DNA指纹方法》(NY/T1432- 2007), 用DNA指纹图谱鉴定其他物种的品种也将成为一种趋势, 本研究为花生DNA指纹图谱行业标准的制定奠定了基础。
本研究使用ABI 3500XL全自动测序仪进行毛细管电泳分离检测较常规的聚丙烯酰胺凝胶电泳更高效和准确。由于花生是四倍体物种, 常规的聚丙烯酰胺凝胶电泳跑出的带较多, 人工读胶不容易区分主带和副带, 且易受操作过程影响, 重复性较差, 在全自动测序仪上进行毛细管电泳分离后, 使用Genographer v2.1软件进行基因分型, 得到的带型清晰, 主带、副带易区分, 且操作不易受外界影响, 重复性好。
在遗传相似系数0.98处仅有一个差异位点的漯花6号(7)和豫花9327 (18), 豫花11 (15)和濮科花4号(53)分别聚集在一起; 而郑农花9号(10)和郑农花12 (72)虽然也只有一个差异位点, 但并未聚集在一起(图2)。因为根据Dice遗传相似系数[14]的计算方法C= 2a/(2a+b+c), 其中, a为2个品种共有的条带, b和c分别为2个品种独自出现的条带, 虽然3对品种中的b和c是一样的, 但a的不同会导致遗传相似系数不同, 进而影响聚类结果。
聚类分析使用了构建指纹图谱的14个SSR标记(图2), 群体结构分析使用了60个SSR标记(图4), 导致两种分群结果略有差异。图2中在相似系数0.62处可将90个审定品种划分为5类。第1类包含39个品种, 第3类包含5个品种, 第5类包含2个品种, 这3个类群可对应于图4中第2个亚群; 第2类包含29个品种, 对应于图4中第1个亚群; 第4类包含11个品种, 对应于图4中第3个亚群, 其余4个品种未被归类。
本研究发现河南省育成品种间的遗传相似系数最大值为0.979, 最小值为0.154, 平均为0.594, 在最大遗传相似系数阈值为0.95时, 有74.4%的品种具有特异性。目前, 国际上关于划分品种特异性的临界值尚无统一标准[20], 在玉米[19]、油菜[20]、芝麻[21]的研究中将0.90确定为品种特异性的划分标准, 本研究结果表明, 以遗传相似系数T=0.95作为河南省花生品种特异性的划分标准较为合适。一方面由于花生本身的遗传基础相对较窄[1], 另一方面育种单位较多地使用一些已审定的品种作亲本进行新品种的选育, 如濮科花4号以豫花11作母本, 漯花6号以豫花9327作母本等。因此, 总体来看, 河南省审定的花生品种特异性较差, 遗传背景狭窄。
根据《玉米品种鉴定— — DNA指纹方法》(NY/ T1432-2007)规定, 在品种间分析检测20对SSR核心引物, 差异位点数≥ 2时判定为不同品种, 差异位点数为1时判定为近似品种。本研究的90个花生品种间大多具有2个或2个以上差异位点, 有3对品种间仅有一个SSR差异位点, 因此, 应筛选出更多的标记来判定品种间的特异性。
利用14对SSR引物构建了河南省2015年前审定的90个花生品种的指纹图谱, 这些标记产生的95个多态性位点可以将90个花生品种完全区分开; 以遗传相似系数0.95为划分标准, 74.4%的品种具有特异性。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|