第一作者联系方式: E-mail: ninghailongneau@126.com, Tel: 0451-55191042
蛋白质和油分含量是大豆重要的育种目标, 蛋白质和油分含量QTL定位和优异等位变异的发掘对大豆分子设计育种具有重要意义。本研究以(垦丰14×垦丰15)×(黑农48×垦丰19)衍生的后代株系为材料, 构建含有204个株系的大豆四向重组自交系群体, 利用区间作图法, 应用前期构建的SSR遗传图谱, 对2013、2014和2015年在哈尔滨和克山2地共8个环境下的蛋白质和油分含量进行QTL定位分析。结果表明, 8个环境中检测到29个蛋白质含量QTL和39个油分含量QTL。在所定位的蛋白质含量QTL中, 有5个能够在2个以上环境被定位到, 这些蛋白质含量QTL分布在 A1、D2、J、N和O等6个连锁群上, 对表型效应的贡献率为7.65%~20.08%, 其中 qPC-A1-1、 qPC-D2-1、 qPC-J-1和 qPC-O-2的贡献率在10%以上。在39个油分含量QTL中, 有10个在多环境下被重复检测到, 这些QTL分布在8个(A1、A2、B1、D1b、G、I、J、N)连锁群上, 对表型效应的贡献率为7.30%~25.68%, 其中 qOC-A2-1、 qOC-B1-1、 qOC-G-1和 qOC-J-1的贡献率在10%以上。
Increasing protein content (PC) and oil content (OC) are main goals in soybean improvement, so mapping quantitative trait locus (QTL) and mining elite alleles underlying PC and OC are of importance for molecular design breeding in soybean. In this research a four-way recombinant inbred line population derived from double cross (Kenf 14 × Kenf 15) × (Hein 48 × Kenf 19) with 204 lines was used to analyze the data of PC and OC from the field experiments in eight environments across Harbin and Keshan in 2013, 2014, and 2015 by interval map method based on a linkage map constructed in previous research. The 29 PC QTLs and 39 OC QTLs were detected from eight planting environments. Among the twenty-nine PC QTLs, five were detected across over two environments, which distributed on six linkage groups, i.e. A1, D2, J, N and O, with explained phenotypic variation (PVE) ranging from 7.65% to 20.08%. Four of them, i.e. qPC-A1-1, qPC-D2-1, qPC-J-1, and qPC-O-2, showed PVE over 10%. Of the thirty-nine OC QTLs, ten were found in more than two environments, which were located on linkage groups A1, A2, B1, D1b, G, I, J, and N with PVE ranging from 7.30% to 25.68%. Four out of the ten QTLs that included qOC-A2-1, qOC-B1-1, qOC-G-1, and qOC-J-1had PVE above 10%.
大豆(Glycine maxL. Merr.)是重要的油料和经济作物, 为人类食用植物蛋白质和油脂的主要来源, 提高大豆籽粒中蛋白质含量和油分含量是大豆品质育种的重要研究方向。国内外关于大豆蛋白质含量和油分含量QTL定位的报道已有很多。例如, Mehrzad等[1]利用OAC Wallace× OAC Glencoe杂交F4:6RIL单株203个群体定位5个蛋白质含量和11个油分含量QTL, 分布在B2、D1a、D2、H、K、J、F、L连锁群上; Pandurangan等[2]用X3145-B-B- 3-15× AC Brant杂交得到F4:5 RIL单株201个群体, 共定位4个蛋白质含量QTL, 分别在I、E、C2、D1a连锁群上; 葛振宇等[3]用TK780× Hidaka 4杂交96个RILs定位2个蛋白质含量和3个油分含量QTL, 分布于E、H、I连锁群上; 林延慧等[4]用齐黄26× 滑皮豆杂交F2代分离群体170株, 在2个地点共定位到4个与蛋白质含量有关的QTL, 分布在D2、E、K连锁群上。截至目前, 在http://soybase.org/网站里共收集了已经定位到的152个蛋白质含量QTL, 188个油分含量QTL。国内外定位的品质性状QTL数量虽多, 但多是运用单一环境下两亲本衍生的群体定位[9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21]。由于大豆蛋白质和油分为数量遗传, 受多基因控制, 很容易受到环境的影响, 且2个亲本的杂交后代在进行连锁分析时1个位点涉及2个等位基因, 所以检测效率低。最近, 四向重组自交系群体(four-way recombinant inbred line, FW-RIL)被提出并应用于遗传分析, 与双亲本衍生的自交系群体相比多态性的标记数量增加, 遗传图谱的密度增加, 分子标记的多态性更为丰富, 同时还可以在1个基因位点分析4个复等位基因效应, 提高QTL检测功效[22]。在前期研究中, 我们创制了大豆四向重组自交系群体[23, 24, 25]并构建了遗传图谱[26]。
本研究应用前期构建的四向重组自交系群体及其遗传图谱, 进行3个年份8个环境的QTL定位分析, 以期探索多环境重复定位的QTL, 提高主效QTL的真实性, 并应用四向重组自交系群体多等位基因的优势, 探寻有利于蛋白质含量和油分含量改良的优异等位基因, 为大豆品质性状分子设计育种提供理论依据与技术支撑。
用蛋白质含量和油分含量差异较大的大豆亲本垦丰14 (蛋白质含量为39.69%, 油分含量为20.34%)、垦丰15 (蛋白质含量为38.68%, 油分含量为22.76%)、黑农48 (蛋白质含量为44.71%, 油分含量为19.05%)和垦丰19 (蛋白质含量为42.52%, 油分含量为19.26%) 4个亲本配制双交组合(垦丰14× 垦丰15)× (黑农48× 垦丰19), F1进行杂交, 采用单粒传方法获得RIL群体204个株系。
2013年在黑龙江省哈尔滨(E1)、克山(E2)进行田间试验。2014年在哈尔滨分2个播期(E3为5月10日、E4为5月30日)种植。2015年在黑龙江省哈尔滨分2个密度种植(E5为2.22× 105株 hm-2、E6为3.08× 105株 hm-2)、在克山分2个密度种植(E7为2.58× 105株 hm-2、E8为3.51× 105株 hm-2)。将4个亲本和FW-RIL在田间种植。采取随机区组试验设计, 小区行长5 m, 垄距65 cm, 株距6 cm, 3行区, 3次重复, 田间管理同一般大田栽培。
成熟后, 随机取四向重组自交系群体每个株行5株, 利用近红外谷物品质分析仪测定其蛋白质含量和油分含量, 5株的平均值作为该株系的品质数据。
参照2003年Gregan等[27]发表的大豆公共图谱, 初步挑选了638对SSR引物在4个亲本之间进行多态性筛选, 其中275对引物在4个亲本之间表现出良好的多态性。根据SoyBase网站提供的大豆SSR序列合成引物。利用275对SSR引物在4个亲本及FW-RIL群体的160个后代株系上进行PCR扩增。PCR扩增体系含3 µ L总DNA (50 ng µ L-1)、上下游引物(100 nmol µ L-1)各3 µ L、0.3 µ L dNTPs (10 mmol L-1)、2 µ L 10× 缓冲液、0.2 µ L Taq酶(5 U µ L-1)、用超纯水补足20 µ L。PCR扩增条件为94℃预变性10 min, 进入循环: 94℃变性30 s; 50℃复性30 s; 72℃延伸30 s; 循环38次后在72℃延伸5 min, 于4℃保存。每个PCR体系加上8 µ L上样缓冲液, 置PCR仪中变性10 min, 然后放入冰上冷却。PCR产物在6%的聚丙烯酰胺凝胶上, 1500 W恒功率电泳约1.5 h。用20 mL酒精(95%)+10 mL冰乙酸+3 mL AgNO3+1500 mL蒸馏水染色10 min, 清水漂洗30 s后放入30 g NaOH+6 mL甲醛+1500 mL蒸馏水显色5~10 min。
利用4个亲本配制双交组合(垦丰14× 垦丰15)× (黑农48× 垦丰19) F1进行杂交, 采用单粒传方法获得RIL群体160个株系。构建了一张含有275个SSR引物、包含20条连锁群的大豆遗传图谱, 该图谱覆盖基因组长度为3636.26 cM, 标记间平均长度为15.47 cM。每个连锁群长度范围为49.36~319.02 cM, 标记范围为6~20个[26]。
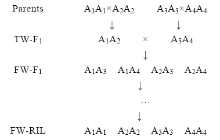
假定控制某一性状的QTL位点涉及4个等位基因, 4个亲本的基因型为A1A1、A2A2、A3A3和A4A4, 按照(A1A1× A2A2)× (A3A3× A4A4)方式(图1)进行交配设计, 可获得四向重组自交系群体, 在该群体中有4种可能的主基因A1A1、A2A2、A3A3和A4A4。
群体中第j个体的表型值向量为yj, 则具有如下统计模型:
yj = μ + x1ja1 + x2ja2+ x3ja3 + ej
式中, μ 为性状的均值, ai (i = 1、2、3)分别为等位基因型A1A1、A2A2和A3A3的加性效应, ej为剩余效应, 包括多个微基因效应和误差效应, 服从N (0, σ 2)。x1、x2、x3为主基因型的指示变量, 具有如下定义:
当等位基因型为A1A1时, x1= 1, x2= 0, x3= 0
当等位基因型为A2A2时, x1= 0, x2= 1, x3= 0
当等位基因型为A3A3时, x1= 0, x2= 0, x3= 1
当等位基因型为A4A4时。x1= -1, x2= -1, x3= -1
对于某一个体, 上述模型为具有x1、x2、x3缺失值的广义线性模型。依据第j个体主基因型, 具有如下条件概率分布:
yj| A1A1~ N (μ + a1, σ 2)
yj| A2A2~ N (μ + a2, σ 2)
yj| A3A3~ N (μ + a3, σ 2)
yj| A4A4~ N (μ -a1-a2-a3, σ 2)
模型中待估计的参数包括a1、a2、a3和σ 2, 可用EM算法实现的极大似然方法估计, 对各位点应用似然比方法测验是否存在QTL, LOD取值2.5。统计方法的详细过程在另文中论述。
应用以上方法分别对各环境的数据分析, 检测QTL, 本文仅列出能够在2个以上环境中重复检测到的QTL。
从3年8个环境下蛋白质含量和脂肪含量的描述性分析(表1、表2和图2)可看出, FW-RIL间和环境间的蛋白质含量和油分含量存在较大的变异。因此, 可用该群体进行QTL定位研究, 并且从不同环境定位结果可能发现不同的QTL。
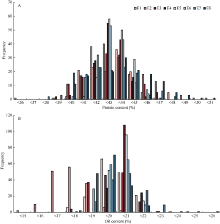 | 图2 不同环境蛋白质含量(A)和油分含量(B)的次数分布Fig. 2 Frequency distribution of protein content (A) and oil content (B) under different environments |
| 表1 不同环境FW-RIL蛋白质含量和油分含量的描述性分析 Table 1 Descriptive analysis of protein content and oil content via FW-RIL under different environments |
| 表2 8个环境间大豆蛋白质、油分含量的方差分析 Table 2 Analysis of variance for protein and oil contents in soybean among eight environments |
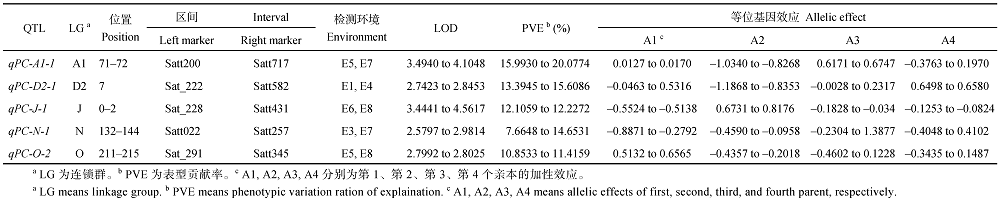
通过3年8个环境检测到的大豆蛋白质含量QTL 29个, 其中能够在2个以上环境被定位的有5个(表3)。蛋白质含量QTL主要定位在大豆20个连锁群中的14个连锁群上(表3)。由表3可知, 在E5、E7环境中, qPC-A1 (Satt200-Satt717)被定位于A1连锁群上, LOD值为3.4940~4.1048、PVE值为15.9930~20.0774、加性效应值为-0.8268~0.6171, 可提高蛋白质含量的优异等位基因来自第1、第3、第4亲本; 在E1、E4环境中, qPC-D2-1 (Sat_222- Satt582)被定位于D2连锁群, LOD值为2.7423~ 2.8453、PVE值为13.3945~15.6086、加性效应值为-1.1868~0.658, 优异等位基因来自第1、第2、第4亲本; 在E6、E8环境中, qPC-J-1 (Sat_228-Satt431)被定位于J连锁群, LOD值为3.4441~4.5617、PVE值为12.1059~12.2272、加性效应值为-0.5524~ 0.6731, 优异等位基因来自第2亲本; 在E3、E7环境中, qPC-N-1 (Satt022-Satt257)被定位于N连锁群, LOD值为2.5797~2.9814、PVE值为7.6648~14.6531、加性效应值为-0.8871~1.3877, 优异等位基因来自第1、第3、第4亲本; 在E5、E8环境中, qPC-O-1 (Sat_291-Satt345)被定位于O连锁群, LOD值为2.7992~2.8025、PVE值为10.8533~11.4159、加性效应值为-0.4602~0.6565, 优异等位基因来自第1、第3、第4亲本。
| 表3 8个环境中大豆蛋白质QTL定位结果 Table 3 QTL location of soybean protein content under eight environments |
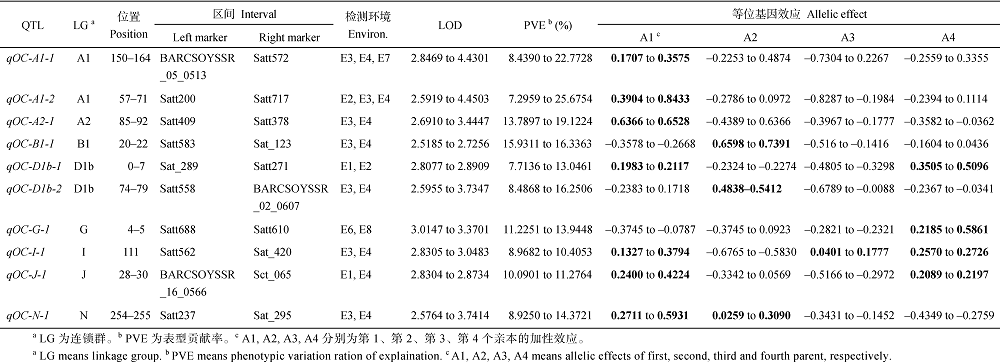
通过3年8个环境检测到大豆油分含量QTL39个, 其中能够在多个环境中重复检测到的有10个(表4)。油分QTL主要定位在大豆20个连锁群中的17个连锁群(表4)。由表4可知, 在E3、E4、E7环境中, qOC-A1-1(BARCSOYSSR_05_0513-Satt572)被定位于A1连锁群, LOD值为2.8469~4.4301、PVE值为8.4390~22.7728、加性效应值为-0.7304~0.4874, 可提高油分含量的优异等位基因来自第1、第2、第4亲本; 在E2、E3、E4环境中, qOC-A1-2(Satt200- Satt717)被定位于A1连锁群, LOD值为2.5813~ 4.4503、PVE值为7.2959~4.4503、加性效应值为4.4503~4.4503, 优异等位基因来自第1、第2亲本; 在E3、E4环境中, qOC-A2-1 (Satt409-Satt378)被定位于A2连锁群, LOD值为2.6910~3.4447、PVE值为13.7897~19.1224、加性效应值为-0.4389~0.6528, 优异等位基因来自第1、第2亲本; 在E3、E4环境中, qOC-B1-1 (Satt583-Sat_123)被定位于B1连锁群, LOD值为2.5185~2.7256、PVE值为15.9311~ 16.3363、加性效应值为-0.5160~0.7391, 优异等位基因来自第2、第4亲本; 在E1、E2环境中, qOC-D1b-1 (Sat_289-Satt271)被定位于D1b连锁群, LOD值为2.8077~2.8909、PVE值为7.7136~13.0461、加性效应值为-0.4805~0.5096, 优异等位基因来自第1、第4亲本; 在E3、E4环境中, qOC-D1b-2 (Satt558- BARCSOYSSR_02_0607)被定位于D1b连锁群, LOD值为2.5955~3.7347、PVE值为8.4868~16.2506、加性效应值为-0.6789~0.5412, 优异等位基因来自第1、第2亲本; 在E6、E8环境中, qOC-G-1 (Satt688- Satt610)被定位于G连锁群, LOD值为3.0147~ 3.3701、PVE值为11.2251~13.9448、加性效应值为-0.3745~0.5861, 优异等位基因来自第2、第4亲本; 在E3、E4环境中, qOC-I-1 (Satt562-Sat_420)被定位于I连锁群, LOD值为2.8305~3.0483、PVE值为8.9682~10.4053、加性效应值为-0.6765~0.3794, 优异等位基因来自第1、第3、第4亲本; 在E1、E4环境中, qOC-J-1 (BARCSOYSSR_16_0566-Sct_065)被定位于J连锁群, LOD值为2.8304~2.8734、PVE值为10.0901~11.2764、加性效应值为-0.5166~0.4224, 优异等位基因来自第1、第3、第4亲本; 在E3、E4环境中, qOC-N-1 (Satt237-Sat_295)被定位于N连锁群, LOD值为2.5764~3.7414、PVE值为8.9250~ 14.3721、加性效应值为-0.4349~0.5931, 优异等位基因来自第1、第2、第4亲本。
| 表4 8个环境中大豆油分含量QTL定位结果 Table 4 QTL location of soybean oil content under eight environments |
国内外关于大豆蛋白质含量、油分含量QTL的研究日益增多, 研究方法也有所不同, 以往研究多是单环境分析, 采用较高的LOD值, 导致相关的QTL未被检测到而丢失数据; 或采用较低的LOD值, 导致检测QTL时可能会有假阳性出现。而本文采用同一地点不同年份、同一年份不同地点、相同地点年份不同密度和相同地点年份密度不同播种期等试验方案涉及的8个环境进行试验, 虽然包含了年份、地点、密度、播种期的效应, 但是可看为广义的环境, 应用多环境的数据联合分析, 能增大QTL的检测强度, 准确估计QTL的位置和效应, 更有利于搜索稳定的QTL。
关于四向杂交群体的遗传图谱构建理论方法已有报道[28, 29], 这些方法适用于四向杂交设计的F1代(four-way F1, FW-F1)(图1), 但是本文所涉及的FW-RIL与FW-F1的遗传世代不同导致遗传结构也不同, 这导致重组率的估计方法不同, 其中, FW-F1是根据2个双亲F1的基因型结合后代个体的基因型估计重组率[28, 29], FW-RIL是根据4个亲本的基因型结合后代个体的基因型估计重组率。关于四向重组自交系群体遗传图谱构建的算法作者已在前文[26]做了初步的介绍。
在FW-RIL群体中, 对于某一个QTL, 可能因为4个亲本可能携带的等位基因型数量有1、2、3、4个, 除1个等位基因没有遗传效应差异外, 其他3种等位基因数目可能对应1:1、1:2:1、3:1和1:1:1:1等4种遗传模式。在本文的模型中虽然只是考虑了1:1:1:1的一种情况。在理论上, 可把1:1、1:2:1、3:1等三种模式看作1:1:1:1 模式的特例, 即当其中的2个等位基因效应相等时, 模型即等同于1:2:1的情况; 当有2个等位基因效应相等, 同时另外2个等位基因效应也相等时, 模型即等同于1:1的情况; 当其中的3个等位基因效应相等时, 模型即等同于3:1的情况。但是在构建实际的统计方法过程中, 需要对等位基因的效应设置约束条件。关于QTL定位的模型选择需要深入研究, 有待进一步完善。
大豆蛋白质含量和油分含量受到环境条件的影响, 为克服环境对QTL分析结果的影响, 检测到不同环境下稳定表达的QTL, 准确估计QTL的位置和效应, 常在多个环境进行试验, 例如Fasoula等[5]在1年3个环境定位油分含量QTL 48个; Lee等[6]1年3个环境定位蛋白质含量QTL 16个, 油分含量QTL 10个; Panthee等[7]在2年6个环境定位蛋白质含量QTL 1个, 油分含量QTL 3个; Rossi等[8]2年2个环境共定位4个蛋白质含量QTL, 4个油分含量QTL。本研究采用区间作图法(IM)对蛋白质含量和油分含量进行QTL定位, 其中有5个蛋白质含量的QTL在多环境下被重复检测到, 10个油分含量的QTL在多环境下被重复检测到, 可认为这些QTL可用于分子设计育种。
本研究通过IM作图法定位在8个环境中定位到5个在2个以上环境中重复表达的蛋白质含量QTL。由于不同亲本组合衍生的群体遗传背景不同, 在亲本间具有多态性的分子标记也不同, 导致构建的遗传图谱不同, 为进行不同群体间定位结果的互相比较和验证, 将本文与前人研究检测的QTL根据所连锁的标记整合到公共图谱[30], 进行目标性状基因的基因组位置的比较。在检测到的5个蛋白质含量QTL所在的基因区域中, 有4个与以往研究的QTL区域重合, 有1个区域为本研究首次定位到QTL。蛋白质含量QTL qPC-A1-1在公共图谱的基因组位置为51.95 cM (Satt717)~92.88 cM (Satt200), 所在的基因组区域与已经定位的4个蛋白质含量的QTL [18, 13, 31, 32]和1个色氨酸含量的QTL [33]重叠。qPC-J-1在公共图谱的基因组位置为23.91 cM (Sat_228)~78.57 cM (Satt431), 所在的基因组区域包含已经定位的1个蛋白质含量QTL [6]、1个组氨酸QTL和1个苯丙氨酸QTL [33]。qPC-N-1在公共图谱的基因组位置为92.55 cM (Satt257)~102.57 cM (Satt022), 与1个蛋白质含量的QTL (Chen等, 2007)重叠。qPC-O-1的基因组位置为51.9 cM (Sat_291)~ 59.43 cM (Satt345), 与1个蛋白质含量的QTL重叠[16]。qPC-D2-1在公共图谱的基因组位置为53.84 cM (Satt582)~76.69 cM (Sat_222)位曾报道过在, 所在区域检测到蛋白质含量相关QTL, 但在其临近两侧检测到了亮氨酸、甲硫氨酸+半胱氨酸的QTL[33]和蛋白质含量QTL [34], 说明该区域也是蛋白质含量QTL富集区域, 该QTL可能参与蛋白质的代谢, 可认为该QTL为新发现的控制蛋白质含量的位点, 为分子设计育种奠定基础。
在所定位的10个多环境稳定表达的油分含量QTL中, 有6个区域在以往研究中定位到了油分含量相关性状的QTL。qOC-A1-1在公共图谱的基因组位置为51.95 (Satt200)~92.88 cM (Satt717), 在此区域内已经检测到8个油分含量QTL [11, 31, 32, ]。qOC-D1b-1在公共图谱的基因组位置为131.91 (Sat_ 289)~137.05 cM (Satt271), 在该区域已经定位了一个油分含量QTL [35]。qOC-D1b-2在公共图谱的基因组位置为43.91 (Satt558)~55.82 cM (BARCSOYSSR_ 02_0607), 在该区域已经定位了1个硬脂酸含量QTL[11]和1个亚麻酸含量QTL[37]。qOC-I-1在公共图谱的基因组位置为22.84 cM (Satt562)~98.38cM (Sat_420), 在该区域已经定位了18个油分含量QTL [18, 20, 34, 35, 38, 39, 40, 41, 42, 43, 44]、2个亚麻酸QTL[45]和2个油酸QTL [11, 45]。qOC-J-1在公共图谱的基因组位置为32.09 cM (Sct_065)~44.62 (BARCSOYSSR_16_0566), 在该区域已经定位了1个油分含量QTL [45]、1个亚麻酸QTL和1个油酸QTL [37]。qOC-N-1在公共图谱的基因组位置为74.98 (Satt237)~95.00 cM (Sat_295), 在该区域已经定位了2个油分含量QTL [16, 35]、1个亚麻酸QTL、1个油酸QTL和1个硬脂酸QTL[45]。有4个区域没有油分含量相关性状的QTL定位的报道, 但是在邻近区域却发现了油分含量或脂肪酸组分的QTL。qOC-A1-1在公共图谱的基因组位置为14.65 (Satt572)~26.81 cM (BARCSOYSSR_05_0513), 所在的区域未有油分含量QTL的报道, 但在其临近的3.54~14.37 cM区域定位到一个亚麻酸含量QTL [37], 28.00~31.28 cM区域定位到3个油分含量QTL [31, 35, 46]。qOC-A2-1在公共图谱的基因组位置为145.57 (Satt409)~ 165.72 cM (Satt378), 所在的区域未有油分含量QTL的报道, 但在131.97~145.57 cM区域定位到一个亚麻酸含量QTL [37]。qOC-B1-4在公共图谱的基因组位置为84.19 (Satt583)~100.87 cM (Sat_123), 所在的区域未有油分含量QTL的报道, 但在80.3~82.3 cM区域定位到一个油分含量QTL[35]。qOC-G-1在公共图谱的基因组位置为10.92 (Satt610)~12.54 cM (Satt688), 所在的区域未有油分含量QTL的报道, 但在0.84~2.84 cM和20.88~22.88 cM区域各定位到一个亚麻酸含量QTL [34, 47]。说明这4个油分含量QTL所在该区域也是油分含量QTL富集区域, 这些QTL可能参与油脂的代谢, 可认为这4个QTL为新发现的控制油分含量的位点。
定位到5个多环境稳定表达的蛋白质含量QTL, 10个油分含量QTL。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|