第一作者联系方式: E-mail: 16828333@qq.com **同等贡献(Contributed equally to this work)
SBP基因家族是一类植物基因组特有的转录因子, 参与植物生长发育及多种生理生化过程。近来, 大量研究已在多种植物中鉴定出 SBP转录因子, 但关于玉米( Zea mays L.) SBP转录因子家族的系统分析报道尚少。本研究通过对拟南芥、水稻等植物已知的转录因子与玉米基因组数据比对, 并设置一系列严格的筛选标准从玉米基因组中挖掘 SBP转录因子, 系统发育分析显示单子叶植物玉米和水稻的 SBP基因保守性更强、亲缘关系更近; 共鉴定37个 SBP基因, 分布在9条染色体上; 通过基因分析注释以及启动子功能预测, 进一步发现 SBP家族基因参与植物生长发育、形态建成、逆境胁迫响应、花器官发育以及植物光反应等过程。并且, 玉米 SBP转录因子可通过参与赤霉素、生长素、脱落酸、水杨酸等多条激素信号调控途径来调节植物的生长发育。
SBP gene family, as a plant special transcription factor, is involved in plant growth and development, as well as many physiological and biochemical processes. Recently, SBP transcription factor family has been identified in model plants, such as Arabidopsis and Oryza sativa; however, systematic analysis of SBP transcription factor family in maize ( Zea mays L.) is scarcely. In this study, based on homology alignment technology, we aligned all known SBP TFs from Arabidopsis and Oryza sativa with those from maize genome sequence to mine novel SBP TFs in maize. A total of 37 SBP TFs distributed in eight chromosomes were identified. Phylogenetic analysis indicated that SBP transcription factor genes have stronger homology, especially between Zea maysand Oryza sativa. Moreover, promoters- ciselements analysis of those SBP TFs demonstrated that they might be involved in plant growth and development, morphogenesis, adversity response, the development of flower organs and photosynthesis. It is probable that SBP TFs regulate plant growth and development through the multiple hormone of signaling transduction pathway, such as gibberellin, auxin, abscisic acid, and salicylic acid.
目前, SBP转录因子已在大量植物中被鉴定, 具有多种功能, 在植物的生长发育过程中发挥重要调控作用。例如, 拟南芥SBP8和SBP14基因分别与调控花粉发育和真菌毒素FB1诱导的程序性死亡的抗性相关[4], 拟南芥SPL3基因的转录受发育调控, 主要集中在花序顶端分生组织、花分生组织和花器官原基等, 可能与控制开花时间有关[5]。水稻基因组SBP转录因子主要在花和愈伤组织表达[6]。番茄SBP基因(LeSPL-CNR)是控制果实发育的关键基因[7]。玉米SBP转录因子lg1与玉米舌叶和叶耳的发育密切相关, 若该基因发生突变, 植株将不能形成正常形态的舌叶和叶耳组织[8, 9]。Andrea等[10]发现玉米SBP转录因子tsh4与玉米雄花分枝形态构成有密切关系。Eveland等研究表明玉米SBP转录因子tga1调控玉米果穗的颖片发育, 是玉米进化过程中的一个关键基因, 同时Eveland等和Lä nnenpä ä 等[11, 12]研究揭示玉米SBP转录因子家族成员果实构造基因tga1控制有麸玉米的种皮形态特征。Chuck等[13]研究显示, 玉米SBP转录因子ub2和ub3基因可以通过控制玉米雌雄花序的分支和分化影响玉米的产量。研究进一步提示, ub2和ub3双突变玉米植株的雄花分支数减少, 花药数量减少, 散粉量严重减少, 同时玉米雌穗穗长变短、穗行数增加, 产量下降[13]。另外, 当tsh4基因也缺失时, 玉米产量下降更严重。Chuck等[14]研究显示, 玉米Cg1基因受Zma-miR156b/c的调控, 与玉米开花期SBP-box基因表达密切相关, 提示Cg1基因能够通过抑制玉米SBP-box基因表达, 进而调控玉米幼嫩叶片发育和雄花分枝。
目前, 大量研究陆续从在拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、葡萄(Vitis amurensis)、苹果(Malus pumilaMill)、大豆[Glycine max(Linn.) Merr]、草莓(Fragaria ananassa)、番茄(Lycopersicon esculentumMiller)等植物基因组中鉴定出SBP基因[3, 6, 15, 16, 17, 18, 19]。随着玉米基因组测序的完成为加快玉米的遗传育种和分子生物学研究提供了机遇, 然而, 关于玉米SBP基因的信息仍然较少。本研究利用生物信息学方法, 在玉米全基因水平鉴定SBP转录因子, 同时结合拟南芥、水稻SBP家族基因成员, 分析植物中SBP基因的进化关系及其表达模式, 研究结果不仅有助于鉴定玉米SBP基因家族的功能, 还可为以后玉米育种过程提供重要的理论依据。
水稻和拟南芥SBP蛋白序列来源于植物转录因子数据PlnTFDB网站(http://plntfdb.bio.uni-potsdam. de/v3.0/)。
从蛋白质家族数据库Pfam网站(http://pfam. sanger.ac.uk/)下载SBP基因家族的隐马氏模型序列谱(PF03110)文件, 应用HMMER 3.0程序包中的hmmbuild 命令将其转化成隐马氏模型序列模式。首先应用GENSCAN将在NCBI下载的玉米基因组B73 V5b+参考序列基因组(http://www.maizesequence. org/v5b+)数据预测得到蛋白序列, 并利用HMMER 3.0软件中的hmmsearch对这些蛋白进行转录因子预测(参数为默认设置)[20]。然后, 利用在线数据库SMART (http://smart.embl-heidelberg.de/)对候选SBP转录因子结构域进行鉴定, 确定真实的玉米SBP转录因子基因。利用玉米基因组mRNA数据库(http:// www.maizesequence.org/index.html)进行玉米SBP转录因子对应mRNA序列的比对分析(参数设置为BLASTp, 数据库基因集B73_RefGen_v3, E=1e-10, 其他参数默认); 收集所有阈值小于1.0的非冗余的数据集, 与PlnTFDB (http://plntfdb.bio.uni-potsdam. de/v3.0/)和PlantTFDB (http://planttfdb.cbi.pku.edu.cn) 中的SBP家族成员比对; 重新注释的序列通过其SMART (http://smart.embl-heidelberg.de/), CDD (http:// www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi)和Inter- ProScan (http://www.ebi.ac.uk/Tools/InterProScan/)来确认SBP家族结构域的存在, 进而查明玉米SBP转录因子mRNA序列。
利用ClustalX 2.1软件[21]对玉米SBP转录因子的SBP保守域蛋白序列进行多序列比对, 结合比对结果与水稻、拟南芥SBP转录因子的分类依据, 进行玉米SBP转录因子的分类。
首先, 从玉米、拟南芥和水稻的SBP转录因子中提取完整SBP结构域(包括I组成员N端和C端的SBP结构域, 发生缺失的SBP结构域被舍弃)的蛋白序列以及完整的SBP结构域。然后, 利用ClustalX 2.1软件进行蛋白质多序列比对。利用Mega 5.0软件Neighbor-Joining算法构建系统进化树[22, 23], 设置JTT模型, 位点替换速率Gamma分布, Bootstrap值为1000。
利用在线软件MEME 4.8.1 (http://meme.sdsc. edu/meme/cgi-bin/meme.cgi)对玉米SBP转录因子蛋白序列进行氨基酸保守域的预测分析。搜索得到的保守域数目上限为30; 保守域氨基酸残基的数量为10~300; 保守域可重复出现。
利用玉米转录组数据库qTeller (http://qteller. com/)中公布的RNAseq在各种组织中的表达结果分析玉米SBP转录因子(ZmSBP)表达模式, 并用MEV软件对所检测到的ZmSBP家族成员进行聚类分析。进一步利用PlantCARE (htp://bioinformatics.psb.ugent. be/webtols/plantcare/html/)网站预测候选ZmSBP可能包含的转录因子顺式作用元件, 预测ZmSBP功能。
利用Pfam工具搜索和在线数据库SMART、CDD和Inter-ProScan比对鉴定, 共获得37个典型结构域的玉米SBP基因, 分别命名为ZmSBP1~ ZmSBP37 (表1)。如图1所示, 除第9染色体外, 玉米SBP基因被锚定于所有染色体, 几乎呈均匀分布状态; 第4和第5染色体上SBP基因数目分布最多, 分别有8个和7个家族成员; 第3、第6、第7染色体上SBP基因数目最少, 为2个成员; 其余染色体上包含SBP基因3~5个。
| 表1 玉米SBP基因家族信息 Table 1 Information of SBP-domain gene family in maize |
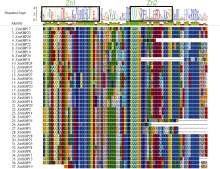
利用Clustal X程序对玉米SBP蛋白家族氨基酸序列进行多序列比对表明(图2), 除ZmSBP16、ZmSBP31、ZmSBP1外, 各玉米SBP蛋白的SBP结构域包含约79个氨基酸残基并具备2个锌指结构, 分别为C3 (C-C-C-H)和C2HC (C-C-H-C)类型, 命名为Zn1和Zn2; 其中, ZmSBP16、ZmSBP31和ZmSBP1中只含有Zn1结构而Zn2缺失结构; 另外, 除ZmSBP35、ZmSBP3、ZmSBP4外, 所有转录因子在SBP保守结构域的C端都有一个NLS (nuclear localization signal)核定信号位点, 其他ZmSBP均具有完全保守的SBP结构域。
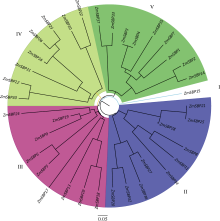
进一步依据SBP转录因子氨基酸序列的ClustalW多序列比对结果, 结合Meighbor-joining (J)和Bootstrap方法, 分析玉米ZmSBP转录因子蛋白的同源性和系统进化关系。结果显示(图3), 玉米SBP基因家族成员可以分为5种类型(I~V), 其中I类只有1个家族成员ZmSBP15, II和III类包含10个ZmSBP家族成员, IV和V类包含8个ZmSBP家族成员。
利用MEME软件分析结果, 构建ZmSBP转录因子蛋白保守序列的结构特征图, 即motif分布图。结果表明(图4), ZmSBP转录因子蛋白保守序列主要包括4个motif结构, 分别为motif 1~4, 其中所有氨基酸位点都包括C2HC结构域(motif 1)和C3H结构域(motif 2)。进一步分析显示, ZmSBP1和ZmSBP31在进化过程中缺失motif 1结构, ZmSBP19缺失motif 2结构, ZmSBP17及ZmSBP22在进化过程中motif 3结构发生了2~3次复制; ZmSBP12和ZmSBP30在进化过程中motif 4结构发生了2次复制, 推测这些ZmSBP蛋白在进化过程中, 通过对功能区序列的复制产生了新的功能。
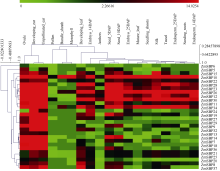
利用玉米转录组数据库qTeller中公布的RNA-seq数据, 对玉米不同组织SBP转录因子的表达模式及功能分析。图5表明, ZmSBP10、ZmSBP15、ZmSBP22、ZmSBP28、ZmSBP23、ZmSBP26、ZmSBP12和ZmSBP30在成熟玉米根茎、叶、果穗、胚及胚乳中大量表达。大部分ZmSBP基因在胚珠、幼穗和未受精果穗中大量表达, 但在花粉、维管束鞘组织和叶肉细胞中表达量很低。其中, ZmSBP10是唯一一个在维管束鞘和叶肉细胞中大量表达的转录因子, 推测ZmSBP10在进化过程中具有不同于其他玉米SBP家族成员的功能。
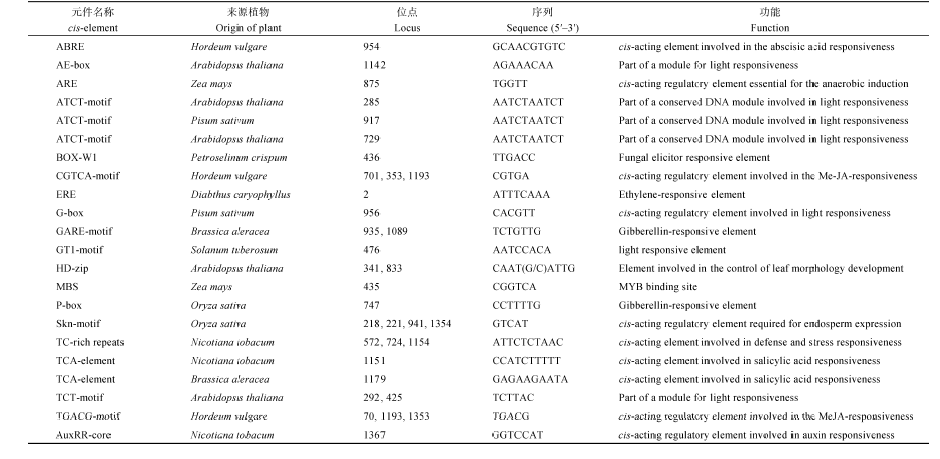
利用在线软件PlantCARE (http://bioinformatics. psb.ugent.be/webtools/plantcare/html/)预测ZmSBP转录因子启动子包含的顺式作用元件, 进而预测ZmSBP转录因子成员的功能显示(表2), 37个转录因子在启动子区的各种功能元件有所不同, 除了最基本的TATA-box、CAAT-box外, 个别转录因子还包括如W-box、MBS、AuxRR-core等其他转录因子的结合位点。其中, W-box是AP2/EREBP类转录因子结合位点, 涉及植物生长发育, 如糖酵解和脂肪酸循环途径中关键酶的合成。同时W-box也是真菌诱导反应元件, 能够参与植物生物胁迫响应过程; MBS是MYB转录因子家族特有的结合位点, 推测其可能参与诱导抗性基因表达, 进而参与调控植物逆境胁迫响应; AuxRR-core一类是生长素响应因子, 在植物形态建成, 如胚乳、子叶和侧根发育中起着重要作用。另外, 还发现大量光刺激应答元件, 推测其可能与植物光合作用相关, 参与调控植物开花等光反应。
| 表2 玉米SBP转录因子启动子区预测结果 Table 2 Forecasting result of promoter area of SBP transcription factors of maize |
玉米、水稻和拟南芥的SBP基因系统发育树, 表明(图6) 3个物种的基因家族成员可分为5个类群(I~V), 其中第II类群仅包括水稻和玉米SBP基因家族成员, 其余4个类群均包括来自3个物种的SBP家族成员。另外, 进化发育树末端同一分支的两个外部结点很可能就是亲缘关系比较近的同源基因对。进一步对进化树末端分析显示, 水稻和玉米中存在大量直系同源基因, 如LOC_Os05g33810.1和ZmSBP17、LOC_Os05g44860.1和ZmSBP13、LOC_ Os01g69830.1和ZmSBP37等, 但是在玉米和拟南芥、水稻和拟南芥中未能发现直系同源基因, 提示单子叶植物的SBP转录因子家族进化模式存在相似性, 推测双子叶植物(如拟南芥) SBP转录因子家族的基本特征形成时间早于单子叶植物。此外, 在物种内还发现了大量SBP基因旁系同源基因, 如玉米ZmSBP29和ZmSBP18、ZmSBP5和ZmSBP6、ZmSBP36和ZmSBP27、ZmSBP32和ZmSBP20、ZmSBP2和ZmSBP14等均为旁系同源基因, 提示SBP基因家族在物种内以大量同源基因形式存在, 这些基因家族在物种中按照各自物种特异性方式扩展。
玉米、水稻和拟南芥的SBP基因系统发育树, 表明(图6) 3个物种的基因家族成员可分为5个类群(I~V), 其中第II类群仅包括水稻和玉米SBP基因家族成员, 其余4个类群均包括来自3个物种的SBP家族成员。另外, 进化发育树末端同一分支的两个外部结点很可能就是亲缘关系比较近的同源基因对。进一步对进化树末端分析显示, 水稻和玉米中存在大量直系同源基因, 如LOC_Os05g33810.1和ZmSBP17、LOC_Os05g44860.1和ZmSBP13、LOC_ Os01g69830.1和ZmSBP37等, 但是在玉米和拟南芥、水稻和拟南芥中未能发现直系同源基因, 提示单子叶植物的SBP转录因子家族进化模式存在相似性, 推测双子叶植物(如拟南芥) SBP转录因子家族的基本特征形成时间早于单子叶植物。此外, 在物种内还发现了大量SBP基因旁系同源基因, 如玉米ZmSBP29和ZmSBP18、ZmSBP5和ZmSBP6、ZmSBP36和ZmSBP27、ZmSBP32和ZmSBP20、ZmSBP2和ZmSBP14等均为旁系同源基因, 提示SBP基因家族在物种内以大量同源基因形式存在, 这些基因家族在物种中按照各自物种特异性方式扩展。
 | 图6 玉米、拟南芥和水稻SBP转录因子基因的系统进化树Fig. 6 Phylogenetic trees constructed with SBP domain protein sequences from maize (ZmSBP), rice (OsSPL), and Arabidopsis (AtSPL) |
利用所有SBP基因的核心序列用于构建基因的系统进化树, 在图上标注每个SBP基因的序号、ID位点和数据来源。
SBP domain sequences of all genes were used for phylogenetic tree construction, the accession numbers or locus IDs and data sources of SBP-box genes are mached in the figure.
随着植物基因组学研究的深入尤其是测序技术的迅猛发展, 已陆续完成大量植物全基因组序列鉴定, 转录因子全基因组的鉴定及表达模型研究逐渐成为当前植物基因功能研究的热点。近年来, 不同植物种类的SBP基因家族成员及大小已得到分析, 这些研究结果提示植物SBP转录因子家族成员在调节植物生长发育及多种生理生化过程中发挥重要作用。同时, 玉米基因组测序的完成为我们在全基因
组水平分析SBP转录因子奠定了基础。本研究基于玉米全基因组测序数据库, 在玉米全基因水平鉴定SBP转录因子并分析其表达功能, 共鉴定出37个SBP基因, 包括1个I型基因, 10个II型基因, 10个III型基因, 8个IV型基因, 8个V型基因。通过与水稻、拟南芥转录因子系统发育关系比对分析, 在拟南芥、水稻中分别发现了25个和27个SBP转录因子, 并且水稻和玉米中大量SBP基因具有直系同源关系, 而在玉米和拟南芥、水稻和拟南芥中未能检出, 提示与双子叶植物如拟南芥比较, 单子叶植物玉米和水稻中SBP基因保守性更强、亲缘关系更近。利用系统发育树将3类物种SBP基因分为5个类群(I~V), 单子叶植物的SBP转录因子家族进化模式存在相似起源方式, 推测双子叶植物(如拟南芥) SBP转录因子家族的基本特征形成时间早于单子叶植物。
SBP转录因子保守结构域的C端是核定位信号区域, 能够引导蛋白进入细胞核行使功能[3]。本研究在分析SBP基序时发现, 玉米蛋白的结构域包含约79个氨基酸残基并具备2个锌指结构, 分别为C3 (C-C-C-H)和C2HC (C-C-H-C)类型, 推测其可能特异识别SQUAMOSA (SQ-UA)启动子。另外, ZmSBP转录因子蛋白保守序列分析结果显示, ZmSBP蛋白在进化过程中可能经历片段重复事件, 通过功能区序列复制而产生新的功能。
本研究进一步利用玉米转录组数据库所公布RNA-seq数据, 分析玉米SBP转录因子表达模式及功能, 结果显示, 大部分ZmSBP基因在玉米胚珠、幼穗和未受精果穗中大量表达, 但在玉米花粉、维管束鞘组织和叶肉细胞中表达量很低, 其中ZmSBP10是唯一一个在维管束鞘和叶肉细胞中大量表达的转录因子。研究表明, 植物花序发育与其产量密切相关, 如拟南芥同源基因SPL3、SPL4和SPL5能够调控拟南芥花发育, 拟南芥SBP8和SBP14基因能够调控其花粉发育[4, 5], 进而影响拟南芥产量。随着玉米SBP基因的揭示及功能研究迅猛发展, 大量研究显示玉米控制花序发育的部分SBP基因与玉米产量密切相关。例如, 玉米Cg1基因能够通过抑制玉米SBP基因表达, 调控玉米幼嫩叶片发育和雄花分枝[14]; 玉米ub2和ub3基因能够通过控制玉米雌雄花序的分支和分化[13], 调控玉米产量, 提示增强ub2和ub3基因表达可以增加玉米雄花分支和花药数, 进而增长玉米雌穗穗长, 提高玉米产量。部分研究揭示, 玉米tsh4能够调控玉米雄花分枝形态构成[10], 提示增强tsh4基因表达, 能够有效增加玉米产量。
Chen等[24]研究显示AtSPL8 (SPB转录因子)功能缺失会导致拟南芥花丝变短、萼片和表皮毛数量减少等半不育表型, 而外施赤霉素(GA)药剂, 其育性得到恢复, 这表明AtSPL8参与了GA信号转导途径, 推测SPB基因能通过参与调控赤霉素(GA)信号转导途径来影响植物的生长发育; Martin等[25]研究表明SBP转录因子可以通过参与信号传导途径调控种子萌发、根系生长、下胚轴及茎的伸长、植物发育阶段转变、成花诱导以及花器官发育过程。本研究进一步预测ZmSBP转录因子启动子顺式作用元件并分析ZmSBP转录因子成员的功能结果显示, SBP家族基因与花发育等植物生长发育、形态建成、逆境胁迫以及植物光反应等相关。并且, 玉米SBP转录因子可通过参与赤霉素、生长素、脱落酸、水杨酸等多条激素信号调控途径来调节植物的生长发育。
近几年研究表明, SBP转录因子不仅参与玉米根、茎、叶、果穗、胚、胚乳、胚珠等器官的生长发育, 而且涉及多条激素信号调控途径, 提示SBP转录因子在植物生长发育各进程具有重要的作用, 然而SBP转录因子上游调控机制尚不清楚。本文基于玉米全基因组测序的完成, 鉴定分析玉米SBP的基因结构、进化关系以及表达模型与功能, 为未来深入解析玉米SBP转录因子在玉米生长发育和调节激素信号传递途径中作用提供基因资源和理论依据。
共鉴定分布在9条染色体上的37个SBP基因, 单子叶植物玉米和水稻的SBP基因保守性更强、亲缘关系更近; 玉米SBP家族基因参与植物生长发育、形态建成、逆境胁迫响应、花器官发育以及植物光反应等过程, 可通过参与多条激素信号调控途径来调节植物的生长发育。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|