**第一作者联系方式: 刘峰, E-mail: 760733016@qq.com; 苏炜华, E-mail: 410946470@qq.com
Na+/H+逆向转运蛋白基因 SOS1(salt overly sensitive 1)是植物耐盐性的必需基因之一, 在植物抵御盐胁迫过程中发挥十分重要的作用。本研究以小麦EST序列KJ563230为探针, 利用电子克隆技术结合RT-PCR,获得一条甘蔗 SOS1基因的cDNA序列, 命名为 ScSOS1 (GenBank登录号为KT003285)。序列分析结果表明, 该基因全长1403 bp, 包含一个1272 bp的开放阅读框, 编码423个氨基酸的蛋白质。ScSOS1蛋白的相对分子质量为47.6 kD, 理论等电点(pI)为9.12。氨基酸序列分析表明, ScSOS1蛋白具有一个CAP-ED superfamily结构域。生物信息学预测显示, ScSOS1的编码蛋白为亲水性蛋白, 不存在信号肽, 二级结构元件多为无规则卷曲, 主要参与中间代谢。实时荧光定量PCR分析表明, ScSOS1基因的表达具有组织特异性, 在甘蔗叶鞘、蔗皮、蔗髓、侧芽和根中均有表达, 其中在叶鞘中的表达量最高, 根中的表达量最低。此外在NaCl、PEG、ABA、SA和MeJA的胁迫过程中, 该基因表达均受到调控, 其中受NaCl和PEG诱导后上调表达, 均在24 h表达量达最高, 分别约为对照组的1.5倍和4.0倍。推测该基因的表达与甘蔗耐盐性和抗渗透胁迫有关。
Salt overly sensitive 1 ( SOS1) gene, encoding a Na+/H+ antiport protein, plays an important role in biological processes of plants against salt stress. Using a SOS1mRNA sequence from Triticum aestivum (KJ563230) as the probe, the homologous ESTs of sugarcane were obtained from NCBI database. A sugarcane cDNA sequence of SOS1 gene was cloned by in silico cloning combined with RT-PCR, and named as ScSOS1 (GenBank accession number: KT003285). The bioinformatics analysis showed that ScSOS1has a length of 1403 bp with a complete open reading frame (ORF, 107 to 1423 bp), encoding a 423 amino acid residues of sugarcane SOS1 protein with an estimated molecular weight of 47.6 kD and a calculated isoelectric point (pI) of 9.12. The protein of ScSOS1 belongs to a conserved CAP-ED superfamily. Yet the ScSOS1 protein has no signal peptide and belongs to hydrophilic protein with the main function for intermediary metabolism. The mainly secondary structure element of ScSOS1 protein is random coil. Real-time quantitative PCR (RT-qPCR) analysis revealed that ScSOS1 was tissue-specifically expressed in leaf sheath, bark, pulp, bud and root of sugarcane, with the highest expression in leaf sheath and the lowest in root. Besides, the expression of ScSOS1gene could be regulated by the treatments of NaCl, PEG, ABA, SA, and MeJA, and up-regulated by the stresses of NaCl and PEG, with the highest inducible expression levels of 1.5 times and 4.0 times as high as those of control at 24 hours, respectively. This paper suggested that ScSOS1 involves in sugarcane tolerance salt and osmotic stresses. It can set up a basis for the elucidation of sugarcane salt resistance mechanism.
土壤盐渍化是一个世界性难题, 我国约有3.3× 107公顷的盐渍化土壤, 严重影响我国农业的生产效率和可持续发展[1]。土壤盐分过多会对植物生长发育造成危害, 如引起离子平衡和渗透平衡的失调, 影响酶的活性, 甚至会抑制光合作用等一些重要的代谢途径[2, 3, 4, 5]。前人研究表明, 在受到盐胁迫时, 植物细胞内活性氧的增加会加快叶绿体的氧化降解速度, 进而抑制叶绿体片层的合成, 降低叶片中的叶绿素含量, 最终降低光合作用[6, 7, 8, 9, 10, 11, 12]。同时, 在盐胁迫下, 植物细胞内还会产生大量氧自由基, 降低超氧化物歧化酶(SOD)的活性, 改变细胞膜通透性, 从而损伤细胞结构和细胞功能[13, 14, 15]。
植物抗盐或耐盐的生理基础主要包括渗透调节、离子区域化、光合作用途径改变、抗氧化防御系统的活性增加和植物激素调节[16]。有机小分子物质和无机离子如脯氨酸、甜菜碱、甘露醇、Na+、K+等可以调节植物的渗透压平衡, 还可以通过离子区域化将盐离子储存在细胞器或组织中, 减轻盐离子对植物的损害[17]。另一方面, 在盐胁迫下, 植物主要通过增加水的利用率和改变光合碳同化途径增强耐盐性, 同时体内的SOD、过氧化物酶(POD)、过氧化氢酶(CAT)等酶的活性升高, 以有效清除过多的活性氧, 防止膜脂过氧化[16]。当植物处于盐胁迫环境时, 植物内源信号激素脱落酸(ABA)被诱导合成, 通过改变植物体内水分和离子平衡, 对盐胁迫作出适应性反应, 最终提高植物的耐盐性[18]。陈义强等[19]研究发现, 在200 mmol L-1NaCl处理下, 斑茅体内的超氧化物歧化酶和过氧化物酶(POD)活性升高, 表现出较强的抗盐性。
迄今, 离子区域化酶类基因, 尤其是Na+/H+逆向转运蛋白基因SOS(salt overly sensitive)的克隆和功能分析, 是植物耐盐分子机制研究的热点。Gaxiola等[20]从拟南芥克隆到一个液泡膜Na+/H+逆向转运蛋白基因AtNHX1 (Na+/H+exchanger, NHX), 发现该基因与拟南芥H+-PPase基因AVP1(Arabidopis thaliana vacuolar-type H+-pumping pyrophosphatase 1)共同执行Na+的液泡转运功能。Waditee等[21]的研究发现, 藻青菌的质膜Na+/H+逆向转运蛋白基因ApNhaP(a halotolerant cyanobacterium)编码蛋白的主要作用是驱使Na+的外排, 这也是光合生物耐盐性的关键因素。前人研究揭示, SOS信号通路与植物耐盐性密切相关, 目前, 研究者[22, 23, 24, 25, 26]已从拟南芥中获得6个耐盐基因SOS1、SOS2、SOS3、SOS4、SOS5和SOS6, 其中SOS1在SOS信号通路中扮演十分重要的角色[27, 28, 29]。拟南芥SOS1基因编码一个质膜Na+/H+逆向转运蛋白, SOS1的C-末端为一长亲水性尾链, 位于胞质中, 作为感受器感知胞质中Na+浓度的变化, 进而引发胞质Ca2+信号的产生, 随后Ca2+信号刺激SOS3-SOS2复合物激活SOS1, 最后将Na+排出胞外, 提高植物的耐盐性[27, 28, 29]。拟南芥SOS1蛋白是质膜Na+/H+的逆向运输体, 同时也是SOS信号通路中最初的调节对象[30]。拟南芥SOS1突变体对盐非常敏感, 表明SOS1基因在拟南芥耐盐性中发挥重要的作用。权庚等[31]研究发现, 过量表达互花米草SaSOS1的转基因水稻对盐胁迫具有较强的适应能力, 说明该基因可以增强植株的耐盐性。此外, SOS1基因也已经在包括水稻[32]、小麦[33]、大叶补血草[34]等植物上被克隆和鉴定。但是, 国内外尚未见甘蔗SOS1基因的克隆和表达分析的报道。
我国甘蔗主要种植在盐碱地和旱地[35], 研究表明, 盐胁迫对甘蔗的发芽率和植株生长具有重要的影响, 是造成甘蔗产量和糖分下降的主要原因之一[36, 37]。因此, 了解甘蔗耐盐机理和挖掘甘蔗耐盐基因对培育耐盐甘蔗品种具有非常重要的意义。本研究以甘蔗品种崖城05-179 (YC05-179)为材料, 利用电子克隆结合RT-PCR及RT-qPCR技术, 分析甘蔗中ScSOS1基因的表达情况, 以期进一步揭示该基因的功能和作用模式。
甘蔗品种YC05-179由福建农林大学农业部福建甘蔗生物学与遗传育种重点实验室提供。主要试剂为PrimeScript TM RT-PCR Kit反转录试剂盒(TaKaRa, 中国大连、TRIzol Reagent (Invitrogen, Carlsbad, CA, USA)、Gel Extracti on Kit (Tiangen Biotech Co., 中国北京)、SYBR Green PCR Master Mix Kit (Roche, USA)。
从田间挑选健康无病长势相近的YC05-179蔗株, 将其切成单芽茎段, 并种植于经过高温高压灭菌的营养土中催芽(16 h/8 h, 光/暗, 28℃), 直至蔗苗长出4~6叶。其次, 选取长势一致的蔗苗, 用于组培苗的诱导、壮苗、生根。最后, 将组培甘蔗幼苗移出并在温室内开放水培一周, 再挑选长势均一的组培苗分为对照和4个试验组, 设置3次生物学重复。试验组处理[35]为5 mmol L-1水杨酸(SA: 6、12和24 h)、100 μ mol L-1茉莉酸甲酯(MeJA: 6、12和24 h)、100 μ mol L-1脱落酸(ABA: 6、12和24 h)、25.0% PEG (模拟干旱: 6、12和24 h)和250 mmol L-1 NaCl (高盐: 12、24和48 h) 水溶液培养, 以处理0 h的蔗苗为对照。取以上所有植物材料的样本立即投入液氮速冻并保存于-80℃冰箱至RNA提取。
用TRIzol Reagent试剂提取样品RNA, 包括组织特异性材料YC05-179的叶片、叶鞘、侧芽、蔗皮、蔗髓、根及经SA、MeJA、ABA、PEG和NaCl胁迫处理的蔗苗, 根据Prime-Script RT Reagent Kit操作说明书, 将RNA反转录合成cDNA, 用1.0%琼脂糖凝胶电泳检测质量。
采用电子克隆方法, 以小麦SOS1基因(KJ563230)的EST序列作为探针, 应用NCBI的BlastN工具检索甘蔗EST数据库, 将获得的甘蔗EST序列群(表1), 同源性达到84%以上者, 用CAP3 sequence Assembly Program (CAP3) (http://pbil.univ- lyon1.fr/cap3.php)软件拼接组装获得cDNA序列。最后利用ORF finder (http://www.ncbi.nlm.nih.gov/ projects/gorf/)在线软件, 对所获得的cDNA序列进行ORF预测分析。
以上述电子克隆获得的cDNA序列为模板, 设计一对特异性的RT-PCR扩增引物SOS1-1F和SOS1-1R (表2)用于验证电子克隆序列的正确性。采用TRIzol法, 以NaCl处理过的YC05-179蔗苗为材料, 提取总RNA并反转录为cDNA作为模板。PCR扩增体系总体积25 μ L, 含10× Ex Taq buffer 2.5 μ L、10 mmol L-1 dNTPs 2 μ L、20 μ mol L-1上下游引物各1.0 μ L、Ex Taq酶0.125 μ L、cDNA模板1.0 μ L、ddH2O 17.375 μ L。PCR反应程序为: 95℃预变性4 min, 95℃变性30 s, 48℃退火30 s, 72℃延伸2 min, 35个循环; 72℃延伸10 min。将PCR产物切胶回收后连接到pMD19-T载体上, 随后转化到大肠杆菌DH5α 感受态细胞中。将PCR扩增验证筛选获得的阳性单菌落的菌液送往上海生工生物工程技术服务有限公司测序。
生物信息学分析方法[38]如下。利用ORF finder在线软件, 对甘蔗SOS1基因进行开放阅读框分析并翻译; 利用ExPASy中ProtParam pI/Mw (http://web. expasy.org/compute_pi/)在线工具对甘蔗SOS1基因所编码蛋白一级结构进行预测; 利用GOR IV (http:// npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)进行甘蔗SOS1二级结构预测; 利用SWISSMODEL (http://swissmodel.expasy.org/)进行甘蔗SOS1三级结构预测; 利用SignalP 4.1 Server (http://www.cbs.dtu.dk/services/SignalP/)进行甘蔗SOS1蛋白信号肽分析; 利用ProtScale (http://web. expasy.org/protscale/)进行甘蔗SOS1疏水性/亲水性预测; 利用Profun 2.2 Server (http://www.cbs.dtu. dk/services/ProtFun/)对甘蔗SOS1进行功能预测以及利用Psort (http://www.psort.org/)在线工具预测其亚细胞定位。同时, 用NCBI上Blast工具进行同源氨基酸序列查找, 并利用DNAMAN对所获得同源氨基酸序列以及甘蔗SOS1蛋白序列进行序列比对, 最后在MEGA6.0中采用NJ (Neighbor-Joining)法(BootStrap 1000)构建进化树。
根据甘蔗ScSOS1基因序列设计定量PCR引物SOS1-2F和SOS1-2R (表1)。本试验采用2个内参基因CUL和CAC, 内参基因的定量引物见(表2)[39]。采用RT-qPCR技术检测ScSOS1基因在甘蔗YC05-179上的相对表达量。PCR反应体系(20 μ L)含: SYBRGreen Primix Ex Taq (2× ) 10 μ L、上/下游引物(10 μ mol L-1)各0.8 μ L、cDNA 2.0 μ L、ddH2O 6.4 μ L。根据ABI7500实时荧光定量PCR仪默认程序设定实时定量PCR扩增程序为50℃ 2 min; 95℃ 10 min; 95℃ 15 s; 60℃ 1 min; 45个循环; 反应时设置3次技术重复, 以无菌水作对照[40]。荧光定量PCR反应结束后, 在ABI PRISM7500 Real-time PCR System软件上对定量数据进行初步分析, 导出数据文件, 采用2-Δ Δ Ct [41]算法分析试验结果。
以小麦SOS1基因(GenBank登录号为KJ563230)的EST序列为探针, 借助电子克隆技术结合RT-PCR扩增验证, 从甘蔗中分离获得一条SOS1基因的cDNA序列。利用特异性引物进行RT-PCR扩增, 产物经2%琼脂糖凝聚电泳得到单一条带如图1。经测序验证, 电子克隆所得核酸序列与测序序列(图2)的同源性达98.34%, 表明电子克隆序列是正确的, 最终得到的序列是使用2个克隆的测序结果校对所得的, 其中部分碱基的差异可能是PCR扩增中的错配所致。该基因被命名为ScSOS1, GenBank登录号为KT003285。其cDNA序列片段长1403 bp, 包含一个1272 bp的开放阅读框, 编码一个423个氨基酸的蛋白。
| 表1 本研究电子克隆中用到的甘蔗EST序列 Table 1 ESTs of sugarcane used in silicon cloning of this study |
| 表2 ScSOS1基因克隆与表达所用引物 Table 2 Primers used in ScSOS1 gene cloning and expression analysis |
2.2.1 甘蔗SOS1基因编码氨基酸的一级结构预测
通过在线工具ExPASy中ProtParam pI/Mw, 预测甘蔗ScSOS1基因编码蛋白质的一级结构显示, 该蛋白分子式为C2078H3357N621O622S19, 分子量Mw为47.6 kD, 包含423个氨基酸残基。SOS1蛋白等电点(pI)为9.12, 有负电荷残基(Asp+Glu) 47个, 正电荷残基(Arg+Lys) 54个, 蛋白质三维结构不稳定系数(II)为60.01, 平均疏水性(GRAVY)-0.396, 脂肪系数(AI) 86.69。蛋白质不稳定系数大于40, 蛋白质表现为不稳定状态[42], 因此推测甘蔗ScSOS1基因编码的蛋白质为不稳定的碱性蛋白质。
2.2.2 甘蔗ScSOS1蛋白二级结构预测和分析
根据GOR IV软件预测, 甘蔗SOS1蛋白无规则卷曲所占的比例高达51.30%, 其次是α -螺旋占32.86%, 延伸链所占比例是15.84%, β -螺旋的比例为0 (表3)。
| 表3 甘蔗ScSOS1蛋白二级结构预测分析 Table 3 Secondary structure prediction of sugarcane ScSOS1 protein |
2.2.3 甘蔗ScSOS1蛋白质三级结构预测
根据SWISSMODEL软件预测, ScSOS1空间结构以无规则卷曲和螺旋为主。比较甘蔗、玉米、小麦和水稻的ScSOS1蛋白的三维结构虚拟图, 结果如图3显示, 甘蔗ScSOS1蛋白的三级结构与高粱、水稻、小麦和玉米SOS1蛋白的三级结构都存在一定的差异。
2.2.4 甘蔗ScSOS1蛋白信号肽预测和分析
根据SignalP 4.1 Server软件预测可知, 第40位赖氨酸残基具有最高的原始剪切位点分值0.130和最高的信号肽分值0.125, 第39位丙氨酸残基具有最高的综合剪切位点分值0.267。由于最后获得氨基酸残基的加权平均值较小, 为0.0.118 (远远小于0.5), 推测ScSOS1基因所编码的蛋白不存在信号肽, 即为非分泌型蛋白, 说明该蛋白在细胞质中合成后不能被转运(表4)。
2.2.5 甘蔗ScSOS1蛋白疏水性/亲水性的预测和分析
蛋白质的疏水性可根据蛋白的平均疏水性(GRAVY)值来预测, 当GRAVY大于零时, 为疏水蛋白; 当GRAVY小于零时, 为亲水蛋白[43]。根据ProtScale软件预测, 第77位具有最高分值, 为2.589, 疏水性最强; 第332位具有最低分值, 为-3.644, 亲水性最强。平均疏水性(GRAVY) -0.396, 故推测甘蔗S0S1蛋白是一种亲水蛋白。
 | 图3 甘蔗、高粱、水稻、小麦和玉米SOS1蛋白三级结构预测Fig. 3 Predicted third structure of SOS1 protein in sugarcane, sorghum, nice, wheat, and maize |
| 表4 甘蔗ScSOS1蛋白信号肽预测 Table 4 Signal P-NN prediction for sugarcane ScSOS1 protein |
2.2.6 甘蔗ScSOS1蛋白的功能预测
根据Profun 2.2 Server软件预测, 该蛋白主要参与中间代谢, 可能性为3.189, 其次也可能参与翻译、氨基酸生物合成及脂肪酸代谢, 可能性依次为2.957、2.656和1.729。
2.2.7 甘蔗ScSOS1亚细胞定位预测
根据Psort软件预测, 甘蔗ScSOS1蛋白可能被定位于细胞核(60.0%), 其次是微体(过氧化物酶体) (30.0%), 再次是线粒体基质的空间(10.0%)、溶酶体(10.0%)。
2.2.8 甘蔗ScSOS1蛋白的保守结构域预测
如图4所示, 甘蔗ScSOS1蛋白包含CAP-ED (catabolite gene activator protein-effector domain) superfamily和一个Crp domain (cAMP-receptor proteins), 含有一个ligand结合位点和一个cNMP (cyclic nucleotide-monophosphate)结合位点。
2.2.9 甘蔗ScSOS1蛋白的氨基酸同源性分析
选择高粱(Sorghum bicolor, gb|XP_002443674.1|)、海滨盐草(Distichlis spicata, gb|ACZ05629.1|)、海枣(Phoenix dactylifera, gb|XP_008798100.1|)、黑麦草(Lolium perenne, gb| AAY42598.1|)、芦苇 (Phragmites australis, gb|BAF41924.1|)、水稻(Oryza sativa IndicaGroup, gb|EEC69753.1|)、小麦(Triticum aestivum, gb|AEI28609.1|)、小米(Setaria italica, gb|XP_004963354.1|)、玉米(Zea mays, gb|XP_008645743.1|)、中华大叶竹(Indosasa sinica, gb|AGB06353.1|)进行氨基酸同源性分析, 与甘蔗ScSOS1蛋白的氨基酸序列相似性分别为87%、58%、53%、63%、72%、68%、64%、71%、81%和69%。从图5可以看出, 甘蔗ScSOS1蛋白与同属单子叶植物的高粱的氨基酸相似性最高, 而与海滨盐草和海枣的的相似性较低。
 | 图5 甘蔗ScSOS1蛋白与其他植物种蛋白的氨基酸序列比对Fig. 5 Homology analysis of sequences from sugercane ScSOS1 and those of other species |
系统进化树(图6)显示, 同属禾本科植物的甘蔗和高粱同源性最高。甘蔗、高粱、玉米、小米、黑麦草、小麦、水稻、中华大节竹、海滨盐草和芦苇为一个分支, 海枣属于棕榈科, 独立地成为一个分支, 但是甘蔗与高粱的亲缘关系最近。
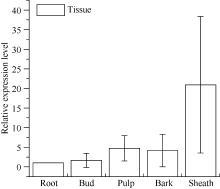
组织特异性表达的RT-qPCR分析显示(图7), 该基因的表达具有组织特异性, 在甘蔗品种YC05-179的叶鞘、蔗皮、蔗髓、侧芽、根等组织中均有表达, 但是在根中的表达量最少, 叶鞘中的表达量最高, 约是根的23倍; 其次是在蔗髓中的表达量, 约为根的5倍。
RT-qPCR表达分析结果表明, 甘蔗ScSOS1基因在不同外源胁迫下具有不同的表达特性(图8)。从图中可以看出, 在NaCl处理下, ScSOS1基因在甘蔗品种YC05-179中的表达量上调, 在24 h达到最高表达量, 约为0 h的1.5倍。此外, 该基因在48 h后的表达量逐渐下降但还是高于0 h的表达量。在PEG处理的3个时间点(6、12和24 h), ScSOS1基因的表达呈现上调的趋势, 在24 h达到最高, 约为0 h的4.0倍。在SA和ABA处理下, 该基因的表达出现上调和恢复两个阶段, 在6 h和12 h时呈现上调表达, 在12 h后表达量均达到最高, 分别是0 h的2.0和1.5倍, 且都在处理24 h后恢复到与0 h相当的表达水平。值得注意的是, 与SA和ABA胁迫下的表达模式相反, 在MeJA的处理下, ScSOS1基因的表达量先减少后增加, 在6 h的表达量最低, 约为0 h的0.5倍; 12 h后该基因的表达量逐渐上升并在24 h到达最高, 约为0 h的1.5倍。
植物耐盐性研究主要集中在盐胁迫的信号传导机制[44]。SOS信号途径是植物盐胁迫信号传导的重要途径之一[25]。其中, SOS1是SOS信号途径的一个重要组成部分, 与植物耐盐性密切相关[25]。权庚等[31]的水稻转基因实验证明, 过量表达SaSOS1能够增强水稻植株的耐盐性。因此, 克隆并研究甘蔗SOS1基因的特征特性及其在甘蔗耐盐机制中的作用机理具有较为重要的理论和实践意义。
本研究以小麦SOS1基因的EST序列为探针, 采用电子克隆和RT-PCR扩增验证相结合的方法, 成功克隆到甘蔗ScSOS1基因。该基因具有完整的开放阅读框。同源比对结果显示, ScSOS1蛋白与高粱SbSOS1蛋白的同源性最高, 达87.0%, 说明SOS1基因的编码蛋白在不同物种间具有较高的保守性。生物信息学分析显示, 甘蔗ScSOS1蛋白是一种亲水性的不稳定蛋白, 这与Zhao [45]对玉米ZmSOS1基因的生物信息学分析的结果相同。Tang等[46]研究发现虽然胡杨SOS1蛋白主要定位于细胞膜, 也有少量定位在细胞质中。但是, 本研究分析结果显示, 甘蔗ScSOS1蛋白可能定位于细胞核。此外, 该蛋白的二级结构主要为无规则卷曲和α -螺旋, 这与徐立新等[47]等获得的木榄质膜型Na+/H+逆向转运蛋白的二级结构特征相符。从三级结构预测结果可以看出, 甘蔗ScSOS1与其他物种该蛋白的三级结构都存在差异, 与高粱的差异较小, 而与水稻、小麦、玉米的差异较大, 其原因可能是甘蔗与高粱的亲缘关系较近而与其他物种的较远。保守结构域分析结果显示, ScSOS1蛋白含有一个CAP-ED superfamily, 该结构域包含一个分解代谢物激活蛋白并且该蛋白主要位于离子通道中, 推测ScSOS1参与中间代谢和离子转运。cAMP为细胞内的第二信使, 当细胞外信号与相应受体结合后, 导致细胞内cAMP (cyclic adenosine monophosphate)的水平变化而引起细胞发生反应。本研究中, ScSOS1蛋白还含有一个Crp结构域和一个cAMP受体结构域, 推测该蛋白可能是胞内SOS信号通路最早的作用对象, 这与Zhang等[30]的研究结论相符。
 | 图8 甘蔗ScSOS1基因在不同外源胁迫下的表达 误差线为每组处理的标准误差(n=3)。Fig. 8 ScSOS1 gene expression of sugarcane under different exogenous stresses The error bars represent the standard error of each treating group (n=3). |
前人研究表明, SOS1基因在植物耐盐和抵抗渗透胁迫方面发挥了重要的作用[48, 49, 50, 51, 52, 53]。周玲玲等[48]研究发现, 过量表达大叶补草LgSOS1基因的拟南芥, 耐盐性显著提高。王姝杰等[49]研究表明, 过表达蓝藻质膜Na+/H+逆向转运蛋白基因Nhap能够显著提高转基因烟草Fl代的耐盐性。此外, 还有研究表明, 在盐胁迫下, 质膜Na+/H+逆向转运蛋白基因在拟南芥、苔藓、胡杨、海草等植物中均呈现上调表达趋势[50, 51, 52, 53]。Munns等[54]认为, 植物激素与植物耐盐性之间有着密切的联系, 其中ABA在植物应答盐胁迫过程中具有重要的作用, ABA是植物适应盐胁迫过程中的最初的信号物质。RT-qPCR表达分析结果表明, ScSOS1基因在甘蔗叶鞘、蔗皮、蔗髓、侧芽和根中均有表达, 其中在叶鞘中的表达量最高, 在根中的表达量最低, 说明该基因在甘蔗中的表达具有组织特异性。此外, 在NaCl处理条件下, ScSOS1基因被诱导表达, 并在24 h的表达量达到最高, 这与Zhao[45]研究玉米SOS1基因的表达分析结果相同, 推测该基因可能在甘蔗耐盐机制中起作用。ScSOS1基因的表达量在PEG、SA和ABA胁迫下都会增加, 其中受PEG诱导最强烈并随处理时间的延长而持续增加。前人研究表明, 当植物处于高盐环境时, 最先受到水分胁迫的影响[55], 因此PEG是模拟渗透胁迫的理想物质[56]。有文献报道, ABA处理植株能增强其在盐胁迫条件下的适应能力[57]。谢崇波等[58]构建的SOS转录调控网络中有依赖ABA途径和不依赖ABA途径, 其中有25个基因与ABA信号途径相关。本研究结果揭示, 甘蔗ScSOS1基因可能在甘蔗耐盐性方面发挥一定的作用, 同时还可能参与了甘蔗的抗旱反应。
从甘蔗中成功克隆到一个ScSOS1基因, 该基因包含一个1272 bp的开放读码框, 编码423个氨基酸。ScSOS1蛋白与高粱SbSOS1蛋白的氨基酸同源性最高, 达87.0%。同时, 该基因的表达具有组织特异性, 在叶鞘中的表达量最大。ScSOS1基因在高盐和其他非生物胁迫下的表达量都会增加, 推测该基因可能在甘蔗耐盐和抗旱的分子机制上发挥一定的作用。研究结果为甘蔗SOS家族基因中其他成员的克隆及其深入的功能解析积累了基础资料。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|
| [49] |
|
| [50] |
|
| [51] |
|
| [52] |
|
| [53] |
|
| [54] |
|
| [55] |
|
| [56] |
|
| [57] |
|
| [58] |
|