第一作者联系方式: E-mail: 18505143324@163.com, Tel: 18505413324
甘蓝型油菜作为多倍体起源和发生的历史较短, 遗传背景较为狭窄, 人工合成甘蓝型油菜可作为植物多倍化研究的优选模型, 本文以人工合成的甘蓝型油菜为材料, 通过HPLC分析发现白菜型油菜和甘蓝的甲基化率分别为8.33%和15.88%, 2个杂种株系的全基因组甲基化水平介于双亲之间, 分别为10.29%和12.83%。MSAP分析发现杂种F1代及其亲本的甲基化水平存在明显差异(白菜型油菜<杂种F1<甘蓝), 杂种F1代的甲基化变异(23.71%)中来自A、C基因组的变异分别占6.60%和10.16%。MSAP差异性条带的序列分析发现多倍化过程中与甲基化变化相关的基因参与了多种生物学过程, 且差异甲基化基因在人工合成甘蓝型油菜及其亲本间的表达差异与甲基化修饰模式是一致的。本研究为了解甘蓝型油菜多倍化过程中发生的表观变异奠定了基础。
The genetic background of Brassica napus, one of the most important oil crops in China, is relatively narrow due to the short history of its polyploid origin. Resynthesized B. napusprovides an optimal model for researches on plant polyploidization. In the present study, we compared the DNA methylation levels in synthesized B. napus (F1 generation) and diploid parents using high-performance liquid chromatography (HPLC) and DNA methylation-sensitive amplification polymorphism (MSAP) analysis. HPLC analysis indicated methylation rates of 8.33% and 15.88% in B. rapa and B. oleracea, respectively. While the methylation rate of two hybrids was 10.29% and 12.83%, which were in-between of the parents’ values. MSAP analysis proved the different methylation levels in F1 generation and diploids, with the lowest and highest methylation levels identified in B. rapa and B. oleracea, respectively. Variance of the DNA methylation in F1was 23.71%, among which 6.60% and 10.16% were inherited from A and C genome, respectively. Sequence analysis of MSAP polymorphic fragments indicated genes involved in multiple molecular functions were changed during polyploidization. Expression analysis of these genes agreed to the corresponding methylation changes. This study provides preliminary basis for understanding epigenetic variations during B. napus polyploidization.
多倍体化是植物进化的重要原动力, 被子植物中约70%的植物在进化过程中经历了至少一次多倍化事件[1]。近年来通过基因组研究和比较基因组序列分析发现, 很多先前认为的典型二倍体植物(如拟南芥、水稻、大豆、玉米)究其来源也是古多倍体经过一系列演化而形成的[2]。通过研究植物多倍体有助于开发利用更多具有经济价值的多倍体植物资源。芸薹属(Brassica)是十字花科(Brassicaceae)中最为重要的一个属, 其中包括很多重要的经济作物。Nagahararu[3]证实该属包括白菜型油菜(B. rapa, AA, 2n= 20)、黑芥(B. nigra, BB, 2n= 16)和甘蓝(B. oleracea, CC, 2n = 18) 3个二倍体基本种, 以及由3个基本种通过杂交和自然加倍形成的甘蓝型油菜(B. napus, AACC, 2n = 38)、埃塞俄比亚芥(B. carinata, BBCC, 2n= 34)和芥菜型油菜(B. juncea, AABB, 2n= 36) 3个异源四倍体种。其中甘蓝型油菜的起源和驯化历史较短, 但现已成为世界上仅次于大豆的第二大油料作物[4]。然而, 狭窄的遗传基础也限制了甘蓝型油菜的发展[5]。白菜型油菜和甘蓝的品种较多, 且农艺性状变异较大, 利用白菜型油菜、甘蓝对甘蓝型油菜进行改良是一个重要的育种途径[6, 7]。通过人工合成甘蓝型油菜可以对其多倍化形成机制进行研究, 这也是拓宽甘蓝型油菜遗传基础的重要途径之一[8, 9, 10]。目前已有很多关于人工合成甘蓝型油菜的报道, 且这些人工合成甘蓝型油菜在早期世代具有很多优异的农艺性状[8, 11]。
植物多倍体基因组中来自不同亲本的大量重复基因在多倍化过程中会发生基因沉默或产生新的功能基因等[8, 12, 13]。研究表明, 异源多倍体油菜、小麦、黄瓜等的基因组在多倍化过程中均发生了大量变化, 以维持新形成多倍体基因组的稳定性, 包括基因的选择性丢失或沉默[14, 15]、染色体重排[16]等。此外, 异源多倍体基因组的稳定性还有赖于表观遗传修饰, 目前多倍体表观遗传现象中研究最透彻的是甲基化[17]。研究发现, 很多异源多倍体的重复及低拷贝序列的甲基化状态和基因表达与亲本基因组存在很大差异[18]。DNA甲基化除了与人工合成异源多倍体中的快速变化密切相关, 也参与新合成异源多倍体的稳定及进化[19, 20, 21]。Lukens等[18]对新合成甘蓝型油菜S0代表观遗传变异分析发现, 48%的CpG甲基化位点在亲本之间存在差异, 人工合成多倍体中存在甲基化位点的基因组偏好性; Xu等[22]通过甲基化敏感扩增多态性(MSAP)分析发现, 甘蓝型油菜异源多倍体化过程中, 杂种后代与亲本相比有6.84%基因发生甲基化变异, 其中1.94%的变异与多倍化过程相关; Gaeta等[8]发现人工合成甘蓝型油菜S0代中A、C基因组的甲基化变异是一致的, 但随后的5个世代中A基因组特异的甲基化变异远高于C基因组, 这表明异源多倍体的甲基化修饰存在基因组偏好性。此外, 研究发现人工合成甘蓝型油菜中发生表观修饰的变异是非常广泛的, 包括反转录转座子、编码基因、非编码DNA。Xu等[22]认为异源多倍化过程中的甲基化变异可以在全基因组范围内发生, 且19%的甲基化修饰发生在转座子区域, 这表明异源多倍体过程伴随着对反转座子频繁的表观遗传修饰作用。此外, Gaeta等[8]发现大多数S0代的甲基化变异在随后的S5代中得以保持, 但也有一部分甲基化修饰未能稳定遗传。
本实验室前期以白菜型油菜为母本、甘蓝为父本, 通过胚胎挽救的方法获得了人工合成甘蓝型油菜, 可作为研究甘蓝型油菜多倍化的实验材料。该多倍体具有分枝部位降低、分枝数多等优良性状。本研究拟通过高效液相色谱(HPLC)和MSAP技术, 比较分析人工合成甘蓝型油菜F1代、二倍体亲本的甲基化水平, 为了解甘蓝型油菜多倍化过程中发生的表观变异奠定基础。
白菜型油菜(Brassica rapa, AA, 2n = 20)品种扬州青、甘蓝(B. oleracea, CC, 2n = 18)品种永绿7号由江苏省里下河地区农业科学研究所提供, 由上述二倍体杂交而来的人工合成甘蓝型油菜F1代(B. napus, AACC, 2n = 38)由本实验室创制并保存。
取母本扬州青、父本永绿7号、人工合成甘蓝型油菜F1代的苗期叶片, 每个品种取3个单株作为重复, 通过CTAB法[23]提取全基因组DNA。参照Demeulemeester等[24]的方法, 向装有50 µ g DNA的玻璃管中加入300 µ L高氯酸, 沸水浴1 h裂解, 然后用1 mol L-1 KOH溶液将pH值调整至3~5, 在形成KClO4沉淀后取上清液, 13 523× g离心5 min后, 吸取上清液待测。室温下将DNA裂解液通过自动加样器进样至Agilent 1200型高效液相色谱仪(USA)的Alltima C18色谱柱(5 µ m, 250.0 mm × 4.6 mm)中, 柱温箱温度为40℃, 以10%甲醇、0.1 mol L-1戊烷磺酸钠和0.2%三乙胺的混合液(pH 4.0)为流动相, 洗脱速度为0.8 mL min-1, 紫外检测器检测波长为273 nm。以胞嘧啶(C)和5-MeC标样为对照, 通过计算5-MeC摩尔数/(5-MeC摩尔数+C摩尔数)的百分比检测基因组DNA中5-MeC的含量(DNA甲基化水平), 每个样品重复3次。
同上所述, 通过CTAB法提取母本扬州青、父本永绿7号、人工合成甘蓝型油菜F1代的全基因组DNA, 每个试验材料取3株重复的DNA进行混池。参考Xu等[22]的MSAP分析方法稍作修改。用EcoR I (15 U)/Msp I (10 U)和EcoR I (15 U) /Hpa II (10 U)两组限制性内切酶对每个样品分别双酶切。MSAP分析所用接头、预扩增、选择扩增引物见表1。酶切产物与10 pmol EcoR I接头和10 pmol Hpa II/Msp I接头连接。利用表1中的预扩增引物对上述连接产物预扩后, 以预扩增产物为模板、表1中的选择性扩增引物PCR扩增。扩增产物经8%聚丙烯酰胺凝胶电泳分离检测, 统计分析条带。MSAP条带用“ +” 和“ -” 分别代表“ 有带” 和“ 无带” , 据此可以将条带分为(+, +)、(+, -)、(-, +)和(-, -) 4种类型。
将MSAP检测的差异片段切下、装至EP管内, 加入200 µ L TE, 浸泡30 min, 弃上层液体, 重复同样的操作2次; 再加入30 µ L ddH2O, 煮沸10 min, 然后用枪头捣碎胶块, 煮沸10 min后, 1286× g离心15 min, 上清液即为回收产物。胶回收产物经pMD19-T Vector (TaKaRa, Code: D102A)进行TA克隆后, 利用相应的选扩引物对单克隆进行PCR检测, 选取阳性克隆送生工生物工程(上海)股份有限公司测序。对所得序列在甘蓝型油菜基因组数据库(http://www.genoscope.cns.fr/brassicanapus/)中进行同源性检索。
采用RNAiso plus试剂盒(TaKaRa)提取母本扬州青、父本永绿7号、人工合成甘蓝型油菜F1代苗期叶片的总RNA。20~50 μ g总RNA中加入6 μ L 10× DNase I缓冲剂(TaKaRa)、4 μ L DNase I (RNase-free, 5 U μ L-1) (TaKaRa)、0.8 μ L RNase抑制剂(40 U μ L-1)进行基因组DNA消化。通过Transcriptor First Strand cDNA Synthesis Kit (Roche)合成cDNA。qRT-PCR反应体系为20 μ L, 包括1 μ L cDNA、正反引物(表2)各0.3 μ mol L-1、10 μ L FastStart Universal SYBR Green Master (ROX, Roche)。反应程序为50℃ 2 min, 95℃ 10 min; 40个循环反应95℃ 15 s, 60℃ 30 s, 72℃ 30 s。使用96孔板(Bio-Rad Laboratories)于ABI 7500 System (Applied Biosystems)上进行qRT-PCR反应。内参为甘蓝型油菜β -actin (NCBI AF111812), 以2-Δ Δ Ct法分析各基因的相对表达量。
| 表1 MSAP分析所用接头和引物序列 Table 1 The sequences of adapters and primers in MSAP analysis |
| 表2 qRT-PCR分析所用引物 Table 2 Primers for qRT-PCR analysis |
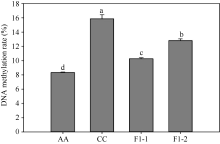
以胞嘧啶/5-甲基胞嘧啶为标准品, 通过HPLC技术对人工合成甘蓝型油菜F1代、二倍体亲本的全基因组甲基化水平分析(图1), 发现扬州青的全基因组甲基化率为8.33%, 永绿7号的甲基化率为15.88%; 而新合成的异源四倍体甘蓝型油菜F1代2个株系(F1-1、F1-2)的甲基化率分别为10.29%和12.83% (图2), 都与亲本存在显著差异。杂种后代的甲基化水平相对于扬州青呈上升趋势, 但未达到永绿7号的甲基化水平。
不同生物基因组中DNA甲基化胞嘧啶所占比率存在较大的差异, 某些动物如线虫中不含甲基化胞嘧啶, 昆虫中占0~3%, 哺乳类和鸟类占5%, 两栖类和鱼类占10%, 但在有些植物中能达到30%。对于同种生物的不同组织和器官, 或者同一组织的不同发育阶段, 甲基化胞嘧啶所占的比率也可能存在差异[25]。DNA甲基化胞嘧啶的分布存在种属特异性以及组织特异性, 并可随生物发育阶段的不同而发生改变[26, 27]。本研究中, 白菜型油菜扬州青以及甘蓝永绿7号是芸薹属的2个二倍体物种, 而由这2个亲本杂交所获得的甘蓝型油菜是属于芸薹属异源四倍体物种。利用HPLC对二倍体白菜型油菜、甘蓝及其杂种F1代的同一发育阶段、同一组织进行DNA甲基化分析, 结果显示三者的DNA甲基化率存在明显的差异, 且A基因组的全基因组甲基化率明显小于C基因组的甲基化率, 而人工合成甘蓝型油菜的甲基化率高于母本白菜型油菜、低于父本甘蓝, 这可能是由于杂种植株的基因组同时包含了A、C两套基因组, 同源染色体组在形成多倍体的过程中发生了复杂的表观遗传修饰, 以维持多倍体基因组的稳定性。
选用68对引物对扬州青、合成种F1代(2.1中甲基化比率较高的株系)、永绿7号进行MSAP分析, 共统计1181个条带, 根据条带的不同类型可分为7大类(图3), a类, 片段类型同A基因组(136条), 包括9个亚类; b类, 片段类型同C基因组(107条), 也包括9个亚类; c类, 包括所有的其他叠加类型(658条), 共3个亚类; d类, 包括3个亚类, 只有A基因组条带发生变化(78条); e类, 包括3个亚类, 只有C基因组发生变化(120条); f类, 包括3个亚类, A和C基因组条带都发生了变化(25条), 其中一个类型条带较弱未显示; g类, 包括3个亚类, 甲基化变化不属于A和C基因组, 即出现了新增条带(57条)。
根据甲基化变异类型可将上述7大类归结为2种类型: ①甲基化叠加型, 包括a、b、c三个大类; ②甲基化变化型: 包括d、e、f、g四种。综上, 杂种F1的甲基化变化率为23.71%, 其余76.29%只是亲本条带的简单叠加, A基因组的甲基化变异为6.60%, C基因组的变化(10.16%)明显高于A基因组。此外, M位点(全甲基化)变化率占总变化的28.21%, 而H位点(半甲基化)占40%, 2个位点都发生变化的比率为31.79% (表3)。杂种F1代的DNA甲基化类型发生变化的比率占23.71%。
根据甲基化水平差异对所有MSAP条带统计发现, 扬州青、永绿7号及杂种植株F1所扩增总条带数差异很小, 扬州青的总甲基化率最低(20.34%), 永绿7号的总甲基化率最高(22.44%), 杂种植株F1的总甲基化率介于两者之间(21.40%)。其中全甲基化率与该趋势一致, 半甲基化率则是杂种植株中最高, 扬州青次之, 永绿7号最低(表4)。扬州青和杂种F1代植株的半甲基化率高于全甲基化率, 而永绿7号的全甲基化率高于半甲基化率, 由此推测前者基因组CCGG位点发生甲基化的方式主要是以单链外侧胞嘧啶甲基化(mCCGG)为主, 后者基因组主要以双链内部胞嘧啶甲基化(CmCGG)为主。
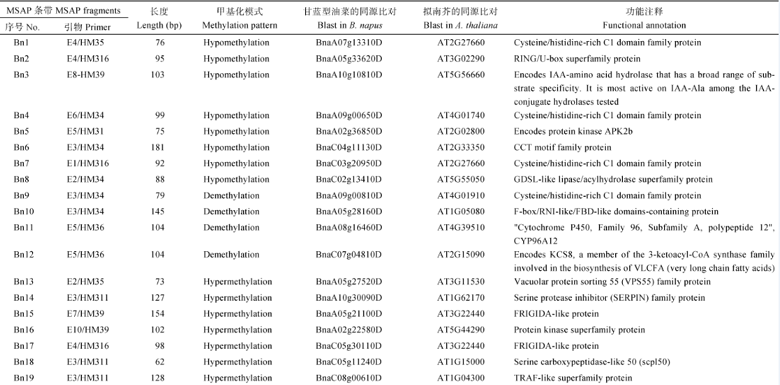
对MSAP分析的多态性片段进行TA克隆、测序, 共获得19个MSAP差异条带的序列信息, 将测序结果在甘蓝型油菜数据库(http://www.brassica. info/)中进行基因的同源性比对, 比对结果在拟南芥数据库(http://www.arabidopsis.org/)中进行基因的功能比对, 推测差异条带的具体功能信息(表5)。由比对信息可知, 甘蓝型油菜人工合成过程中所涉及的基因包括半胱氨酸/组氨酸C1域家族蛋白、RING/U盒蛋白超家族、CCT修饰家族蛋白、类GDSL脂肪酶/乙酰水解酶蛋白超家族、F-盒/类RNI/类FBD结构域蛋白质、液泡蛋白排序55 (VPS55)家族蛋白、丝氨酸蛋白酶抑制剂家族蛋白、蛋白激酶家族蛋白、类FRIGIDA家族蛋白、类丝氨酸羧肽酶50、类TRAF蛋白超家族, 以及IAA氨基酸、蛋白激酶APK2b、KCS8、类SABP2 (水杨酸甲酯酯酶)蛋白的编码基因。
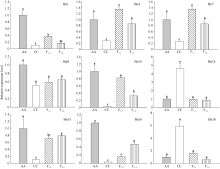
对上述测序成功的MSAP差异片段在人工合成甘蓝型油菜(F1代)及其二倍体亲本中的表达量分析(图4)发现, Bn1、Bn4、Bn7、Bn8、Bn10等在杂种F1代植株中发生去甲基化修饰的基因, 在杂种F1中的表达量高于双亲或介于两亲本之间; Bn14、Bn15、Bn16、Bn18等在杂种植株中发生高甲基化的基因, 在杂种植株中的表达量均介于两亲本之间。Bn1的同源基因编码半胱氨酸/组氨酸C1域家族蛋白, 是一种锌指结构域。该蛋白可通过与RNA和DNA结合, 参与调控植物细胞的凋亡和应激反应(如抗病反应)[28]。该基因在杂种F1代与亲本相比表现为低甲基化修饰, 但该基因在杂种F1代的表达水平介于双亲之间, 且亲本AA中Bn1的表达量最高, 推测亲本AA对外源病菌(如野油菜黄单胞菌)的抗性可能高于父本CC, 且杂种F1代的抗性介于双亲之间。Bn4的同源基因同样编码半胱氨酸/组氨酸C1域家族蛋白, 但该基因在杂种F1代的表达量显著高于父本CC, 且其中株系F1-1中Bn4的表达量高于母本AA。通过对杂种后代中Bn1/Bn4的表达变化分析表明, 异源四倍体中相关抗性基因的表达水平发生了变化, 这对于甘蓝型油菜的品质改良有重要的意义。但杂种后代的这种基因表达变异是否优于现有甘蓝型油菜品种还有待后续研究。Bn7在杂种F1代和亲本之间的甲基化修饰模式、基因表达变化与Bn1是一致的。Bn8的同源基因编码类GDSL脂肪酶/乙酰水解酶蛋白超家族, 该基因在甘蓝型油菜的非生物胁迫应答、生物胁迫应答、以及种子发育和脂质代谢中发挥着不可忽视的重要作用[29]。Bn8在杂种F1代与亲本相比表现为低甲基化修饰, 且该基因在母本AA中的表达量最高, 杂种F1代中的表达量略高于父本CC。Bn10的同源基因编码F-box结构域蛋白, 对植物调节蛋白的降解起着重要的调控作用, 在一些重要的信号传导系统和信号通路中发挥重要的功能[30]。Bn10在杂种F1代与亲本相比表现为去甲基化修饰, 且其在F1代的表达水平介于双亲之间, 亲本AA中
| 表3 人工合成甘蓝型油菜F1代植株及其二倍体亲本的甲基化模式 Table 3 Patterns of methylation in resynthesized B. napus(F1) and its diploid parents |
| 表4 人工合成甘蓝型油菜F1代植株及其亲本的DNA甲基化水平 Table 4 DNA methylation level in resynthesized B. napus(F1) and diploid parents |
的表达量最高。最近有研究报道, 很多与昼夜调节相关的基因在白菜型油菜的全基因三倍化过程中得到了选择性的保留, 但编码F-box蛋白的基因家族(ZEITLUPE)却是例外[31]。Bn14编码丝氨酸蛋白酶抑制剂家族蛋白, 前人研究发现过表达丝氨酸蛋白酶抑制基因(如MTI-2)可以提高甘蓝型油菜对小菜蛾的抵御能力[32]。本研究发现杂种F1代中Bn14与亲本相比发生超甲基化修饰, 其在杂种F1代的表达量显著低于父本CC, 与母本AA无显著差异, 由此推测, 父本CC对某些生物胁迫的抵御能力可能优于母本AA和杂种后代。Bn15编码类FRIGIDA家族蛋白, 最新研究发现, 该基因可以在长日照条件下通过调节开花基因的表达延迟拟南芥的开花时间[33]。本研究发现杂种F1代中该基因的表达水平明显高于父本CC, 但低于母本AA。该基因在不同材料中的表达差异是否与它们的花期差异有关还有待进一步研究。Bn16编码蛋白激酶家族蛋白, 参与的生物学功能比较广泛, 其在F1代表现为超甲基化修饰, 且在F1中的表达量介于双亲之间。Bn18编码类丝氨酸羧肽酶50, 该蛋白的脂肪酰转移酶可以催化油菜籽中芥子酸酯类化合物的形成[34]。本研究发现该基因在杂种F1中发生超甲基化修饰, 其在父本CC中的表达量最高, 杂种株系F1-1中Bn18的表达量介于双亲之间。
| 表5 MSAP 差异片段的序列分析 Table 5 Sequence analysis of MSAP polymorphic fragments |
异源四倍体甘蓝型油菜是研究植物多倍化的良好模型, 但天然多倍体的遗传背景不清晰, 通过人工合成甘蓝型油菜可以对其多倍化形成机制进行研究[10]。目前, 已有很多关于人工合成甘蓝型油菜的报道, 且这些人工合成甘蓝型油菜在早期世代具有很多优异的农艺性状[8]。本实验通过HPLC技术分析, 发现杂种后代(10.29%和12.83%)的甲基化水平介于母本扬州青(8.33%)和父本永绿7号(15.88%)之间; MSAP分析发现, 扬州青(20.34%)、永绿7号(22.44%)及杂种植株F1 (21.40%)的总甲基化率差异很小。在植物基因组中, 胞嘧啶甲基化通常发生在CAG、CTG和CCG位点, HPLC分析所检测的是全基因组水平的胞嘧啶甲基化; 而MSAP分析只能揭示CG或部分CCG位点的甲基化修饰, 无法揭示CAG和CTG三核苷酸残基中胞嘧啶的甲基化程度以及其他碱基的胸嘧啶甲基化水平。此外, MSAP中每一个酶切和扩增位点可能包括同一序列的多个重复。因此, MSAP分析得到的DNA甲基化水平往往低于实际情况[35]。此外, 杂种F1代的DNA甲基化类型发生变化的比率占23.71%, 这有别于前人的研究。Lukens等[18]发现甘蓝型油菜中48.0% CpG甲基化位点发生改变; 而Xu等[25]研究发现玫瑰离体分化的甲基化变化率是6.8%。异源四倍体拟南芥中甲基化模式变化比率占8.3%[36]; 异源四倍体小麦中该比率是6.9%[21]; 水稻栽培种杂交后代中4.1%甲基化位点发生变化[37]; 而高狐草× 小麦属间杂种的不同株系的甲基化变化率在1.6%~9.2%之间[38]; 米草杂种中则有高达30%的变化比率[39]。Shaked等[21]研究发现, 小麦和甘蓝型油菜多倍体中的甲基化变异存在基因组偏好性。甘蓝型油菜中甲基化变异的基因组偏好性表现为: 若杂种细胞质来自A基因组, 则甲基化优先出现于C基因组, 反之亦然[8, 25]。本研究的统计结果也发现杂种F1代种源自母本白菜型油菜的甲基化变化(6.6%)低于C基因组甲基化(10.16%), 这与前人的研究结果是一致的。
我们通过对MSAP分析所获甲基化修饰基因进行表达分析, 发现多个基因在F1代中的表达量介于双亲之间, 这可能与多倍化过程中的遗传修饰和表观遗传修饰相关。Gaete等[40]认为甲基化修饰并不是引起人工合成甘蓝型油菜(S0代)基因组水平基因表达变异的主要机制, 而多倍化过程中的基因重组是引起基因表达变异的主要原因。本文所发现F1代基因表达水平介于双亲之间, 这除了与甲基化修饰相关外, 可能与多倍化过程中的染色体重组、基因片段的丢失、转座等相关。此外, 杂种F1代中基因表达水平高于亲本AA或CC, 据此推测杂种后代的某些性状优于亲本AA或CC, 这种杂种植株与任一亲本相比具有优势、或杂种后代某些性状优于双亲均值的现象也属于杂种优势[41], 具有一定的应用价值。
发现大量在多倍化过程中发生甲基化修饰变异的条带, 这些基因在杂种后代及双亲之间均存在表达差异, 为了解甘蓝型油菜多倍化过程中发生的表观变异奠定了基础。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|